.
Condición fitosanitaria: Presente
Grupo de cultivos: Hortícolas
Especie hospedante: Brassica spp., repollo (Brassica oleracea), mostaza blanca (Sinapis alba L.)
Rango de hospedantes: Xcc puede causar enfermedad en una gran cantidad de especies de Brassicaceae (ex-Cruciferae), incluidos cultivos vegetales de Brassica de importancia económica y varios otros cultivos crucíferos, ornamentales y malezas, incluida la planta modelo Arabidopsis thaliana.
Etiología: Bacteria. Gram negativa. Necrotrófica (hemibiotrófica)
Agente causal: Xanthomonas campestris pv. campestris (Xcc)
Taxonomía: Bacteria > Proteobacteria > Gammaproteobacteria > Xanthomonadales > Xanthomonadaceae > Xanthomonas > Xanthomonas campestris
.
Xanthomonas campestris pv. campestris (Xcc) es un patógeno vascular transmitido por semillas que causa la enfermedad de la pudrición negra en Brassicaceae. Esta bacteria tiene un estilo de vida complejo compuesto por etapas tanto epífitas como endofíticas (Vicente y Holub, 2013) que se han estudiado utilizando genética molecular desde la década de 1980 (Daniels et al., 1984). La vida epífita de Xcc está asociada con tensiones ambientales como la luz ultravioleta o la deshidratación y depende, por ejemplo, de la producción de exopolisacáridos de xantano (EPS) o pigmentos protectores como la xantomonadina. En condiciones favorables, Xcc obtiene acceso a los tejidos internos de la hoja a través de heridas o hidatodos (Cerutti et al., 2017).
Las colecciones de aislados de Xcc se han diferenciado en razas fisiológicas basándose en la respuesta de varias líneas de especies de Brassica. El genoma de Xcc consta de un solo cromosoma de aproximadamente 5 100 000 pb, con un contenido de GC de aproximadamente el 65% y un número medio previsto de secuencias de ADN codificantes (CDS) de 4308 (Vicente y Holub, 2012). Se han identificado y estudiado tres sistemas de secreción diferentes en Xcc. Los clusters de genes xps y xcs codifican un sistema de secreción de tipo II y los genes xps se han relacionado con la patogenicidad. Los genes que codifican el sistema de secreción de tipo IV (T4SS) se identificaron en los cromosomas de muchas otras especies de Xanthomonas, incluida Xcc (Vorholter et al., 2008). El grupo de genes hrp codifica un sistema de secreción de tipo III que se asocia con patogenicidad. Se ha reunido un inventario de genes efectores candidatos basándose en la homología con efectores conocidos. Una variedad de otros genes se han asociado con la virulencia y patogenicidad, incluidos los genes rpf, gum y wxc involucrados en la regulación de la síntesis de enzimas degradativas extracelulares, goma xantana y lipopolisacáridos.
El fitocromo bacteriano [bacteriofitocromo (BphPs)] de Xcc (XccBphP) funciona como un regulador de virulencia al modular procesos fisiológicos clave, incluida la producción de xantano, la formación de biopelículas y la capacidad de infección (Bonomi et al., 2016). Este fotorreceptor XccBphP regula la virulencia del patógeno frente a los cambios de luz ambientales. Recientemente, se ha descripto el mecanismo de señalización de longitud completa a nivel atómico (Otero et al., 2021).
.
.
.
Sintomatología
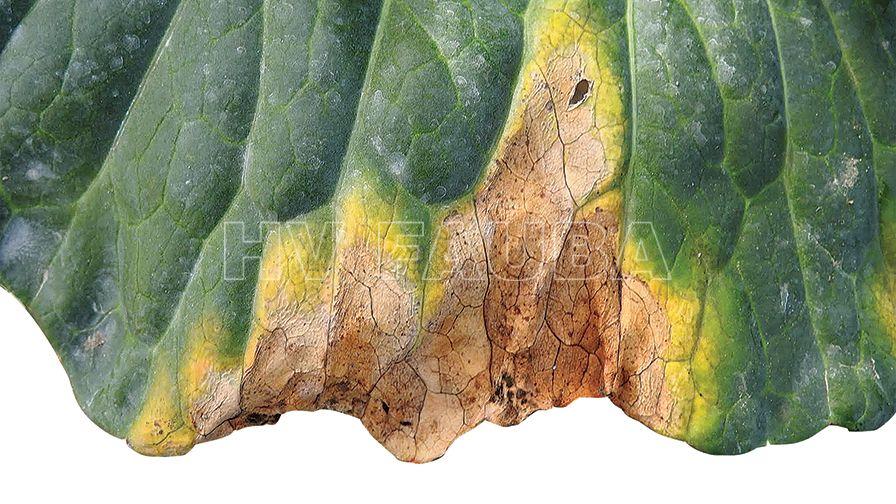
La podredumbre negra es una enfermedad vascular sistémica. Los síntomas típicos de la enfermedad incluyen lesiones amarillas en forma de V que comienzan en los márgenes de las hojas y ennegrecimiento de las nervaduras.
La pudrición negra es una enfermedad transmitida por semillas. La enfermedad se ve favorecida por condiciones cálidas y húmedas y puede propagarse rápidamente por la dispersión de la lluvia y el agua de riego.
Durante su vida, las plantas necesitan un flujo de agua continuo para suministrar agua, moléculas orgánicas e iones inorgánicos a los tallos y las hojas. El transporte de estas sustancias desde las raíces hasta las hojas se realiza a través del xilema y, en su mayor parte, es posible gracias a la acción combinada de la presión de la raíz y la transpiración. Cuando la transpiración disminuye (por ejemplo, oscuridad, alto nivel de CO2, sequía, etc.), el transporte de la savia del xilema depende principalmente de la presión de la raíz y da como resultado la gutación (la secreción de gotitas de agua a través de los poros de las plantas) del líquido del margen de la hoja en los hidatodos (Grunwald et al., 2003). Tanto la gutación como la presencia de hidatodos se han observado en una amplia gama de plantas vasculares pertenecientes a linajes de dicotiledóneas y monocotiledóneas, así como en pteridofitas y algunas plantas acuáticas (Chen y Chen, 2005). Por ejemplo, en Brassicaceae, los hidatodos se encuentran a lo largo del margen de la hoja, en la unión de las superficies adaxial y abaxial y frente a las terminaciones vasculares (Kawamura et al., 2010). En tales hidatodos epiteliales, la gutación se considera un proceso pasivo (Haberlandt, 1914). Se ha propuesto que los líquidos exudan a través de los poros acuosos presentes en la epidermis hidatódica, que se abren a un grupo de células pequeñas y de paredes delgadas con abundantes espacios intercelulares denominados epítema. El epítema se conecta directamente con la vasculatura de la planta. El líquido de gutación contiene minerales (por ejemplo, calcio, potasio, hierro y magnesio), azúcares (por ejemplo, glucosa, galactosa y fructosa) y aminoácidos (por ejemplo, Asp, Gln e His; Goatley y Lewis, 1966; Pilot et al., 2004). Además de la biosíntesis de auxinas (Aloni et al., 2003; Scarpella et al., 2006), las funciones fisiológicas de los hidatodos son en su mayoría desconocidas. Sin embargo, los análisis de su anatomía y fisiología sugieren un papel activo y selectivo en el intercambio de solutos (Lagarde et al., 1996). Curiosamente, las proteínas relacionadas con la patogénesis están enriquecidas en los fluidos de gutación de plantas de cebada sanas en relación con los extractos de hojas totales (Grunwald et al., 2003). Estos resultados sugieren que los hidatodos podrían construir una de las múltiples capas inmunes de las plantas montadas contra patógenos vasculares como Xanthomonas campestris pv campestris, Xanthomonas axonopodis pv dieffenbachiae, Xanthomonas oryzae pv oryzae, que se ha reportado que infectan preferentemente a los hidatodos (Russell, 1898; Hugouvieux et al., 1998; Niño-Liu et al., 2006; Bhat et al., 2010). Se han observado variaciones intraespecíficas en el tropismo por estomas o hidatodos que pueden resultar en distintas enfermedades. Por ejemplo, las cepas de los patovares oryzae y oryzicola de X. oryzae ingresan a las hojas de arroz por hidatodos o estomas causando tizón bacteriano de las hojas y estrías bacterianas de las hojas, respectivamente. De manera similar, las cepas del patovar raphani de X. campestris ingresan a las hojas de las plantas a través de los estomas, y solo causan manchas bacterianas en las hojas mientras que el patovar campestris ingresa por los hidatodos e invade el xilema para causar la enfermedad de la pudrición negra. Algunas cepas de Pseudomonas syringae parecen entrar principalmente por los estomas, pero en algunos casos también pueden iniciar una infección vascular por los hidatodos (Yu et al., 2013). Sin embargo, quedan por identificar los determinantes genéticos bacterianos subyacentes para tales comportamientos diferenciales (Bogdanove et al., 2011).
Una vez dentro de los hidatodos, Xcc se adapta a este nicho y adopta un estilo de vida biotrófico: las bacterias se multiplican lentamente en los espacios apoplásicos entre las células epiteliales sin causar alteraciones tisulares visibles como se observa hasta 3 días después de la infección (dpi) de los hidatodos de coliflor (Cerutti et al., 2017) . Luego se observa un cambio a un comportamiento necrotrófico, lo que resulta en la digestión casi completa del epítema 6 dpi y la vascularización de Xcc. La infección sistémica que llega a las flores provoca la colonización de las semillas y la transmisión a las plántulas (Darsonval et al., 2009; Dutta et al., 2014). Durante la infección, Xcc puede alimentarse del líquido de gutación, savia del xilema o tejidos vegetales (Duge de Bernonville et al., 2014). Este proceso puede facilitarse mediante sistemas de transporte de nutrientes de alta afinidad, como los transportadores dependientes de TonB (TBDT) para la nutrición de minerales (p. ej., hierro) o carbohidratos (p. ej., sacarosa; Blanvillain et al., 2007) y un amplio repertorio de enzimas degradantes de la pared celular secretadas a través del sistema de secreción de tipo II (T2S). Estas adaptaciones metabólicas deben coordinarse con precisión a lo largo del ciclo infeccioso. Los reguladores maestros incluyen el sistema de detección de quorum-sensing con factor de señal difusible rpf (DSF), sensores de disponibilidad de nutrientes y actividad metabólica y sistemas de dos componentes. Estas vías de regulación permiten que las células bacterianas respondan adecuadamente a diversos estímulos extracelulares encontrados durante su ciclo de vida, como estrés oxidativo, niveles de oxígeno, pH, temperatura y señales de la planta (He y Zhang, 2008; Qian et al., 2008; Tao et al. al., 2010). Entre estos reguladores, algunos son bien conocidos por desempeñar un papel importante en la virulencia, como el regulador de respuesta HrpG, que es, junto con el regulador transcripcional HrpX, el regulador maestro del regulón T3S (Noël et al., 2001; Roux et al., 2015; Zhang et al., 2020).
.
- Síntomas de la mancha en «V» en repollo. Autor: Ed Sikora
- Autor: Ed Sikora
- Síntomas de la mancha en «V» en mostaza blanca en Entre Ríos. Autor: Pablo Daniel Velazquez, INTA E.E.A. Concepción del Uruguay, E.E.A. Paraná
- Autor: Ty Torrance
- Autor: Margaret-McGrath
- Autor: Ty Torrance
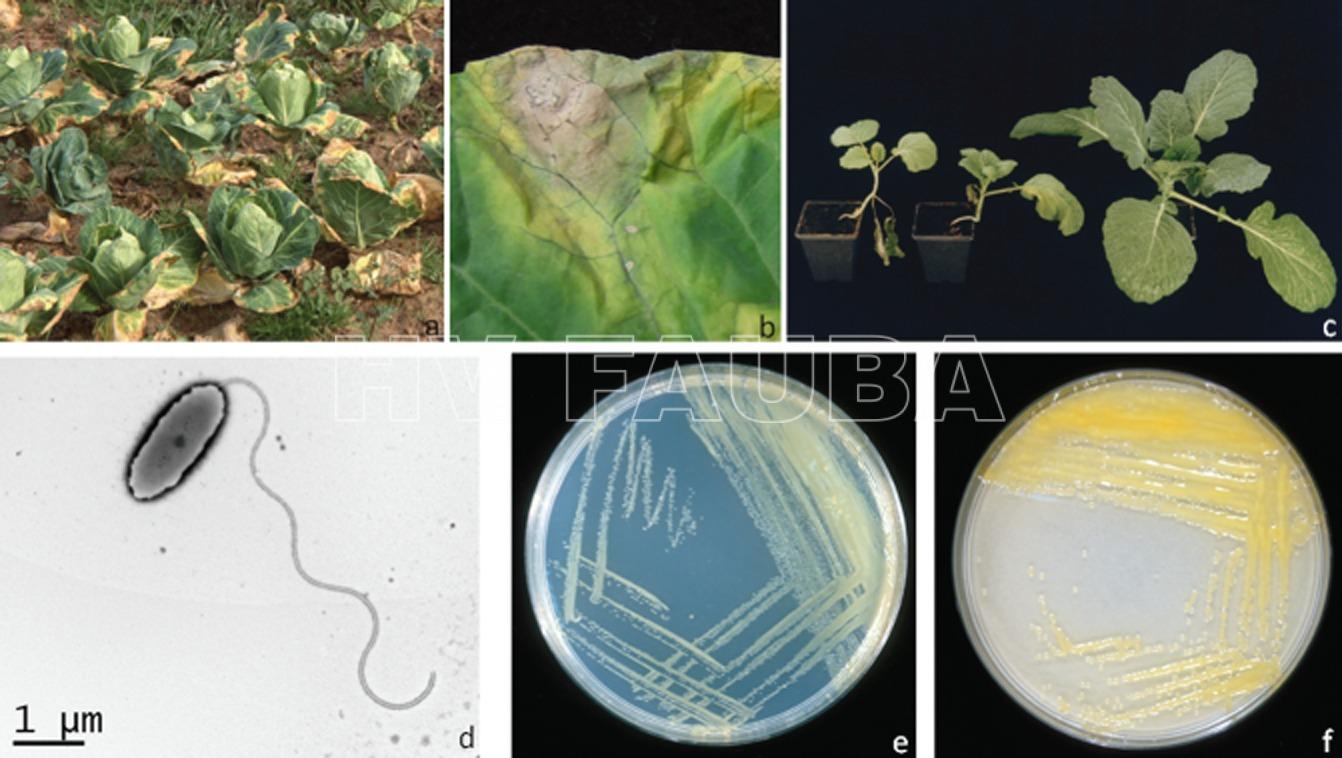
- (a) Síntomas de podredumbre negra en un campo de Brassica oleracea. (b) Lesión típica en forma de V de la pudrición negra en una hoja de repollo. c) Dos plantas de repollo con síntomas de infección sistémica tras la inoculación de Xanthomonas campestris pv. campestris y una planta de control sano. (d) Imagen de microscopía electrónica de una célula de X. campestris pv. campestris en forma de bastón que muestra un solo flagelo polar. (e) Cultivo Xanthomonas campestris pv. campestris en medio de King B. (f) Xanthomonas campestris pv. campestris cultivo en medio de levadura, dextrosa, carbonato cálcico. Autor: Vicente et al., 2012.
- Autor: Ed Sikora
- Autor: Ed Sikora
.
.
Manejo Integrado
El control de la pudrición negra es difícil y depende del uso de material de siembra libre de patógenos y la eliminación de otras posibles fuentes de inóculo (restos de cultivos infestados y malezas crucíferas).
La resistencia por genes mayores es muy difícil de obtener en B. oleracea (genoma de brassica C). La resistencia está más disponible en otras especies, incluidas las fuentes potencialmente útiles de resistencia de amplio espectro en B. rapa y B. carinata (genomas A y BC, respectivamente) y en el pariente silvestre A. thaliana.
.
.
.
Bibliografía
Aloni R, Schwalm K, Langhans M, Ullrich CI (2003) Gradual shifts in sites of free-auxin production during leaf-primordium development and their role in vascular differentiation and leaf morphogenesis in Arabidopsis. Planta 216: 841–853. doi: 10.1007/s00425-002-0937-8
Bhat NA, Syeed N, Bhat KA, Mir SA (2010) Pathogenicity and host range of Xanthomonas campestris pv. campestris—incitant of black rot of crucifers. J Phytol 2: 1–5.
Blanvillain S, Meyer D, Boulanger A, et al. (2007) Plant Carbohydrate Scavenging through TonB-Dependent Receptors: A Feature Shared by Phytopathogenic and Aquatic Bacteria. PLoS ONE 2(2): e224. doi: 10.1371/journal.pone.0000224
Bogdanove AJ, Koebnik R, Lu H, Furutani A, Angiuoli SV, Patil PB, Van Sluys MA, Ryan RP, Meyer DF, Han SW, et al. (2011) Two new complete genome sequences offer insight into host and tissue specificity of plant pathogenic Xanthomonas spp. J Bacteriol 193: 5450–5464. doi:
Bonomi HR, Toum L, Sycz G, et al. (2016) Xanthomonas campestris attenuates virulence by sensing light through a bacteriophytochrome photoreceptor. EMBO Rep. 17: 1565–1577. doi: 10.1128/JB.05262-11
Büttner, D. and Bonas, U. (2010), Regulation and secretion of Xanthomonas virulence factors. FEMS Microbiology Reviews, 34: 107-133. doi: 10.1111/j.1574-6976.2009.00192.x
Cerutti A, Jauneau A, Auriac M-C, et al. (2017) Immunity at cauliflower hydathodes controls systemic infection by Xanthomonas campestris pv. campestris. Plant Physiology 174: 700–716. doi: 10.1104/pp.16.01852
Chen C-C, Chen Y-R (2005) Study on laminar hydathodes of Ficus formosana (Moraceae) I. Morphology and ultrastructure. Bot Bull Acad Sin 46: 205–215.
Costa J, Pothier JF, Boch J, et al. (2021) Integrating science on Xanthomonadaceae for sustainable plant disease management in Europe. Mol Plant Pathol. 22(12): 1461-1463. doi: 10.1111/mpp.13150
da Silva, J.C., da Silva Júnior, T.A.F., Soman, J.M. et al. (2021) Survival of Xanthomonas campestris pv. campestris in the phyllosphere and rhizosphere of crops. Plant Soil 462: 389–403. doi: 10.1007/s11104-021-04832-0
, , , (1984) Cloning of genes involved in pathogenicity of Xanthomonas campestris pv. campestris using the broad host range cosmid pLAFR1. The EMBO Journal 3: 3323–3328. doi: 10.1002/j.1460-2075.1984.tb02298.x
, , , (2009) Adhesion and fitness in the bean phyllosphere and transmission to seed of Xanthomonas fuscans subsp. fuscans. Molecular Plant-Microbe Interactions 22: 747–757. doi: 10.1094/MPMI-22-6-0747
Domingo-Calap ML, Bernabéu-Gimeno M, Aure MC, et al. (2022) Comparative Analysis of Novel Lytic Phages for Biological Control of Phytopathogenic Xanthomonas spp. Microbiol Spectr. e0296022. doi: 10.1128/spectrum.02960-22
, , , et al. (2014) Role of blossom colonization in pepper seed infestation by Xanthomonas euvesicatoria. Phytopathology 104: 232–239. doi: 10.1094/PHYTO-05-13-0138-R
Fujikawa T, Inoue Y (2020) Genome Sequences of Two Pathogens of Cruciferous Crops, Xanthomonas campestris pv. raphani MAFF 106181 and X. campestris pv. campestris MAFF 301176. Microbiol Resour Announc 9(42): e00887-20. doi: 10.1128/MRA.00887-20
Gaetán S, López N (2005) First Outbreak of Bacterial Leaf Spot Caused by Xanthomonas campestris on Canola in Argentina. Plant Disease 89(6): 683. doi: 10.1094/PD-89-0683B
Gazdik F, Magnus S, Roberts SJ, et al. (2021) Persistence of Xanthomonas campestris pv. campestris in Field Soil in Central Europe. Microorganisms 9(3):591. doi: 10.3390/microorganisms9030591
Goatley JL, Lewis RW (1966) Composition of guttation fluid from rye, wheat, and barley seedlings. Plant Physiol 41: 373–375. doi: 10.1104/pp.41.3.373
Grunwald I, Rupprecht I, Schuster G, Kloppstech K (2003) Identification of guttation fluid proteins: the presence of pathogenesis-related proteins in non-infected barley plants. Physiologia Plantarum 119: 192-202. doi: 10.1034/j.1399-3054.2003.00202.x
Guy E, Genissel A, Hajri A (2013) Natural Genetic Variation of Xanthomonas campestris pv. campestris Pathogenicity on Arabidopsis Revealed by Association and Reverse Genetics. ASM Journals mBio 4(3). doi: 10.1128/mBio.00538-12
Haberlandt G (1914) Physiological Plant Anatomy. Macmillan and Co., London
Haq F, Xu X, Ma W, et al. (2021) A Xanthomonas transcription activator-like effector is trapped in nonhost plants for immunity. Plant Communications. doi: 10.1016/j.xplc.2021.100249
He YW, Zhang LH (2008) Quorum sensing and virulence regulation in Xanthomonas campestris. FEMS Microbiology Reviews 32: 842–857. doi: 10.1111/j.1574-6976.2008.00120.x
Hugouvieux V, Barber CE, Daniels MJ (1998) Entry of Xanthomonas campestris pv. campestris into hydathodes of Arabidopsis thaliana leaves: A system for studying early infection events in bacterial pathogenesis. Mol Plant Microbe Interact 11: 537–543. doi: 10.1094/MPMI.1998.11.6.537
Islam MT, Lee B-R, Park S-H, et al. (2017) Cultivar Variation in Hormonal Balance Is a Significant Determinant of Disease Susceptibility to Xanthomonas campestris pv. campestris in Brassica napus. Front. Plant Sci. 8: 2121. doi: 10.3389/fpls.2017.02121
, , , et al. (2021) New insights into the genetic diversity of Xanthomonas campestris pv. campestris isolates from winter oilseed rape in Serbia. Plant Pathol. 70: 35– 49. doi: 10.1111/ppa.13273
Kawamura E, Horiguchi G, Tsukaya H (2010) Mechanisms of leaf tooth formation in Arabidopsis. Plant J 62: 429–441. doi: 10.1111/j.1365-313X.2010.04156.x
Lagarde D, Basset M, Lepetit M, et al. (1996) Tissue-specific expression of Arabidopsis AKT1 gene is consistent with a role in K+ nutrition. Plant J 9: 195–203. doi: 10.1046/j.1365-313X.1996.09020195.x
Luneau JS, Cerutti A, Roux B, et al. (2021) Xanthomonas transcriptome inside cauliflower hydathodes reveals bacterial virulence strategies and physiological adaptations at early infection stages. Molecular Plant Pathology 00, 1– 16. doi: 10.1111/mpp.13117
Melotto M, Underwood W, He SY (2008) Role of stomata in plant innate immunity and foliar bacterial diseases. Annu Rev Phytopathol. 46: 101-22. doi: 10.1146/annurev.phyto.121107.104959
Michalopoulou VA, Mermigka G, Kotsaridis K, et al. (2022) The host exocyst complex is targeted by a conserved bacterial type-III effector that promotes virulence. Plant Cell.: koac162. doi: 10.1093/plcell/koac162
Niño-Liu DO, Ronald PC, Bogdanove AJ (2006) Xanthomonas oryzae pathovars: Model pathogens of a model crop. Mol Plant Pathol 7: 303–324. doi: 10.1111/j.1364-3703.2006.00344.x
, , (2001) cDNA-AFLP analysis unravels a genome-wide hrpG-regulon in the plant pathogen Xanthomonas campestris pv. vesicatoria. Molecular Microbiology 41: 1271–1281. doi: 10.1046/j.1365-2958.2001.02567.x
Otero LH, Foscaldi S, Antelo GT, et al. (2021) Structural basis for the Pr-Pfr long-range signaling mechanism of a full-length bacterial phytochrome at the atomic level. Sci Adv. 7(48): eabh1097. doi: 10.1126/sciadv.abh1097
Pilot G, Stransky H, Bushey DF, et al. (2004) Overexpression of GLUTAMINE DUMPER1 leads to hypersecretion of glutamine from hydathodes of Arabidopsis leaves. Plant Cell 16: 1827–1840. doi: 10.1105/tpc.021642
Qian W, Han ZJ, He C (2008) Two-component signal transduction systems of Xanthomonas spp.: a lesson from genomics. Molecular Plant-Microbe Interactions 21: 151–161. doi: 10.1094/MPMI-21-2-0151
Roberts S, Hiltunen L, Hunter P, et al. (1999) Transmission from Seed to Seedling and Secondary Spread of Xanthomonas campestris pv. campestris in Brassica Transplants: Effects of Dose and Watering Regime. European Journal of Plant Pathology 105: 879–889. doi: 10.1023/A:1008790306489
, , , et al. (2015) Genomics and transcriptomics of Xanthomonas campestris species challenge the concept of core type III effectome. BMC Genomics 16: 975. doi: 10.1186/s12864-015-2190-0
Russell HL (1898) A bacterial rot of cabbage and allied plants. Univ of Wis Agric Exp Sta Bull 8: 1–39.
Scarpella E, Marcos D, Friml J, Berleth T (2006) Control of leaf vascular patterning by polar auxin transport. Genes Dev 20: 1015–1027. doi: 10.1101/gad.1402406
Tao J, He C (2010) Response regulator, VemR, positively regulates the virulence and adaptation of Xanthomonas campestris pv. campestris. FEMS Microbiol Lett. 304(1): 20-28. doi: 10.1111/j.1574-6968.2009.01892.x
, , , (2010) The cyclic nucleotide monophosphate domain of Xanthomonas campestris global regulator Clp defines a new class of cyclic di-GMP effectors. Journal of Bacteriology 192: 1020–1029. doi: 10.1128/JB.01253-09
Teper D, White FF, Wang N (2023) The Dynamic Transcription Activator-Like Effector Family of Xanthomonas. Phytopathology 113(4): 651-666. doi: 10.1094/PHYTO-10-22-0365-KD
Tran TM, Chng CP, Pu X, et al. (2021) Potentiation of plant defense by bacterial outer membrane vesicles is mediated by membrane nanodomains. The Plant Cell koab276. doi: 10.1093/plcell/koab276
Velazquez PD (2014) Podredumbre negra o mancha en “V” en mostaza blanca (Sinapis alba L.) en Paraná, Entre Ríos. LINK
Vicente JG, Holub EB (2013) Xanthomonas campestris pv. campestris. Molecular Plant Pathology 14: 2-18. doi: 10.1111/j.1364-3703.2012.00833.x
Vieira PS, Bonfim IM, Araujo EA, et al. (2021) Xyloglucan processing machinery in Xanthomonas pathogens and its role in the transcriptional activation of virulence factors. Nat Commun 12: 4049. doi: 10.1038/s41467-021-24277-4
Vorholter FJ, Schneiker S, Goesmann A, et al. (2008) The genome of Xanthomonas campestris pv. campestris B100 and its use for the reconstruction of metabolic pathways involved in xanthan biosynthesis. J. Biotechnol. 134: 33–45. doi: 10.1016/j.jbiotec.2007.12.013
Yu A, Lepère G, Jay F, et al. (2013) Dynamics and biological relevance of DNA demethylation in Arabidopsis antibacterial defense. Proc Natl Acad Sci USA 110: 2389–2394. doi: 10.1073/pnas.1211757110
, , (2020) Analysis of HrpG regulons and HrpG-interacting proteins by ChIP-seq and affinity proteomics in Xanthomonas campestris. Molecular Plant Pathology 21: 388–400. doi: 10.1111/mpp.12903
, , , et al. (2023) Phytopathogenic bacteria utilize host glucose as a signal to stimulate virulence through LuxR homologues. Molecular Plant Pathology 24: 359– 373. doi: 10.1111/mpp.13302