.
Condición fitosanitaria: Plaga no cuarentenaria reglamentada **
** / Plaga Cuarentenaria Ausente para Ralstonia solanacearum raza 2
Grupo de cultivos: Hortícolas
Especie hospedante: Papa (Solanum tuberosum L.)
Rango de hospedantes: R. solanacearum tiene un rango de hospedantes inusualmente amplio, pudiendo infectar a más de 450 especies de plantas que pertenecen a más de 50 familias, que abarcan monocotiledóneas y dicotiledóneas y plantas herbáceas y leñosas (Norman et al., 2009; Cho et al., 2019).
Etiología: Bacteria. Gram negativa
Agente causal: Ralstonia solanacearum (Smith 1896) Yabuuchi et al. 1996 emend. Safni et al. 2014
Taxonomía: Bacteria > Proteobacteria > Betaproteobacteria > Burkholderiales > Burkholderiaceae > Ralstonia
Ralstonia solanacearum es una bacteria Gram-negativa, con forma de bastón, estrictamente aeróbica, de 0.5-0.7 x 1.5-2.0 μm de tamaño. Es muy sensible a la desecación y se inhibe en cultivo por bajas concentraciones (2%) de cloruro de sodio (NaCl). Para la mayoría de las cepas, la temperatura óptima de crecimiento es de 27-32ºC, sin embargo, algunas cepas tienen una temperatura óptima inferior de 27ºC.
.
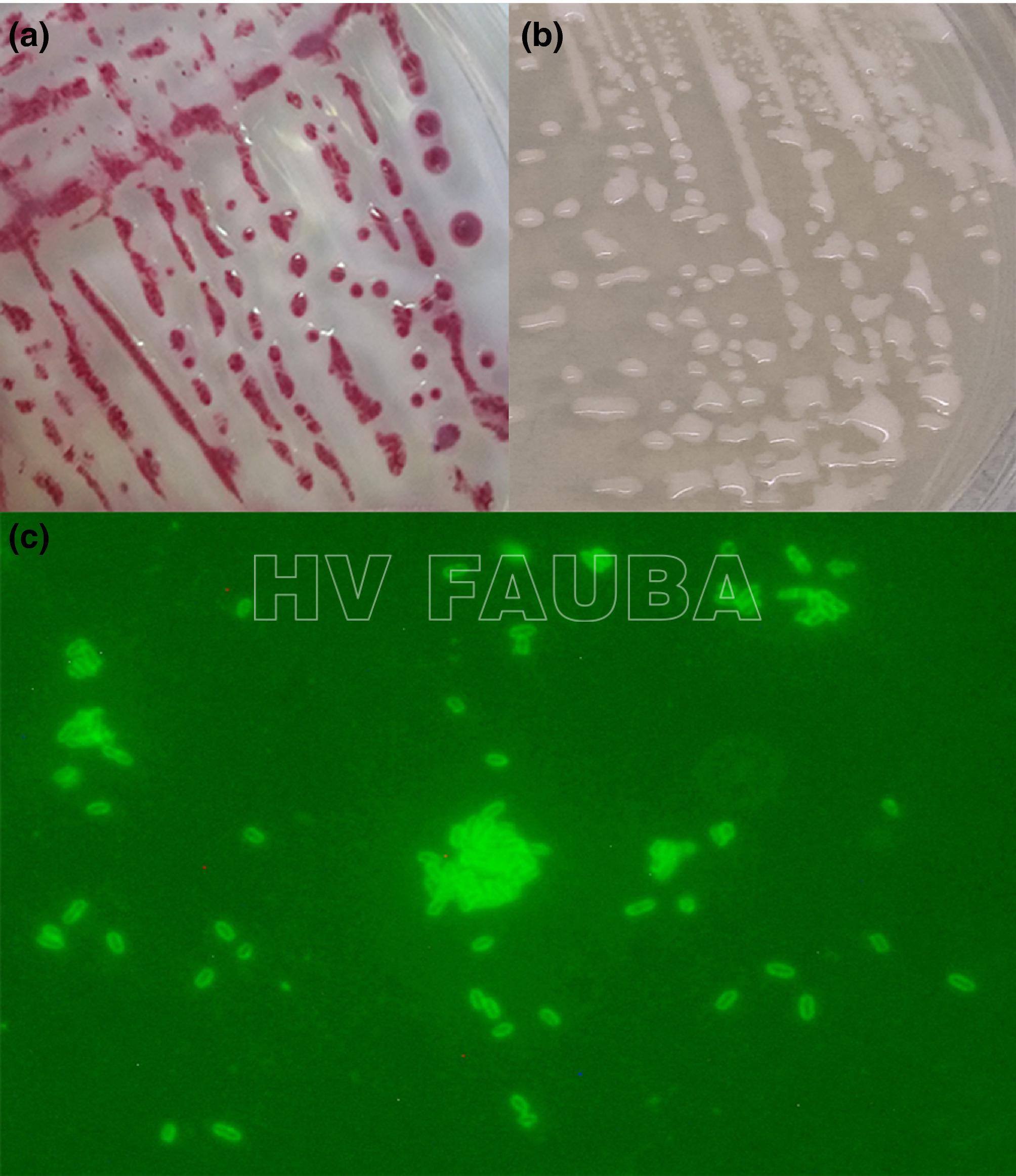
- Ralstonia solanacearum. (a) Colonias en SMSA, (b) colonias en SPA, (c) células fluorescentes verdes visibles bajo microscopio de fluorescencia (objetivo de inmersión en aceite 100x) en el ensayo de inmunofluorescencia para suspensión bacteriana usado a una dilución de aproximadamente 105 ufc / ml. Autor: Marković et al., 2021.
.
.
.
.
Sintomatología
.
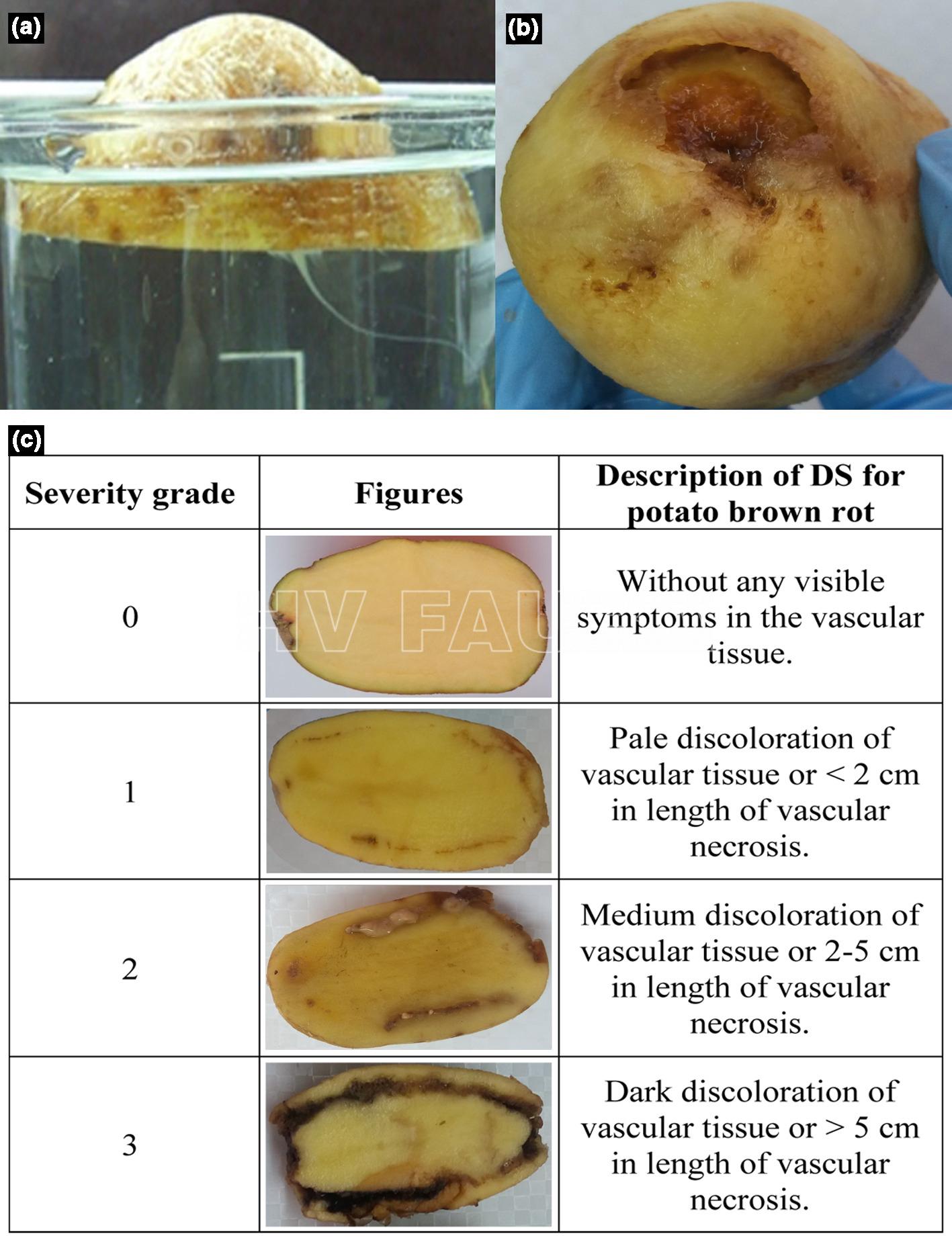
- Pudrición parda de la patata en tubérculos. (a) Exudado bacteriano (prueba de flujo) en un vaso con agua (cv. Lady Claire), (b) Síntomas externos (cv. Brooke), (c) Síntomas internos (cv. Brooke) presentados como diagrama utilizado para la estimación de la intensidad de la enfermedad. Las imágenes son de infecciones naturales. Autor: Marković et al., 2021.
- Ralstonia solanacearum en pruebas de patogenicidad. (a) Plantas de papa, planta de control negativo (izquierda), plantas después de 6 días (medio) y 2 semanas después de la inoculación (derecha); (b) plantas de tomate, plantas de control negativo (izquierda), plantas después de 6 días de inoculación (derecha); (c) plantas de berenjena, planta de control negativo (izquierda), plantas después de 6 días de inoculación (derecha). Autor: Marković et al., 2021.
Diagnóstico
.
Epidemiología
La principal fuente de inóculo de R. solanacearum son los tubérculos de papa infectados que se usan como papa semilla. Estos muchas veces son asintomáticos, pudiendo expresar síntomas cuando existen condiciones de humedad y temperaturas favorables para el desarrollo de la enfermedad. Esta es la forma de introducción del patógeno en lotes que antes no estaba presente. El suelo es otra fuente de inóculo importante, ya que esta bacteria puede sobrevivir en restos culturales, raíces y rizósfera de papas voluntarias, malezas y hospedantes alternativos (por ej. Solanum lycopersicum) (Sebastià et al., 2021).
La enfermedad puede ser diseminada por el agua de riego y por suelo adherido a maquinaría agrícola, herramientas y zapatos. Existe evidencia científica que R. solanacearum puede sobrevivir varios años en las raíces acuáticas de S. dulcamara, planta trepadora que crece con sus raíces inmersas en aguas superficiales, siendo foco de infección y dispersión del patógeno en los cursos de agua, favoreciendo la transmisión de la enfermedad por agua de riego, además de afectar a los cultivos agua abajo.
.
.
Bibliografía
Beutler J, Holden S, Georgoulis SJ, et al. (2022) Whole genome sequencing suggests that “non-pathogenicity on banana (NPB)” is the ancestral state of the Ralstonia solanacearum IIB-4 lineage. PhytoFrontiers™. doi: 10.1094/PHYTOFR-06-22-0068-SC
Champoiseau PG, Jones JB, Allen C (2009) Ralstonia solanacearum race 3 biovar 2 causes tropical losses and temperate anxieties. Online. Plant Health Progress. doi: 10.1094/PHP-2009-0313-01-RV
, , , et al. (2024) The Ralstonia solanacearum effector RipV1 acts as a novel E3 ubiquitin ligase to suppress plant PAMP-triggered immunity responses and promote susceptibility in potato. Plant Pathology 00: 1–13. doi: 10.1111/ppa.13885
, , , et al. (2023) Positive regulation of the PhcB neighbouring regulator PrhX on expression of the type III secretion system and pathogenesis in Ralstonia solanacearum. Molecular Plant Pathology 00: 1–13. doi: https://doi.org/10.1111/mpp.13398
, , , et al. (2021) Ralstonia solanacearum as a potato pathogen in Serbia: Characterization of strains and influence on peroxidase activity in tubers. Plant Pathology 00: 1– 15. doi: 10.1111/ppa.13421
Mizubuti ESG, Yamada JK, Santiago TR, et al. (2022) On the assessment of the sources of inoculum of bacterial wilt in Brazil. Trop. plant pathol. doi: 10.1007/s40858-022-00515-y
Nakano M, Mukaihara T (2018) Ralstonia solanacearum Type III Effector RipAL Targets Chloroplasts and Induces Jasmonic Acid Production to Suppress Salicylic Acid-Mediated Defense Responses in Plants. Plant Cell Physiol. 59(12): 2576-2589. doi: 10.1093/pcp/pcy177
Prieto Romo J, Morales Osorio JG, Salazar Yepes M (2012) Identification of new hosts for Ralstonia solanacearum (Smith) race 2 from Colombia. Revista de Protección Vegetal 27(3): 151-161. Link
Qi P, Huang M, Hu X, et al. (2022) Ralstonia solanacearum effector targets TGA transcription factors to subvert salicylic acid signaling, The Plant Cell koac015. doi: 10.1093/plcell/koac015
Robertson AE Fortnum BA, Wood TC, Kluepfel DA (2001) Diversity of Ralstonia solanacearum in the Southeastern United States. Contributions to Tobacco & Nicotine Research 19: 323-331. doi: 10.2478/cttr-2013-0719
Sebastià P, de Pedro-Jové R, Daubech B, et al. (2021) The Bacterial Wilt Reservoir Host Solanum dulcamara Shows Resistance to Ralstonia solanacearum Infection. Front. Plant Sci. 12: 755708. doi: 10.3389/fpls.2021.755708
Shen L, Yang S, Guan D, He S (2020) CaCML13 Acts Positively in Pepper Immunity Against Ralstonia solanacearum Infection Forming Feedback Loop with CabZIP63. International Journal of Molecular Sciences 21(11): 4186. doi: 10.3390/ijms21114186
Shi JL, Zai WS, Xiong ZL (2021) NB-LRR genes: characteristics in three Solanum species and transcriptional response to Ralstonia solanacearum in tomato. Planta 254(5): 96. doi: 10.1007/s00425-021-03745-7
, , , et al. (2023) Exploring the competitive potential of Ralstonia pseudosolanacearum and Ralstonia solanacearum: Insights from a comparative adaptability study. Plant Pathology 00: 1–17. doi: 10.1111/ppa.13848
Sun H, Jiao WB, Krause K, et al. (2022) Chromosome-scale and haplotype-resolved genome assembly of a tetraploid potato cultivar. Nat Genet. doi: 10.1038/s41588-022-01015-0
, , , et al. (2023) The transcription regulator ChpA affects the global transcriptome including quorum sensing-dependent genes in Ralstonia pseudosolanacearum strain OE1-1. Molecular Plant Pathology 24: 1370–1384. doi: 10.1111/mpp.13374
Tariq A, Sachin R (2020) Organic Management of Bacterial Wilt of Tomato and Potato Caused by Ralstonia solanacearum. Link
Truchon AN, Dalsing BL, Khokhani D, et al. (2022) Plant pathogenic Ralstonia phylotypes evolved divergent respiratory strategies and behaviors to thrive in xylem. bioRxiv 2022.11.09.515861; doi: 10.1101/2022.11.09.515861
, , , et al. (2023) A bacterial type III effector targets plant vesicle-associated membrane proteins. Molecular Plant Pathology 00: 1– 14. doi: 10.1111/mpp.13360
Wang Z, Luo W, Cheng S, et al. (2023) Ralstonia solanacearum – A soil borne hidden enemy of plants: Research development in management strategies, their action mechanism and challenges. Front. Plant Sci. 14: 1141902. doi: 10.3389/fpls.2023.1141902
Xia Y, Zou R, Escouboué M, et al. (2021) Secondary-structure switch regulates the substrate binding of a YopJ family acetyltransferase. Nat Commun 12: 5969. doi: 10.1038/s41467-021-26183-1
Zhang Y, Guo S, Zhang F, et al. (2023) CaREM1.4 interacts with CaRIN4 to regulate Ralstonia solanacearum tolerance by triggering cell death in pepper. Horticulture Research: uhad053. doi: 10.1093/hr/uhad053