Condición fitosanitaria: Presente
Grupo de cultivos: Oleaginosas
Especie hospedante: Girasol (Helianthus annuus)
Rango de hospedantes: estrecho a intermedio, distintos géneros de la familia Asteraceae (Compositae) o compuestas. (**)
Etiología: Pseudohongo (Oomycete). Biotrófico (parásito obligado) de suelo (soil-borne)
Agente causal: Plasmopara halstedii (Farl.) Berl. & De Toni
Taxonomía: Eukaryota > Stramenopiles > Oomycetes > Peronosporales > Peronosporaceae > Plasmopara
.
P. halstedii sensu stricto (Choi et al., 2009) representa un organismo morfológicamente bien caracterizado capaz de infectar a más de 80 especies de la familia Asteraceae (Gulya et al., 1997). Debido a que en la mayoría de los casos el patógeno se determinó de acuerdo con su morfología, no siempre complementado con los postulados de Koch, no es definitivo que estas infecciones fueran causadas por P. halstedii. El concepto de P. halstedii sensu stricto se basa en estudios moleculares que revelaron que los aislados de Plasmopara de Ambrosia artemisiifolia se distinguen claramente de otros aislados de Plasmopara que se originan de H. annuus o X. strumarium (Choi et al., 2009). El rango de hospedantes de P. halstedii puede entenderse de diferentes maneras según los conceptos de especie P. halstedii s.s. o s.l. y la situación se complica significativamente por la ocurrencia de infecciones cruzadas, saltos de hospedantes y la posible hibridación de especies crípticas, como en el caso de las especies Pseudoperonospora cubensis / Pseudoperonospora humuli (Runge et al., 2011).
(**) entre ellos Ambrosia, Dimorphotheca, Iva, Xanthium (Gulya 2002; Komjáti et al., 2007; Leppik, 1965; Viranyi, 1984; Walcz et al., 2000). Sin embargo, el presunto rango de hospedantes de P. halstedii sensu stricto es un tema de discusión constante, y muchos consideran al patógeno como un complejo de especies en lugar de una sola especie (Choi et al., 2009; Goker et al., 2007; Schroder et al., 2007; Schroder et al. al., 2011; Thines et al., 2009). Varios estudios recientes han identificado especies discretas de Plasmopara en hospedantes de Asteraceae que son genética y / o morfológicamente distintas de P. halstedii. Estos incluyen P. angustiterminalis en X. strumarium, P. invertifolia en Helichrysum bracteatum y una especie sin nombre que infecta a A. artemisifolia (Choi et al., 2009; Duarte et al., 2014; Komjáti et al., 2007).
.
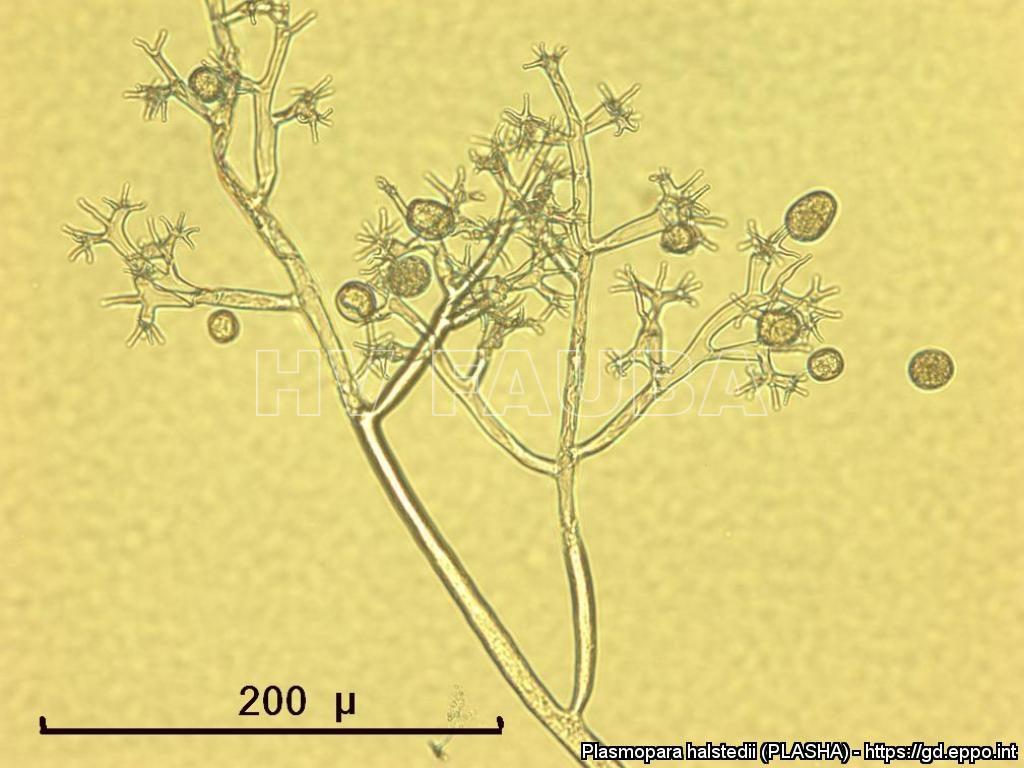
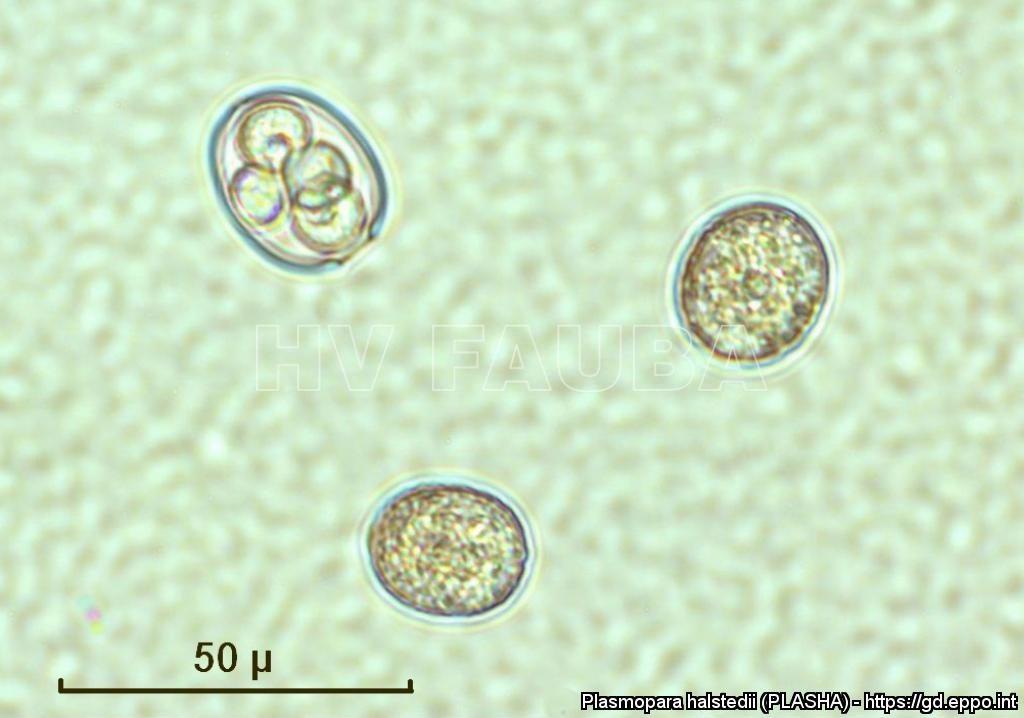
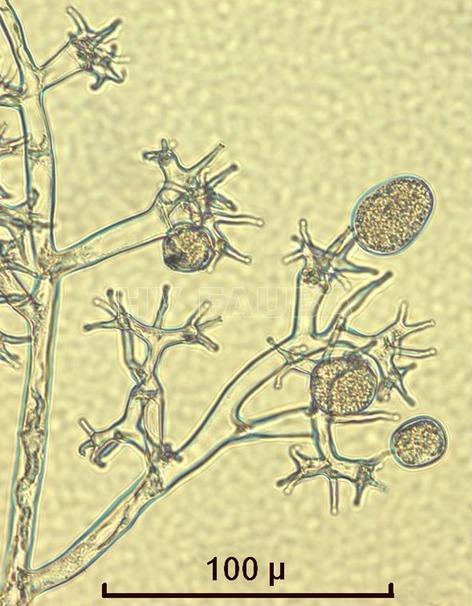
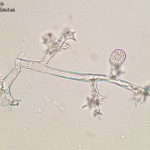
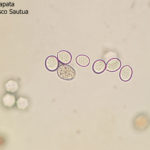
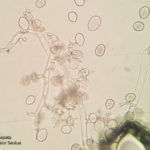
- Zoosporangios sobre zoosporangióforos de P. halstedii. Autor: Aranka Kormany, Fuente: EPPO Global Database
- Zoosporangios y formación de zoosporas en un esporangio de P. halstedii. Autor: Aranka Kormany, Fuente: EPPO Global Database
- Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
.
.
Antecedentes
P. halstedii es originario de América del Norte (Delmotte et al., 2008) donde fue encontrada por primera vez por Halsted cerca de Cambridge (MA, EE. UU.) sobre la planta herbácea perenne Eupatorium purpureum en 1876 y descrita más tarde por Farlow (1883; Virányi y Spring, 2011). El mildiú se registró por primera vez en girasoles cultivados en los Estados Unidos en la década de 1920 (Young y Morris, 1927). El cultivo de variedades de girasol de polinización abierta que eran altamente susceptibles a P. halstedii, tanto en Europa como en América del Norte, ayudó a la rápida propagación de la enfermedad (Virányi, 2002; Virányi et al., 2015) y provocó varios brotes graves (Zimmer, 1971; Göre, 2009). Por lo tanto, el mildiú velloso del girasol ha sido declarado patógeno de cuarentena en la Unión Europea desde 1992 (Delmotte et al., 2008).
.
Sintomatología
Dependiendo de la etapa de desarrollo del girasol en la que se produzca la infección, los síntomas característicos van desde la muerte de las plántulas jóvenes, el enanismo de las plantas, el blanqueamiento de las hojas (esporulación), hasta la producción de flores infértiles (Gascuel et al., 2015). La infección en la etapa de plántula puede ocurrir en condiciones ambientales óptimas, lo que resulta en una enfermedad sistémica. El damping-off o tizón de plántulas ocurre cuando las plántulas están expuestas a altas concentraciones de inóculo en el suelo combinado con suelos fríos (12-14°C) y muy húmedos.
Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base, acompañada en el envés de una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo; hojas pequeñas y encrespadas en toda su superficie; presencia de capítulos en posición horizontal, con el disco floral mirando hacia arriba; hasta muerte de plantas. En ataques severos, el mildew puede causar el enanismo de las plantas de girasol. Esta enfermedad es potencialmente dañina cuando ocurren infecciones tempranas entre pre-emergencia y 6 hojas verdaderas. A menudo ocurre en parches muy enfermos, lo que limita la capacidad del cultivo para compensar las áreas infectadas a escala de todo el campo.
Las infecciones causadas por esta enfermedad se pueden subdividir en dos categorías principales según los síntomas. Los síntomas sistémicos se producen cuando las plántulas jóvenes se infectan a través del sistema de raíces, lo que a menudo resulta en la muerte de la planta. Si las plántulas sobreviven, se atrofian severamente con hojas cloróticas y arrugadas, es decir, enanismo. La clorosis es una indicación de que las porciones de hojas están siendo colonizadas por el patógeno. La clorosis a menudo permanecerá asociada con las venas de las hojas, pero también puede cubrir áreas enteras de la hoja. Con períodos prolongados de clima fresco (15-18°C) junto con una alta humedad de las hojas en forma de lluvia o rocío, se formará un denso crecimiento algodonoso y blanco en las hojas, generalmente en la parte inferior. Si las plantas infectadas sistémicamente alcanzan la madurez, producen pocas semillas viables, si es que las hay, y los capítulos se caracterizan por mirar hacia arriba. El tamaño del capítulo también se reduce, al igual que el número y el peso de las semillas por capítulo. En raras ocasiones, los síntomas sistémicos también pueden aparecer en las hojas de medias o superiores de la planta, sin que se observen síntomas de mildiu en las hojas inferiores.
.
- Retraso severo (enanismo) en el crecimiento de las plantas de girasol debido a una infección sistémica por el patógeno causante del mildiú del girasol, Plasmopara halstedii. Autor: Gulya et al., 2019.
- Áreas cloróticas grandes en hojas de planta de girasol infectada sistémicamente con Plasmopara halstedii. Autor: Gulya et al., 2019.
.
Variabilidad genética
Desde la primera aparición de las razas P. halstedii en la década de 1970 (Zimmer, 1974), los mejoradores han desarrollado germoplasma de girasol utilizando genes de resistencia mayores (R), codificados como genes Pl (Vear et al., 2008). Hasta la fecha, se han descubierto casi 20 genes Pl y posteriormente se han incorporado al cultivo (Hahn y Wieckhorst, 2010; Qi et al., 2014, 2016; Virányi et al., 2015), lo que proporciona una amplia variación de resistencia raza-específica al mildiu (Tourvieille de Labrouhe et al., 2008). Al igual que en otros patosistemas, el cultivo de nuevos híbridos de girasol influye en la variabilidad patógena de P. halstedii.
P. halstedii es un oomycete biótrofo obligado el cual presenta en condiciones naturales, por presión de selección, elevada frecuencia de variantes con patogenicidad diferencial. Durante los últimos 20 años, han aparecido nuevos patotipos de P. halstedii que muestran una mayor virulencia (Ahmed et al., 2012; Nisha et al., 2021), y varios genes de resistencia mayores utilizados en híbridos de girasol se han vuelto ineficientes: por ejemplo, el gen Pl2 que confiere resistencia a los patotipos más frecuentes ha sido superado por diferentes patotipos emergentes (Moinard et al., 2006; Tourvieille de Labrouhe et al., 2000). La comprensión de la evolución del patotipo y la resistencia del girasol es, por lo tanto, una cuestión agronómica clave en el manejo de esta enfermedad. A nivel mundial se ha determinado la existencia de al menos 44 razas de P. halstedii (Roeckel-Drevet et al., 2003; Sedlářová et al., 2016). En Argentina se han confirmado las razas 300, 330, 700, 710, 730 y 770. En 1998 predominaron las razas 300 y 330 en el país. Luego, cuando aparecieron las razas 700, 710, 730 y 770, los mejoradores tuvieron que introducir genes de resistencia correspondientes para tales razas (Vázquez y de Romano, 2006; Bazzalo et al., 2016). La expansión de la siembra directa podría ser la causa del aumento de la prevalencia del Mildiu del girasol. Además, la aplicación de Metalaxyl a las semillas de variedades sin resistencia puede haber favorecido la aparición de razas resistentes a este oomiceticida (Quiróz, 2014). Desde 2013, el gen de resistencia Pl15 ha sido superado, lo que indica la aparición de una nueva raza. El trabajo en la identificación de genes de virulencia podría ayudar al desarrollo de híbridos resistentes (Ma et al., 2020; Bazzalo et al., 2016).
Actualmente en la Argentina se ha comprobado la presencia de razas de Plasmopara halstedii con resistencia al metalaxil. Bazzalo y Piubello (2015) aislaron variantes de P. halstedii resistentes al metalaxil de las localidades de Madariaga, Balcarce (Provincia de Buenos Aires) y Villa Ocampo (Prov. de Santa Fe). Las investigadoras, mediante la inoculación de un set de líneas públicas, determinaron que dichos aislamientos pertenecían a las razas 730, 710 y 770. También confirmaron que los genes de resistencia Pl6 y Pl8 continúan siendo efectivos frente a estos aislamientos.
Trojanová et al. (2017) han realizado una revisión sobre metodologías de detección de virulencia y caracterización de razas de Plasmopara halstedii, y evaluación de resistencia en girasol.
.
- 01 Signo del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 02 Síntomas del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 03 Signo del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 04 Síntomas del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 05 Signo del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 06 Signo del Mildew del girasol, causado por Plasmopara halstedii. Autor: Dirceu Gassen
- 01 Síntomas de Mildew del girasol. Se observa clorosis en la hoja, atrofia y cabeza (capítulo) respingona. Autor: Hedvig Komjati, EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
- 02 Esporulación de P. halstedii sobre cotiledones de girasol. Autor: EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
- 03 Esporulación de P. halstedii a nivel del suelo en el tallo de una plántula de girasol en una cámara climática. Autor: Aranka Kormany, EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
- 04 Crecimiento del Mildew del girasol, desarrollado en la superficie inferior de la hoja (A), correspondiente a las áreas cloróticas en la cara superior de la hoja (B). Autor: The Food and Environment Research Agency (Fera), Crown Copyright.
- 05 Planta de girasol severamente afectada por el Mildew o enanismo del girasol, con síntomas de crecimiento retardado, clorosis foliar y capítulo horizontal. Autor: F. Viranyi, USDA, USA, EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
- 06 Síntomas de enanismo en un lote severamente afectado por el Mildew del girasol.
- 07 Síntomas de enanismo del girasol a campo, causado por el oomycete Plasmopara halstedii. Autor: Markell, NDSU, USA
- 08 Síntomas del Mildew o enanismo del girasol a campo, causado por P. halstedii. Autor: Markell, NDSU, USA
- 09 Signo (esporulación) de P. halstedii sobre el envés de hojas de girasol. Autor: Markell, NDSU, USA
.
.
Epidemiología
Las fuentes de inóculo son el suelo infestado con oosporas, los restos de cultivos infestados de años anteriores y las semillas portadoras de oosporas. Las oosporas que sobreviven en el suelo sirven como inóculo primario para plántulas jóvenes de girasol, y tienen la capacidad de sobrevivir hasta 8-10 años en el suelo. Las oosporas se producen justo debajo de la epidermis de las plantas infectadas y son más comunes en las raíces que en las hojas. Cuando los suelos están frescos y saturados de agua, las oosporas germinan y forman zoosporangios, que dan lugar a zoosporas móviles asexuales. Las plantas que se desarrollan a partir de semillas infectadas usualmente permanecen asintomáticas pero pueden infestar el suelo con propágulos del pseudohongos como esporangios u oosporas (Sackston, 1981). El inóculo transmitido por semillas es difícil de detectar incluso con procedimientos de laboratorio que consumen tiempo. El método ELISA fue reportado como una herramienta exitosa para detectar la infección de P. halstedii transmitida por semilla de girasol (Liese et al., 1982). Recientemente, investigadores argentinos han desarrollado un método de detección molecular basado en la PCR para la detección del patógeno en semilla (Martínez et al., 2021).
Los síntomas de la enfermedad se manifiestan en todas las fases de crecimiento vegetativo, pero los daños son más graves cuanto más temprana ocurra la infección, ya que es cuando se produce el enanismo de las plantas de girasol, lo que puede disminuir severamente el rendimiento. Las plantas a menudo se secan y mueren o continúan desarrollándose con una cabeza erecta y horizontal con pocas semillas. Por lo tanto, este aspecto de la enfermedad se la considera de tipo monocíclica. La infección sistémica se produce cuando las zoosporas infectan la plántula de girasol antes de que la raíz alcance 5 cm de largo. Las plantas de girasol que sobreviven a esta infección inicial producen zoosporangios blancos en la parte inferior de las áreas cloróticas de las hojas. Los zoosporangios de P. halstedii pueden ser diseminados por el viento, causando infecciones secundarias en las partes aéreas de la planta, generalmente localizadas, por esporangios dispersos, o incluso por semillas cuando la semilla producida por plantas infectadas transporta micelio y/o oosporas del patógeno. Estas lesiones son generalmente cloróticas y de forma angular, estando limitadas por las nervaduras de las hojas. Directamente debajo de las lesiones amarillas en el envés de las hojas se encuentra la esporulación algodonosa blanca del oomicete (mildew), que es una característica de diagnóstico (signo). Las plantas son susceptibles a la infección secundaria durante períodos de tiempo más prolongados que con las infecciones radiculares sistémicas, pero rara vez provocan una pérdida de rendimiento. La importancia de los esporangios transmitidos por el viento en el inicio de la enfermedad suele ser baja. Además, se descubrió que la infección secundaria por zoosporangios incita a la infección latente en plantas sin síntomas de la enfermedad durante la estación de crecimiento, pero que produce semillas que pueden portar el pseudohongo en una forma latente (Sackston, 1981).
Cuando se producen lluvias intensas o encharcamientos, con temperaturas frescas, las oosporas germinan produciendo zoosporangios que liberan esporas flageladas (zoosporas). Éstas se desplazan (distancias muy cortas) hasta alcanzar las raíces o el hipocótilo de las plantas y así penetran e invaden la planta, produciendo, en híbridos muy susceptibles, muerte de pre-emergencia. Las plantas que sobreviven ó que son atacadas en el estado de cotiledones ó primeras hojas pueden presentar enanismo, como consecuencia de una infección sistémica. Cuanto más temprano sea el ataque y/ó más lento sea el crecimiento de las plantas, más grave será la enfermedad y, por lo tanto, mayores los daños y pérdidas ocasionadas.
La humedad y la temperatura son los factores ambientales más importantes que afectan la infección y la diseminación. Temperaturas por debajo de 16°C, humedad relativa elevada (>70%) y lluvias acumuladas en estadios tempranos son factores ambientales predisponentes. Las zoosporas requieren agua libre para retener la viabilidad y avanzar hacia los sitios de infección. En consecuencia, la lluvia o el riego intensivo serán un requisito previo para el inicio de la infección primaria, particularmente durante las primeras 2-3 semanas críticas después de la siembra (Zimmer y Hoes, 1978; Kolte, 1985). La edad de las plantas y el tejido del hospedante también son importantes para determinar la susceptibilidad del girasol a la infección sistémica por P. halstedii (Sackston, 1981). Desde un punto de vista práctico, se puede afirmar que cuanto antes se produzca la infección en la estación de crecimiento del cultivo, más grave será la enfermedad en la planta.
.
- 01 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 02 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 03 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 04 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 05 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 06 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 07 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 08 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 09 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base, y enanismo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 10 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen enanismo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 11 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen enanismo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 12 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen enanismo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 13 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 14 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 15 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 16 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 21 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 22 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 17 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 18 Síntomas de enanismo causados por el Mildew o enanismo del girasol. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 19 Síntomas de enanismo causados por el Mildew o enanismo del girasol. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 20 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 23 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Autor: Ing. Raúl Zapata.
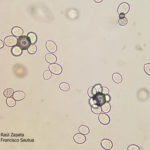
- 24 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 25 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 26 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 27 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 28 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 29 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 30 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 31 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 32 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
- 33 Eflorescencia blanquecina del Mildew o enanismo del girasol, constituida por Zoosporangios. Autor: Ing. Raúl Zapata.
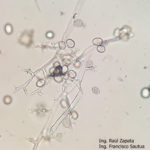
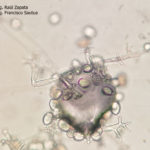
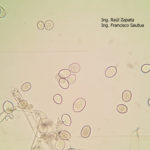
- 34 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 35 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 36 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 37 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 38 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 39 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
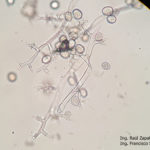
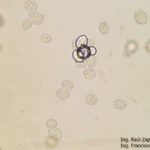
- 40 Zoosporangios de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 41 Zoosporangios de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 42 Zoosporangios de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 43 Zoosporangios de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
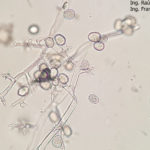
- 44 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 45 Zoosporangios de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
- 46 Zoosporangios sobre zoosporangióforos de Plasmopara halstedii. Autor: Ing. Raúl Zapata.
.
Daños
Las pérdidas de rendimiento por mildiu del girasol dependen de numerosos factores, incluyendo la incidencia (porcentaje de plantas infectadas en el campo) y distribución del patógeno, el clima de la temporada y el momento de mayor infección respecto a la fenología del cultivo. Las plantas que sobreviven a menudo pueden compensar las que mueren prematuramente después de una infección temprana. Sin embargo, las pérdidas de producción también pueden volverse más notorias y graves cuando se infectan grandes áreas de campo, la combinación de la mortalidad de las plantas, semillas más ligeras y menor número de granos producidos por las plantas supervivientes y reducción del contenido de aceite.
Por lo tanto, el nivel de daño dependerá de la susceptibilidad del híbrido de girasol sembrado, las razas predominantes en la región de producción, de la cantidad (densidad) de inóculo disponible, el nivel de agua en el suelo, la edad de la planta en el momento de la infección, condiciones ambientales predisponentes y su velocidad de crecimiento. Cuando ocurre enanismo por infecciones tempranas, las plantas son improductivas. Ante ataques severos las pérdidas pueden llegar al 100%. Se han registrado pérdidas totales por damping off (reducción de stand de plantas).
.
Manejo Integrado
* Híbridos resistentes: se han introducido genes de resistencia mayores (apilamiento). Asimismo, se han identificado numerosas razas del patógeno con capacidad de “quebrar” genes mayores. Es conveniente consultar las publicaciones anuales de comportamiento de híbridos RED INTA-ASAGIR / Red Nacional de cultivares de girasol (ej. Campaña 2019- 2020).
* Usar sólo semillas sanas, producidas en zonas libres de la enfermedad, lo cual permitirá, en caso de ausencia del parásito, no introducirlo y, en todos los casos, limitar la introducción de aislamientos más agresivos o más virulentos, o resistentes a los oomiceticidas.
* Tratamientos de semillas: existen oomiceticidas específicos como el metalaxil, que protege a la planta de girasol durante los primeros estadios vegetativos (período de mayor susceptibilidad). Sin embargo, se han detectado cepas resistentes a metalaxil (Erreguerena et al, 2013; Bannister et al., 2016). El principio activo oomiceticida más usado actualmente es oxathiapiprolin.
* Evitar siembras con temperaturas frescas en condiciones favorables para el patógeno.
* Control de girasoles silvestres y voluntarios hospedantes del patógeno: la eliminación de plantas guachas puede reducir el inóculo de manera efectiva. Además, la eliminación de girasoles silvestres y voluntarios puede reducir el inóculo de otras enfermedades como la roya y Sclerotinia.
* Evitar lotes con historia de la enfermedad.
X La rotación tiene un efecto mínimo en el manejo del mildiu del girasol. Las oosporas pueden sobrevivir en el suelo hasta 10 años, lo que hace que las prácticas de rotación no sean efectivas o muy limitadas.
X Fungicidas foliares: las aplicaciones foliares de oomiceticidas no son efectivas para el control de la infección sistémica del mildiú y no se recomienda su uso. El uso de oomiceticidas foliares para infecciones secundarias no es económico ya que no se produce pérdida de rendimiento.
.
La enfermedad es extremadamente difícil o imposible de erradicar una vez que se establece en un área (Trojanová et al., 2017). Existen híbridos de girasol resistentes al mildiú, pero al mismo tiempo en la naturaleza se forman razas patógenas nuevas del pseudohongo. Los fungicidas (oomyceticidas) con propiedades definidas sistémicas y de larga duración (por ejemplo, metalaxil o compuestos relacionados) son importantes para controlar la enfermedad (Virányi y Oros, 1991). Incluso con el uso de cultivares resistentes, se recomienda encarecidamente el uso de fungicidas (oomyceticidas) aplicados a la semilla para prevenir la infección subterránea de las plántulas (Virányi, 1978).
En Australia, donde P. halstedii está ausente en girasol, y es tratado como una importante plaga cuarentenaria, las semillas importadas están sujetas a tratamiento con agua caliente, se espolvorean con fungicida y crecen en contenedores durante al menos dos temporadas (Anon, 1981).
.
- Fuente: Ing. Agr. Mariano Cracogna, Ing. Agr. Sebastian Zuil, INTA Reconquista
.
.
- 01 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 02 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 03 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base; y enanismo. Se observa en el envés de la hoja el signo: una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 04 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas y enanismo. Comparar con altura y tamaño normal de plantas sanas. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 05 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas y enanismo. Comparar con altura y tamaño normal de plantas sanas. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 06 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas y enanismo. Comparar con altura y tamaño normal de plantas sanas. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 07 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas y enanismo. Comparar con altura y tamaño normal de plantas sanas. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 08 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas y enanismo. Comparar con altura y tamaño normal de plantas sanas. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata, Ing. Francisco Sautua.
- 02 Síntomas del Mildew o enanismo del girasol. Los síntomas incluyen clorosis alrededor de las nervaduras principales de las hojas comenzando desde la base. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
- 21 Signo del Mildew o enanismo del girasol. Se observan en el envés de las hojas una eflorescencia blanquecina constituida por las fructificaciones del pseudohongo. Híbrido Dow MG 303 CP. Autor: Ing. Raúl Zapata.
.
Bibliografía
(2014), PM 7/85 (2) Plasmopara halstedii. EPPO Bull, 44: 350-359. doi:10.1111/epp.12160
ASOCIACION ARGENTINA DE GIRASOL (2015) TALLER SOBRE DOWNY MILDEW (Plasmopara halstedii) EN GIRASOL. LINK
, (2012) Emerging virulence arising from hybridisation facilitated by multiple introductions of the sunflower downy mildew pathogen Plasmopara halstedii. Fungal Genet. Biol. 49: 847–855. doi: 10.1016/j.fgb.2012.06.012
Anon (1981) Sunflower downy mildew. Plant Quarantine Leaflet No. 13. Commonwealth Department of Health, Canberra, Australia.
Bazzalo E, Piubello S (2015) Variante de Plasmopara halstedii tolerantes a Metalaxyl. TALLER SOBRE DOWNY MILDEW (Plasmopara halstedii) EN GIRASOL, ASOCIACION ARGENTINA DE GIRASOL. Link
Bazzalo E, Huguet N, Romano C, et al. (2016) History and present state of downy mildew in Argentina. In: Proc. 19th Int. Sunflower Conf. Edirne, TUR, pp. 828–832
Castaño FD (2018) The sunflower crop in Argentina: past, present and potential future. OCL 25 1: D105. doi: 10.1051/ocl/2017043
(2009) Characterization of a Plasmopara species on Ambrosia artemisiifolia, and notes on P. halstedii, based on morphology and multiple gene phylogenies. Mycol. Res. 113: 1127-1136. doi: 10.1016/j.mycres.2009.07.010
(2015) Host jumps and radiation, not co‐divergence drives diversification of obligate pathogens. A case study in downy mildews and Asteraceae. PLoS One 10:e0133655. doi: 10.1371/journal.pone.0133655
Cracogna M, Zuil S (2016) Enanismo del girasol o Mildiu (Plasmopara halstedii). Editor/es: Ministerio de Agroindustria de la Nacion, INTA Reconquista.
, , et al. (2008) Single nucleotide polymorphisms reveal multiple introductions into France of Plasmopara halstedii, the plant pathogen causing sunflower downy mildew. Infection, Genetics and Evolution 8: 534–40. doi: 10.1016/j.meegid.2008.02.012
Drábková Trojanová Z, Sedlářová M, Gulya TJ, Lebeda A (2017) Methodology of virulence screening and race characterization of Plasmopara halstedii, and resistance evaluation in sunflower – a review. Plant Pathology 66(2): 171–185. doi: 10.1111/ppa.12593
Drábková Trojanová Z, Sedlářová M, Pospíchalová R, Lebeda A (2018) Pathogenic variability of Plasmopara halstedii infecting sunflower in the Czech Republic. Plant Pathology 67: 136–144. doi: 10.1111/ppa.12722
(2014) Plasmopara invertifolia sp. nov. causing downy mildew on Helichrysum bracteatum (Asteraceae). Mycol. Prog. 13: 285-289. doi: 10.1007/s11557-013-0913-7
(1883) Note on some species in the third and eleventh centuries of Ellis’s North American Fungi. Proceedings of the American Academy of Arts and Sciences 18, 71–3.
Gascuel Q, Martinez Y, Boniface M-C, Vear F, Pichon M, Godiard L (2015) The sunflower downy mildew pathogen Plasmopara halstedii. Molecular Plant Pathology 16: 109–122. doi: 10.1111/mpp.12164
Gascuel Q, Bordat A, Sallet E, Pouilly N, Carrere S, Roux F, et al. (2016) Effector Polymorphisms of the Sunflower Downy Mildew Pathogen Plasmopara halstedii and Their Use to Identify Pathotypes from Field Isolates. PLoS ONE 11(2): e0148513. doi: 10.1371/journal.pone.0148513
(2007) How do obligate parasites evolve? A multi-gene phylogenetic analysis of downy mildews. Fungal Genet. Biol. 44: 105-122. doi: 10.1016/j.fgb.2006.07.005
Göre ME (2009) Epidemic outbreaks of downy mildew caused by Plasmopara halstedii on sunflower in Thrace, part of the Marmara region of Turkey. Plant Pathology 58: 396-396. doi: 10.1111/j.1365-3059.2008.01975.x
Gulya TJ, Rashid KY, Masirevic SM (1997) Sunflower diseases. In: Schneiter AA (ed), Sunflower technology and production: 263-276. American Society of Agronomy, Madison USA.
(2002) First report of cross-infectivity of Plasmopara halstedii from marshelder to sunflower. Plant Disease 86: 919. doi: 10.1094/PDIS.2002.86.8.919B
Gulya T, Markell S, McMullen M, Harveson R, Osborne L (2011) Emergence of new virulent races of Plasmopara halstedii inciting downy mildew on sunflower in the United States. Phytopathology 101:S2.3. Abstract In: North Central Division Meeting Abstracts, June 15-17, 2011, Omaha, NE. S2.3-4. Link
Gulya T, Kandel H, McMullen M, Knodel J, Berglund D, Mathew F, Lamey AH, Nowatzki J, Markell S (2013) Prevalence and Incidence of Sunflower Downy Mildew in North Dakota Between 2001 and 2011. Plant Health Progress (online). doi: 10.1094/PHP-2013-0522-01-RS
Gulya T, Harveson R, Mathew F, Block C, Thompson S, Kandel H, Berglund D, Sandbakken J, Kleingartner L, Markell S (2019) Comprehensive Disease Survey of U.S. Sunflower: Disease Trends, Research Priorities and Unanticipated Impacts. Plant Disease 103(4): 601-618. doi: 10.1094/PDIS-06-18-0980-FE
, (2010) Mapping and tagging of simply inherited traits. In: Hu J, Seiler G, Kole C, eds. Genetics, Genomics and Breeding of Sunflower. Enfield, NH, USA: Science Publishers, 111–34.
Kolte SJ (1985) Diseases of annual edible oilseed crops, Vol. 3, sunflower, safflower & nigerseed diseases. CRC Press, Inc., Boca Raton, USA.
(2007) Characteristics of a Plasmopara angustiterminalis isolate from Xanthium strumarium. Eur. J. Plant Pathol. 119: 421-428. doi: 10.1007/s10658-007-9178-9
Leite RMVBC, Henning AA, Rodrigues SR, Oliveira MF (2007) Detecção e variabilidade de Plasmopara halstedii no Brasil e avaliação da resistência de genótipos de girassol ao míldio. Summa Phytopathologica 33(4): 335-340. doi: 10.1590/S0100-54052007000400003
(1965) Plasmopara halstedii and other diseases on Dimorphotheca. Plant Dis. Rep. 49: 940-942.
Leppik EE (1966) Origin and specialization of Plasmopara halstedii complex on Compositae. FAO Plant Protection Bulletin 14: 72-76.
Liese, A.R.; Gotlieb, A.R.; Sackston, W.E. (1982) Use of enzyme-linked immunosorbent assay (ELISA) for the detection of downy mildew (Plasmopara halstedii) in sunflower. Proceedings of the 10th International Sunflower Conference, Surfers Paradise, pp. 173-175.
Ma G, Song Q, Li X, Qi L (2020) High-Density Mapping and Candidate Gene Analysis of Pl18 and Pl20 in Sunflower by Whole-Genome Resequencing. International Journal of Molecular Sciences 21(24): 9571. doi: 10.3390/ijms21249571
Martínez AL, Quiroz FJ, Carrera AD (2021) Detection of Plasmopara halstedii in sunflower seeds: A case study using molecular testing. Journal of the Saudi Society of Agricultural Sciences. doi: 10.1016/j.jssas.2021.04.007
, , , , , , , (2006) An overview of sunflower downy mildew. Phytoma—La Défense des Végétaux, 589, 34–38.
Molinero-Ruiz ML, Melero-Vara JM, Domínguez J (2003) Inheritance of resistance to two races of sunflower downy mildew (Plasmopara halstedii) in two Helianthus annuus L. lines. Euphytica 131(1): 47–51. doi: 10.1023/A:1023063726185
Nisha N, Körösi K, Perczel M, Yousif AIA, Bán R (2021) First Report on the Occurrence of an Aggressive Pathotype, 734, of Plasmopara halstedii Causing Sunflower Downy Mildew in Hungary. Plant Disease. doi: 10.1094/PDIS-05-20-1054-PDN
Nisha N, Vinogradov S, Körösi K, et al. (2023) Assessing the Sensitivity of Plasmopara halstedii Isolates to Mefenoxam through Host Responses. Microorganisms 11(4): 821. doi: 10.3390/microorganisms11040821
Novotel’nova NS (1966) Downy mildew of sunflower. 150 pp. Nauka, Moscow, Russia.
PM 7/85 (2) Plasmopara halstedii. Bulletin OEPP/EPPO Bulletin 44(3): 350–359. doi: 10.1111/epp.12160
, , , , (2014) Pl17 is a novel gene independent of known downy mildew resistance genes in the cultivated sunflower (Helianthus annuus L.). Theoretical and Applied Genetics 128: 757–67. doi: 10.1007/s00122-015-2470-8
Qi LL, Foley ME, Cai XW, et al. (2016) Genetics and mapping of a novel downy mildew resistance gene, Pl 18 , introgressed from wild Helianthus argophyllus into cultivated sunflower (Helianthus annuus L.). Theoretical and Applied Genetics 129(4): 741–752. doi: 10.1007/s00122-015-2662-2
Quiróz F (2014) Éxitos pasados, trabajos en marcha y desafíos futuros en materia de enfermedades. LINK
Radwan O (2014) Recent advances for developing resistance against Plasmopara halstedii in sunflower. In: Arribas JI (Eds.) Sunflowers: Growth and Development, Environmental Influences and Pests/Diseases. pp. 187-200. Nova Science Publishers. ISBN: 978-1-63117-347-9
Rivera Y, Salgado-Salazar C, Gulya TJ, Crouch JA (2016) Newly Emerged Populations of Plasmopara halstedii Infecting Rudbeckia Exhibit Unique Genotypic Profiles and Are Distinct from Sunflower-Infecting Strains. Phytopathology 106(7): 752-761. doi: 10.1094/PHYTO-12-15-0335-R
Roeckel-Drevet P, Tourvieille J, Gulya TJ, Charmet G, Nicolas P, Tourvieille de Labrouhe D (2003) Molecular variability of sunflower downy mildew, Plasmopara halstedii, from different continents. Canadian Journal of Microbiology 49: 492–502. doi: 10.1139/W03-062
, , (2011) Phylogenetic investigations in the genus Pseudoperonospora reveal overlooked species and cryptic diversity in the P. cubensis species cluster. European Journal of Plant Pathology 129: 135–46. doi: 10.1007/s10658-010-9714-x
Sackston WE (1981) Downy mildew of sunflower. In: The downy mildews (Ed. by Spencer DM), pp 545-575. Academic Press, London, UK.
(2011) Cryptic diversity of Plasmopara viticola (Oomycota, Peronosporaceae) in North America. Org. Divers. Evol. 11: 3-7. doi: 10.1007/s13127-010-0035-x
Sedlářová M, Pospíchalová R, Drábková TRojanová Z, Bartůšek T, Slobodianová L, Lebeda A (2016) First Report of Plasmopara halstedii New Races 705 and 715 on Sunflower from the Czech Republic. Plant Protect. Sci. 52(3): 182–187. doi: 10.17221/7/2016-PPS
Sharma R, Xia X, Cano LM, et al. (2015) Genome analyses of the sunflower pathogen Plasmopara halstedii provide insights into effector evolution in downy mildews and Phytophthora. BMC Genomics 16: 741. doi: 10.1186/s12864-015-1904-7
Spring O, Zipper R (2016) Asexual Recombinants of Plasmopara halstedii Pathotypes from Dual Infection of Sunflower. PLoS ONE 11(12): e0167015. doi: 10.1371/journal.pone.0167015
(2009) Identity of the downy mildew pathogens of basil, coleus, and sage with implications for quarantine measures. Mycol. Res. 113: 532-540. doi: 10.1016/j.mycres.2008.12.005
Tourvieille de Labrouhe D, Gulya TJ, Masirevic S, Penaud A, Penaud KY, Viranyi F (2000) New nomenclature of Races of Plasmopara halstedii (Sunflower Downy Mildew). Conference: 15th International Sunflower ConferenceAt: Toulouse (France) Volume: I-61. LINK
, , (2000) Le Mildiou du Tournesol. Points Techniques. Paris: INRA and CETIOM.
, , , , (2008) Quantitative resistance to downy mildew (Plasmopara halstedii) in sunflower (Helianthus annuus). Euphytica 164: 433–444. doi: 10.1007/s10681-008-9698-1
Trojanová Z, Sedlářová M, Gulya TJ, Lebeda A (2017) Methodology of virulence screening and race characterization of Plasmopara halstedii, and resistance evaluation in sunflower – a review. Plant Pathology 66: 171-185. doi: 10.1111/ppa.12593
Vázquez A, de Romano A (2006) Sunflower crop in Argentina to date. Helia 44: 159–164. doi: 10.2298/hel0644159v
, , et al. (2008) Origins of major genes for downy mildew resistance in sunflower. In: Proceedings of 17th International Sunflower Conference. Cordoba, Spain: Consejeria de Agricultura y Pesca, 125–30.
Vear F, Tourvieille de Labrouhe D, Roche S, et al., (2012) Genetic analysis of the sunflower downy mildew resistance gene Pl2. Conference: 18th International Sunflower ConferenceAt: Mar del Plata, Volume: 01-VC-32. LINK
Virányi F (1978) Harmful incidence of Plasmopara halstedii in downy mildew «resistant» sunflowers. Phytopathologische Zeitschrift 91: 362-364.
(1984) Recent research on the downy mildew of sunflower in Hungary. Helia 7: 35-38.
Virányi F (1988) Factors affecting oospore formation in Plasmopara halstedii. Proceedings of the 12th International Sunflower Conference, Novi Sad 2, 32-37.
Virányi F, Oros G (1991) Developmental stage response to fungicides of Plasmopara halstedii (sunflower downy mildew). Mycological Research 95: 199-205. doi: 10.1016/S0953-7562(09)81012-5
Virányi F (2002) The Sunflower — Plasmopara Halsted II Pathosystem: Natural and Artificially Induced Coevolution. In: Spencer-Phillips P.T.N., Gisi U., Lebeda A. (eds) Advances in Downy Mildew Research. Springer, Dordrecht. doi: 10.1007/0-306-47914-1_6
Virányi F, Spring O (2011) Advances in sunflower downy mildew research. European Journal of Plant Pathology 129(2): 207-220. doi: 10.1007/s10658-010-9683-0
, , (2015) Recent changes in the pathogenic variability of Plasmopara halstedii (sunflower downy mildew) populations from different continents. Helia 38: 149–62. doi: 10.1515/helia-2015-0009
Voglmayr H (2008) Progress and challenges in systematics of downy mildews and white blister rusts: new insights from genes and morphology. Eur J Plant Pathol 122: 3. doi: 10.1007/s10658-008-9341-y
(2000) Study on an Ambrosia isolate of Plasmopara. Helia 23: 19-24.
Wieckhorst S, Bachlava E, Dußle CM, et al. (2010) Fine mapping of the sunflower resistance locus PlARG introduced from the wild species Helianthus argophyllus. TAG Theoretical and Applied Genetics 121(8): 1633-1644. doi: 10.1007/s00122-010-1416-4
Wieckhorst S (2011) Characterization of the PlARG locus mediating resistance against Plasmopara halstedii in sunflower. PhD Thesis, Universität München. LINK
, (1927) Plasmopara downy mildew of cultivated sunflowers. American Journal of Botany 14, 551–3.
(1971) A serious outbreak of downy mildew in principal sunflower production in the United States. Plant Disease Reporter 55: 11–2.
Zimmer DE, Hoes JA (1978) Diseases. In: Sunflower science and technology (Ed. by Carter, J.F.), pp 225-262. American Society of Agronomy, Madison, USA.