.
Condición fitosanitaria: Presente
Grupo de cultivos: Hortícolas
Especie hospedante: Tomate (Solanum lycopersicum L.)
Rango de hospedantes: específico / estrecho. Las especies de Fusarium oxysporum causan marchitamiento en más de 150 hospedantes y varían con formae speciales específicas (Bertoldo et al., 2015; Jangir et al., 2021).
Etiología: Hongo. Necrotrófico
Agente causal: Fusarium oxysporum f. sp. lycopersici W.C. Snyder & H.N. Hansen, (1940)
Taxonomía: Fungi > Ascomycota > Pezizomycotina > Sordariomycetes > Hypocreales > Nectriaceae > Fusarium > Fusarium oxysporum species complex > Fusarium oxysporum
.
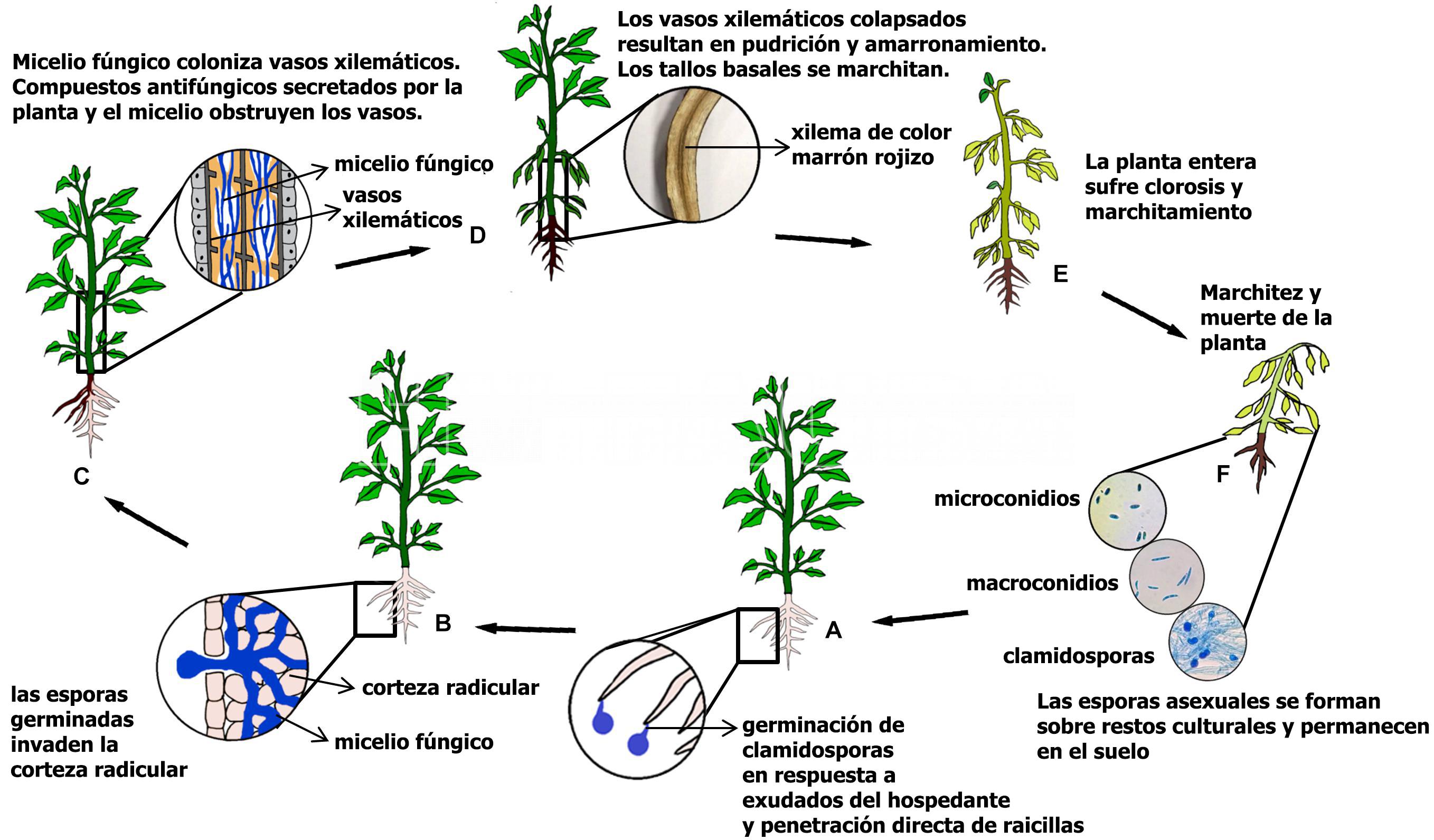
Fusarium oxysporum es un hongo «habitante» del suelo, que produce dos tipos de esporas asexuales: macroconidios, microconidios y estructuras de resistencia, las clamidosporas.
Los macroconidios son rectos a ligeramente curvados, delgados y de paredes delgadas, generalmente con tres o cuatro septos, una célula basal en forma de pie y una célula apical cónica y curva. Generalmente se producen a partir de fialidas sobre conidióforos por división basípeta. Son importantes en la infección secundaria.
Los microconidios son elipsoidales y no tienen tabique o tienen uno solo. Se forman a partir de fialidas en falsas cabezas por división basípeta. Son importantes en la infección secundaria.
Las clamidosporas son globosas y tienen paredes gruesas. Se forman a partir de hifas o, alternativamente, mediante la modificación de células de hifas. Son importantes como órganos de resistencia en suelos donde actúan como inóculos en la infección primaria.
Se desconoce el teleomorfo o etapa reproductiva sexual de F. oxysporum.
A pesar de que las especies de Fusarium tienen un amplio rango de hospedantes, las especies individuales a menudo se asocian con solo una o unas pocas plantas hospedantes (Li et al., 2020). Así por ejemplo, las poblaciones de F. solani patógenas en plantas se han dividido en 12 formae speciales y dos razas (Šišić et al., 2018; Jangir et al., 2021). Fusarium oxysporum es la especie de Fusarium de mayor importancia económica y más común. Se sabe que este hongo del suelo alberga cepas patógenas (de vegetales, animales y humanos) y no patógenas. Sin embargo, en su concepto actual, F. oxysporum es un complejo de especies que consta de numerosas especies crípticas. La identificación y el nombre de estas especies crípticas se complica por los múltiples sistemas de clasificación subespecíficos y la falta de material vivo de tipo ex que sirva como punto de referencia básico para la inferencia filogenética. Por lo tanto, para avanzar y estabilizar la posición taxonómica de F. oxysporum como especie y permitir el nombramiento de las múltiples especies crípticas reconocidas en este complejo de especies, Lombard et al. (2019) designaron un epitipo para F. oxysporum. Utilizando la inferencia filogenética de múltiples locus y las diferencias morfológicas sutiles con el epitipo recién establecido de F. oxysporum como punto de referencia, pudieron resolver 15 taxones crípticos, descriptas como especies.
Las cepas hospedante-específicas de F. oxysporum se asignan a formae speciales (f. sp.), siguiendo una propuesta de Snyder y Hansen (1940) para tratar tales formas biológicas como variantes de una sola especie, en lugar de taxones separados. La designación de forma specialis no tenía valor taxonómico y era simplemente una agrupación de conveniencia que facilitaba la comunicación entre los fitopatólogos. Sin embargo, estar subsumido bajo un nombre científico implicaba cepas en la misma forma specialis constituía un agrupamiento natural, pero finalmente se hizo evidente que este sistema no es perfecto (O’Donnell et al., 1998; Baayen et al., 2000).
El género Fusarium es una de las especies más complejas y adaptativas en el complejo de especies Eumycota y Fusarium oxysporum (Fo) que incluye patógenos de plantas, animales y humanos y una amplia gama de no patógenos (Nirmaladevi et al., 2016; Gordon, 2017; Jelinski et al., 2017; Jangir et al., 2021).
Actualmente se han identificado tres razas de Fusarium oxysporum f. sp. lycopersici (Fol), R 1, 2, 3 (también conocidas como razas 0, 1, 2) y se han identificado tres loci correspondientes en tomate, I-1, I-2 e I-3, que confieren resistencia al patógeno a través de varios genes dominantes importantes (Panthee y Chen, 2010, Scott et al., 2004). Tres genes de avirulencia (avr1, avr2 y avr3) transportados en varias combinaciones en diferentes razas Fol son reconocidos por cultivares de tomate que poseen los genes de resistencia correspondientes, y que desencadenan respuestas de defensa contra el patógeno (Andolfo et al., 2014). Fol se ha caracterizado además por análisis de grupos de compatibilidad vegetativa (VCG) que determina la compatibilidad de apareamiento parasexual en el hongo. Los VCG 0030 a 0032 contienen las razas 1 y 2, y la raza 3 está asignada a los VCG 0030 y 0033 (Elias y Schneider, 1991, Marlatt et al., 1996).
Las cepas toxigénicas poseen el gen Fum1, lo que indica que las cepas que infectan los cultivos de tomate tienen el potencial de producir fumonisina (Nirmaladevi et al., 2016).
.
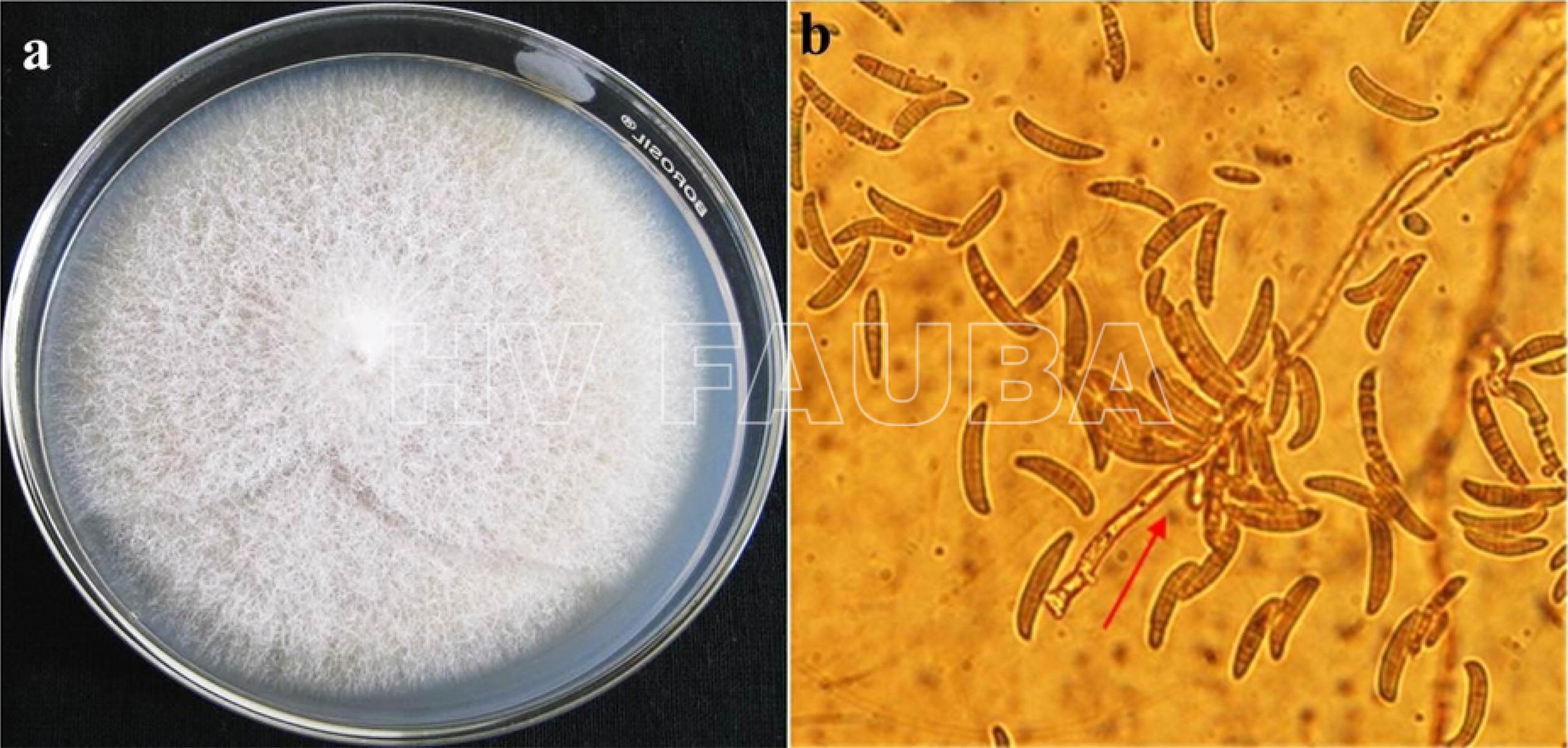
- Características culturales y morfológicas de Fusarium oxysporum f. sp. lycopersici. (a) Colonia de F. oxysporum en agar (PDA); (b) Vista microscópica de macroconidios de F. oxysporum f. sp. lycopersici, comúnmente tri septados y se observa la unión de los macroconidios al micelio. Autor: Srinivas et al., 2019.
.
.
Antecedentes
Las enfermedades causadas en el tomate por Fusarium spp., especialmente el marchitamiento y la pudrición de la corona y la raíz, han estado y siguen estando entre las enfermedades de las plantas más intensamente estudiadas. La marchitez por Fusarium, causada por F. oxysporum f. sp. lycopersici, se describió por primera vez hace más de 100 años en el Reino Unido (Massee, 1895), y la pudrición de corona y raíz, causada por F. oxysporum f. sp. radicis-lycopersici Jarvis y Shoemaker (Forl), se observó por primera vez en 1969 en Japón (Sato y Araki, 1974). En la actualidad, ambos patógenos causan importantes daños y pérdidas a campo y en invernadero, y siguen siendo uno de los principales factores limitantes para la producción de tomate, cuando están presentes en el lote. Las pérdidas por marchitamiento pueden ser muy elevadas dadas las combinaciones de patógeno virulento / hospedante susceptible (Walker, 1971). En la India se informó recientemente de pérdidas de rendimiento de hasta un 45% (Ramyabharathi et al., 2012). Se han estimado pérdidas de pudrición de corona y raíz en tomate de invernadero de hasta 90% y 95% en Túnez y Canadá, respectivamente, y la enfermedad se ha observado con una incidencia del 100% en campos en los EE. UU. (Hibar et al., 2007, Jarvis y col., 1983, McGovern y col., 1998).
.
Síntomas
Fusarium oxysporum f. sp. lycopersici penetra en la planta de tomate, colonizando y necrosando el tejido vascular, dejandolo de color marrón oscuro, y esta decoloración se extiende hasta el ápice, provocando que las plantas se marchiten, colapsen y mueran. El marchitamiento causado por este hongo es el resultado de una combinación de diversas actividades fisiológicas, incluida la acumulación de micelios fúngicos en el xilema y sus alrededores, la producción de micotoxinas, la inactivación de las defensas del hospedante y la producción de tilosas. Sin embargo, los síntomas de marchitez son variables.
.
- Síntomas de marchitamiento por Fol. Autor: Srinivas et al., 2019.
.
Se identificaron varios genes de Fol cuyos productos proteicos se liberan durante la infección en las células hospedantes (Schmidt et al., 2013, Schmidt et al., 2016). Las dos proteínas producidas en el xilema están codificadas por los genes SIX1 y SIX2, los que se encuentran a 8 kb entre sí, en uno de los cromosomas más pequeños (Boix-Ruíz et al., 2015, Rep et al., 2004). El producto SIX1 es una pequeña proteína rica en cisteína que se ha revelado como esencial para la virulencia de Fol (Selim et al., 2015). Se identificaron ocho proteínas fúngicas de la savia del xilema de una planta enferma y el material genético de estas proteínas está presente en la región similar del cromosoma (SIX1, SIX2) (Maldonado et al., 2018, Sasaki et al., 2015). Dentro del mismo cromosoma está presente un homólogo de SIX1, SIX1-H, que codifica un homólogo de salicilato hidroxilasa, y otro gen, SIX3, que codifica una proteína secretada por xilema. SIX1, SIX2, SIX3 y SHH1 fueron exclusivos de los aislados Fol. A pesar de su origen polifilético, todos los aislamientos Fol de ese estudio tuvieron una región genómica que contenía al menos 8 kb de genes idénticos y que comprenden SIX1, SIX2 y SHH1, pero faltaban en otros aislamientos no patógenos y formae speciales. Los factores de virulencia fúngica, como los genes SIX1, SIX2 y SIX3 codifican una proteína, que se secreta en la savia del xilema y puede contribuir al marchitamiento de la planta por colonización fúngica. Esta región genómica existió inicialmente en el ancestral Fo y posteriormente desapareció en todas las líneas clonales excepto Fol (Jelinski et al., 2017, Maldonado et al., 2018, Debbi et al., 2018). La patogenicidad entre las poblaciones naturales de F. oxysporum es un proceso complejo. Por lo tanto, la presencia de genes efectores por sí sola puede no ser un predictor exacto de patogenicidad entre los aislados de suelo de F. oxysporum (Jelinski et al., 2017).
.
- Decoloración del sistema vascular observado en corte de tallo de tomate, indicando infección. Este es un síntoma asociado con la enfermedad, pero no es confirmatorio ya que otras enfermedades fúngicas y bacterianas pueden causar síntomas similares. Autor: Paret, Florida University
- Síntomas de pardeamiento del sistema vascular y la médula en corte transversal de plántula de tomate con un tallo cortado. Esto se asemeja a los síntomas de la marchitez bacteriana, pero una prueba de exudado bacteriano será negativa para la marchitez por Fusarium. Autor: Paret, University of Flordia
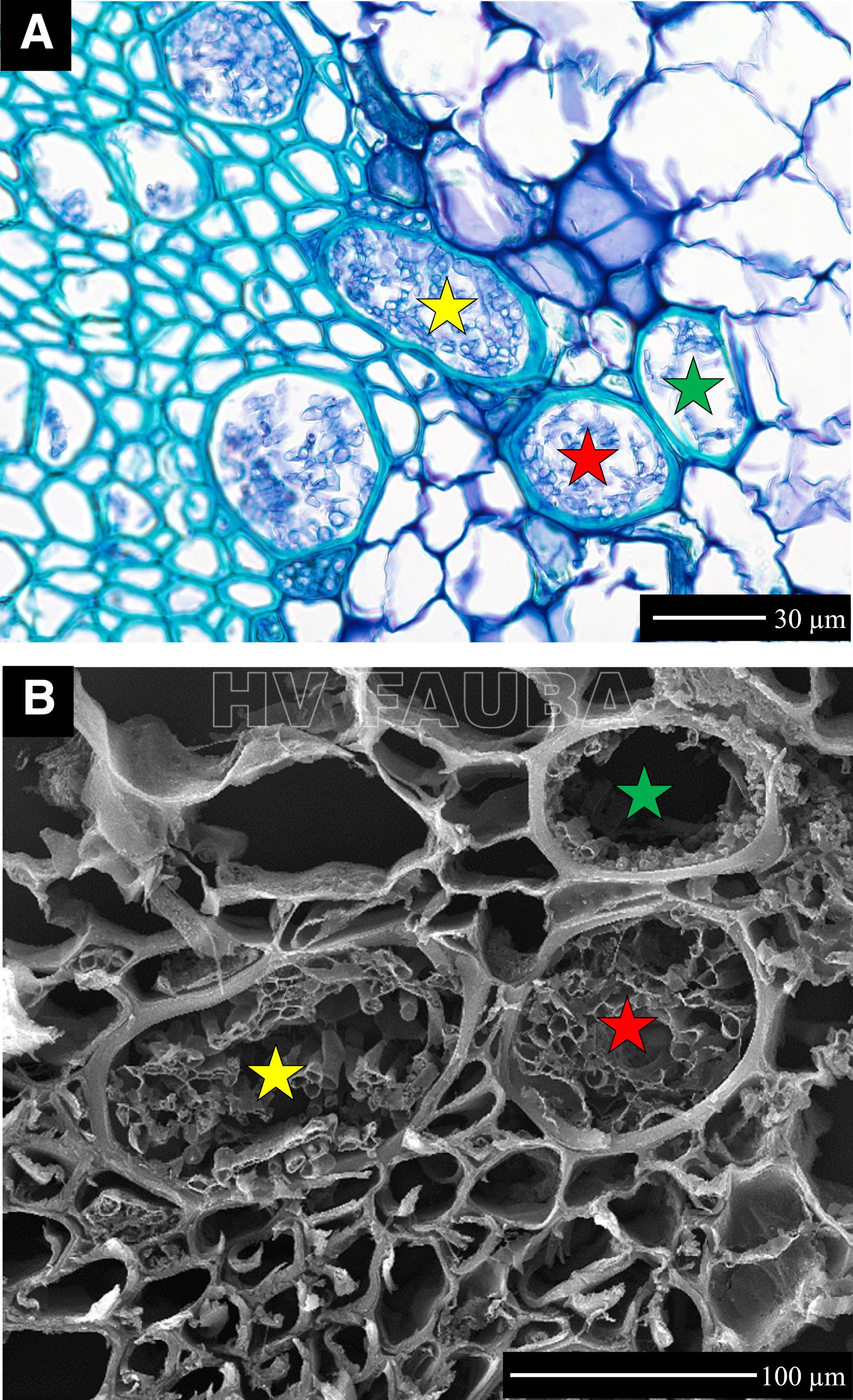
- Fusarium oxysporum f. sp. lycopersici en tallo de una variedad de tomate susceptible que muestra hifas en los vasos del xilema. A, microscopía óptica en una sección transversal de 60× de tallo de tomate teñido con azul de toluidina. B, Misma región que en A pero con microscopía electrónica de barrido (SEM) a 2182 aumentos. Las estrellas indican los vasos del xilema correspondientes en A y B. Barra de escala = 30 µm en A y 100 µm en B. Autor: Caldwell y Iyer-Pascuzzi, 2019.
.
Epidemiología
El Marchitamiento o fusariosis del tomate es 0una enfermedad monocíclica. El hongo sobrevive como clamidosporas libres en el suelo. Estas requieren estimulación proporcionada por los exudados de la raíz, por lo que solo las esporas que se encuentran muy cerca de la raíz tienen la oportunidad de establecer una infección. Esto corresponde a las clamidosporas que se ubican dentro de la rizosfera, que generalmente se extiende a 1 mm o menos desde la superficie de la raíz (Huisman, 1982; Rovira, 1969).
.
- Ciclo del marchitamiento o fusariosis del tomate, causado por (Fusarium oxysporum f. sp. lycopersici). Autor: Jangir et al., 2021.
.
Manejo Integrado
* Plantar cultivares de tomate resistentes (cuando hubiera disponibles). Se conocen fuentes de resistencia a las tres razas (ver tabla 1 en McGovern, 2015). Las razas 1 y 2 crecen en las regiones productoras de tomate de todo el mundo, mientras que la raza 3 se ha reportado en regiones o países como California, Australia, el suroeste de Georgia y México. La mayoría de las variedades comerciales de tomate cultivadas en todo el mundo son resistentes a las razas 1 y 2, y algunas son resistentes a la raza 3 (Biju et al., 2017).
* Injerto (existen materiales con resistencia a las 3 razas, comercialmente disponibles)
* Solarización
* Desinfección Suelos con fumigantes químicos (ej. 1-3 dicloropropeno)
* Cultivo en Sustrato
* Control biológico (ej. Trichoderma spp. (Debbi et al., 2018), Bacillus spp. (Shahzad et al., 2017), bacterias PGPR (Syed Nabi et al., 2021))
* Inducción de defensas. Se han reportado respuestas de control con aplicaciones de acibenzolar-S-methyl, benzotiadiazol, quitosano, compost, ácido jasmónico, ácido salicílico, validamicina A, silicio al transplante, etc. (ver tabla 2 en McGovern, 2015).
* Tratamientos químicos, en drench al transplante o aplicados en riego por goteo (azoxistrobina, trifloxistrobina, cyprodinil+fludioxonil, pydiflumetofen, difenoconazole, tebuconazole, boscalid+pyraclostrobin)
.
.
.
Bibliografía
Ajilogba CF, Babalola OO (2013) Integrated management strategies for tomato Fusarium wilt. Biocontrol Sci. 18(3): 117-127. doi: 10.4265/bio.18.117
Altimira F, Godoy S, Arias-Aravena M, et al. (2022) Genomic and Experimental Analysis of the Biostimulant and Antagonistic Properties of Phytopathogens of Bacillus safensis and Bacillus siamensis. Microorganisms 10(4): 670. doi: 10.3390/microorganisms10040670
Andolfo G, Ferriello F, Tardella L, et al. (2014) Tomato genome-wide transcriptional responses to Fusarium wilt and tomato Mosaic virus. PLoS One 9(5): e94963. doi: 10.1371/journal.pone.0094963
Baayen RP, O’Donnell K, Bonants PJ, et al. (2000) Gene Genealogies and AFLP Analyses in the Fusarium oxysporum Complex Identify Monophyletic and Nonmonophyletic Formae Speciales Causing Wilt and Rot Disease. Phytopathology 90(8): 891-900. doi: 10.1094/PHYTO.2000.90.8.891
Barocio-Ceja NB, et al. (2013) In vitro biocontrol of tomato pathogens using antagonists isolated from chicken-manure vermicompost. FYTON 82: 15-22. Link
Bartholomew ES, Xu S, Zhang Y, et al. (2022) A chitinase CsChi23 promoter polymorphism underlies cucumber resistance against Fusarium oxysporum f. sp. cucumerinum. New Phytol. doi: 10.1111/nph.18463
Bertoldo C, Gilardi G, Spadaro D, et al. (2015) Genetic diversity and virulence of Italian strains of Fusarium oxysporum isolated from Eustoma grandiflorum. Eur J Plant Pathol 141: 83–97. doi: 10.1007/s10658-014-0526-2
, , , et al (2023) The PTI-suppressing Avr2 effector from Fusarium oxysporum suppresses mono-ubiquitination and plasma membrane dissociation of BIK1. Molecular Plant Pathology 24: 1273–1286. doi: 10.1111/mpp.13369
Biju VC, Fokkens L, Houterman PM, et al. (2017) Multiple Evolutionary Trajectories Have Led to the Emergence of Races in Fusarium oxysporum f. sp. lycopersici. Appl Environ Microbiol. 83(4): e02548-16. doi: 10.1128/AEM.02548-16
Boix-Ruíz A, Gálvez-Patón L, de Cara-García M, et al. (2015) Comparison of analytical techniques used to identify tomato-pathogenic strains of Fusarium oxysporum . Phytoparasitica 43: 471–483.
Bost SC (2001) First Report of Fusarium oxysporum f. sp. lycopersici Race 3 on Tomato in Tennessee. Plant Disease 85(7): 802. doi: 10.1094/PDIS.2001.85.7.802D
Caldwell D, Iyer-Pascuzzi AS (2019) A Scanning Electron Microscopy Technique for Viewing Plant-Microbe Interactions at Tissue and Cell-Type Resolution. Phytopathology 109(7): 1302-1311. doi: 10.1094/PHYTO-07-18-0216-R
, , , et al (2023) Synergistic effects of Bacillus velezensis SDTB038 and phenamacril on Fusarium crown and root rot of tomato. Plant Pathology 00: 1– 10. doi: 10.1111/ppa.13769
Choi HW, Hong SK, Lee YK, Shim HS (2013) First Report of Fusarium oxysporum f. sp. lycopersici Race 3 Causing Fusarium Wilt on Tomato in Korea. Plant Disease 97(10): 1377. doi: 10.1094/PDIS-01-13-0073-PDN
Civieta-Bermejo B, et al. (2021) Residuos de repollo para biocontrol de Fusarium spp. en el cultivo de tomate. Revista Mexicana de Ciencias Agrícolas 26: 95-04. doi: 10.29312/remexca.v0i26.2940
Constantin ME, Fokkens L, de Sain M, et al. (2021) Number of Candidate Effector Genes in Accessory Genomes Differentiates Pathogenic From Endophytic Fusarium oxysporum Strains. Front. Plant Sci. 12: 761740. doi: 10.3389/fpls.2021.761740
Debbi A, Boureghda H, Monte E, Hermosa R (2018) Distribution and Genetic Variability of Fusarium oxysporum Associated with Tomato Diseases in Algeria and a Biocontrol Strategy with Indigenous Trichoderma spp. Front. Microbiol. 9: 282. doi: 10.3389/fmicb.2018.00282
Dror B, Amutuhaire H, Frenkel O, et al. (2022) Identification of Bacterial Populations and Functional Mechanisms Potentially Involved in Biochar-Facilitated Antagonism of the Soilborne Pathogen Fusarium oxysporum. Phytobiomes Journal 6: 139-150. doi: 10.1094/PBIOMES-11-21-0071-R
Fuchs JG, Moënne-Loccoz Y, Défago G (1997) Nonpathogenic Fusarium oxysporum strain Fo47 induces resistance to Fusarium wilt in tomato. Plant Disease 81: 492-496. doi: 10.1094/PDIS.1997.81.5.492
Gordon TR (2017) Fusarium oxysporum and the Fusarium Wilt Syndrome. Annu Rev Phytopathol. 55: 23-39. doi: 10.1146/annurev-phyto-080615-095919
Hernández-Aparicio F, Lisón P, Rodrigo I, et al. (2021) Signaling in the Tomato Immunity against Fusarium oxysporum. Molecules 26(7): 1818. doi: 10.3390/molecules26071818
Hirano Y, Arie T (2006) PCR-based differentiation of Fusarium oxysporum f. sp. lycopersici and radicis-lycopersici and races of F. oxysporum f. sp. lycopersici . J Gen Plant Pathology 72: 273–283. doi: 10.1007/s10327-006-0287-7
Huisman OC (1982) Interrelations of root growth dynamics to epidemiology of root-invading fungi. Annu. Rev. Phytopathol. 20: 303–27.
Inami K, Kashiwa T, Kawabe M, et al. (2014) The tomato wilt fungus Fusarium oxysporum f. sp. lycopersici shares common ancestors with nonpathogenic F. oxysporum isolated from wild tomatoes in the Peruvian Andes. Microbes Environ. 29(2): 200-210. doi: 10.1264/jsme2.ME13184
Jangir P, Mehra N, Sharma K, et al. (2021) Secreted in Xylem Genes: Drivers of Host Adaptation in Fusarium oxysporum. Front. Plant Sci. 12: 628611. doi: 10.3389/fpls.2021.628611
Jelinski NA, Broz K, Jonkers W, et al. (2017) Effector Gene Suites in Some Soil Isolates of Fusarium oxysporum Are Not Sufficient Predictors of Vascular Wilt in Tomato. Phytopathology 107(7): 842-851. doi: 10.1094/PHYTO-12-16-0437-R
Ji HM, Mao HY, Li SJ, et al. (2021) Fol-milR1, a pathogenicity factor of Fusarium oxysporum, confers tomato wilt disease resistance by impairing host immune responses. New Phytol. 232(2): 705-718. doi: 10.1111/nph.17436
Khan M, Khan AU, Bogdanchikova N, Garibo D (2021) Antibacterial and Antifungal Studies of Biosynthesized Silver Nanoparticles against Plant Parasitic Nematode Meloidogyne incognita, Plant Pathogens Ralstonia solanacearum and Fusarium oxysporum. Molecules 26(9): 2462. doi: 10.3390/molecules26092462
Kannan V, Sureendar R (2009) Synergistic effect of beneficial rhizosphere microflora in biocontrol and plant growth promotion. J. Basic Microbiol. 49: 158–164. doi: 10.1002/jobm.200800011
Krishnan SV, Anaswara PA, Nampoothiri KM, et al. (2025) Biocontrol Activity of New Lactic Acid Bacteria Isolates Against Fusaria and Fusarium Mycotoxins. Toxins 17(2): 68. doi: 10.3390/toxins17020068
, , , (2021) Target of rapamycin controls hyphal growth and pathogenicity through FoTIP4 in Fusarium oxysporum. Molecular Plant Pathology 00, 1– 17. doi: 10.1111/mpp.13108
, , A single gene in Fusarium oxysporum limits host range. Molecular Plant Pathology 22: 108– 116. doi: 10.1111/mpp.13011
, , , Related mobile pathogenicity chromosomes in Fusarium oxysporum determine host range on cucurbits. Molecular Plant Pathology 21: 761– 776. doi: 10.1111/mpp.12927
Li J, Ma X, Wang C, et al. (2022) Acetylation of a fungal effector that translocates host PR1 facilitates virulence. Elife. 11: e82628. doi: 10.7554/eLife.82628
Lombard L, Sandoval-Denis M, Lamprecht SC, Crous PW (2019) Epitypification of Fusarium oxysporum – clearing the taxonomic chaos. Persoonia 43: 1-47. doi: 10.3767/persoonia.2019.43.01
Lopez-Lima D, Mtz-Enriquez AI, Carrión G, et al. (2021) The bifunctional role of copper nanoparticles in tomato: Effective treatment for Fusarium wilt and plant growth promoter. Scientia Horticulturae 277: 109810. doi: 10.1016/j.scienta.2020.109810
Malbrán I, Mourelos CA, Lori GA (2020) First Report of Fusarium oxysporum f. sp. lycopersici Race 3 Causing Fusarium Wilt of Tomato in Argentina. Plant Disease 104: 978-978. doi: 10.1094/PDIS-08-19-1777-PDN
Maldonado BLD, Villarruel OJL, Calderón OMA, Sánchez EAC (2018) Secreted in xylem (Six) genes in Fusarium oxysporum f.sp. cubense and their potential acquisition by horizontal transfer. Adv. Biotech. Micro. 10 (1): 555779. Link
Marlatt ML, Correll JC, Kaufmann P, Cooper PE (1996) Two genetically distinct populations of Fusarium oxysporum f. sp. lycopersici race 3 in the United States. Plant Disease 80: 1336-1342. doi: 10.1094/PD-80-1336
Martínez-Soto D, Yu H, Allen KS, Ma LJ (2023) Differential Colonization of the Plant Vasculature Between Endophytic Versus Pathogenic Fusarium oxysporum Strains. Molecular Plant-Microbe Interactions. doi: 10.1094/MPMI-08-22-0166-SC
Mazungunye HT, Ngadze E (2021) Evaluation of Trichoderma Strains as Biocontrol of Fusarium oxysporum f. sp. lycopersici in Tomato. J Plant Pathol Microbiol 12: 571. Link
McGovern RJ (2015) Management of tomato diseases caused by Fusarium oxysporum. Crop Protection 73: 78-92. doi: 10.1016/j.cropro.2015.02.021
Mitidieri MS, et al. (2015) Evaluación de parámetros de rendimiento y sanidad de dos híbridos comerciales de tomate (Solanum lycopersicum L.) injertados sobre Solanum sisymbriifolium (Lam.), en un invernadero con suelo biosolarizado. Horticultura Argentina 34(84): 5-17. Link
Modrzewska M, Bryła M, Kanabus J, Pierzgalski A (2022) Trichoderma as a biostimulator and biocontrol agent against Fusarium in the production of cereal crops: opportunities and possibilities. Plant Pathol. doi: 10.1111/ppa.13578
, (2023) Constitutive activation of TORC1 signalling attenuates virulence in the cross-kingdom fungal pathogen Fusarium oxysporum. Molecular Plant Pathology 24: 289– 301. doi: 10.1111/mpp.13292
Nirmaladevi D, Venkataramana M, Srivastava RK, et al. (2016) Molecular phylogeny, pathogenicity and toxigenicity of Fusarium oxysporum f. sp. lycopersici. Sci Rep. 6: 21367. doi: 10.1038/srep21367
Obregón V (2018) Guía para la identificación de las enfermedades de tomate en invernadero. INTA EEA Bella Vista. Link
O’Donnell K, Kistler HC, Cignelnik E, Ploetz RC (1998) Multiple evolutionary origins of the fungus causing Panama disease of banana: concordant evidence from nuclear and mitochondrial gene genealogies. PNAS 95(5): 2044–49. doi: 10.1073/pnas.95.5.2044
O’Donnell K, Whitaker BK, Laraba I, et al. (2022) DNA Sequence-Based Identification of Fusarium: A Work in Progress. Plant Disease 106(6): 1597-1609. doi: 10.1094/PDIS-09-21-2035-SR
Panthee DR, Chen F (2010) Genomics of fungal disease resistance in tomato. Curr. Genomics 11 : 30-39. doi: 10.2174/138920210790217927
Ploetz RC, Haynes JL (2000) First Report of Race 3 of Fusarium oxysporum f. sp. lycopersici in Southeastern Florida. Plant Disease 84(2): 199. doi: 10.1094/PDIS.2000.84.2.199B
Redkar A, Sabale M, Schudoma C, et al. (2022) Conserved secreted effectors contribute to endophytic growth and multi-host plant compatibility in a vascular wilt fungus. Plant Cell.: koac174. doi: 10.1093/plcell/koac174
Reis A, et al. (2005) First report of Fusarium oxysporum f. sp. lycopersici race 3 on tomato in Brazil. Fitopatologia Brasileira 30: 426-428. doi: 10.1590/S0100-41582005000400017
Rep M, Van Der Does HC, Meijer M, et al. (2004) A small, cysteine-rich protein secreted by Fusarium oxysporum during colonization of xylem vessels is required for I-3-mediated resistance in tomato. Molecular Microbiology 53: 1373-1383. doi: 10.1111/j.1365-2958.2004.04177.x
Rovira AD (1969) Plant root exudates. Bot. Rev. 35: 35–57.
Samaras A, Roumeliotis E, Ntasiou P, Karaoglanidis G (2021) Bacillus subtilis MBI600 Promotes Growth of Tomato Plants and Induces Systemic Resistance Contributing to the Control of Soilborne Pathogens. Plants 10(6): 1113. doi: 10.3390/plants10061113
Sasaki K, Nakahara K, Tanaka S, et al. (2015) Genetic and Pathogenic Variability of Fusarium oxysporum f. sp. cepae Isolated from Onion and Welsh Onion in Japan. Phytopathology 105(4): 525-32. doi: 10.1094/PHYTO-06-14-0164-R
Schmidt SM, Houterman PM, Schreiver I, et al. (2013) MITEs in the promoters of effector genes allow prediction of novel virulence genes in Fusarium oxysporum. BMC Genomics 14: 119. doi: 10.1186/1471-2164-14-119
Schmidt SM, Lukasiewicz J, Farrer R, et al. (2016) Comparative genomics of Fusarium oxysporum f. sp. melonis reveals the secreted protein recognized by the Fom-2 resistance gene in melon. New Phytol 209: 307-318. doi: 10.1111/nph.13584
Scott JW, Agrama HA, Jones JP (2004) RFLP-based analysis of recombination among resistance genes to Fusarium wilt Races 1, 2, and 3 in tomato. J. Am. Soc. Hortic. Sci., 129 (3): 394-400. doi: 10.21273/JASHS.129.3.0394
Sepúlveda-Chavera G, Huanca W, Salvatierra-Martínez R, Latorre BA (2014) First Report of Fusarium oxysporum f. sp. lycopersici Race 3 and F. oxysporum f. sp. radicis-lycopersici in Tomatoes in the Azapa Valley of Chile. Plant Disease 98(10): 1432. doi: 10.1094/PDIS-03-14-0303-PDN
Shahzad R, Khan AL, Bilal S, et al. (2017) Plant growth- promoting endophytic bacteria versus pathogenic infections: an example of Bacillus amyloliquefaciens RWL-1 and Fusarium oxysporum f. sp. lycopersici in tomato. Peer J. 5: e3107. doi: 10.7717/peerj.3107
Shanmugam V, Kanoujia N (2011) Biological management of vascular wilt of tomato caused by Fusarium oxysporum f. sp. lycopersici by plant growth-promoting rhizobacterial mixture. Biol. Control 57: 85–93. doi: 10.1016/j.biocontrol.2011.02.001
Snyder WC, Hansen HN (1940) The species concept in Fusarium. Am. J. Bot. 27(2): 64–67.
Srinivas C, Nirmala Devi D, Narasimha Murthy K, et al. (2019) Fusarium oxysporum f. sp. lycopersici causal agent of vascular wilt disease of tomato: Biology to diversity– A review. Saudi Journal of Biological Sciences 26: 1315-1324. doi: 10.1016/j.sjbs.2019.06.002
Srivastava R, Khalid A, Singh US, Sharma AK (2010) Evaluation of arbuscular mycorrhizal fungus, fluorescent Pseudomonas and Trichoderma harzianum formulation against Fusarium oxysporum f. sp. lycopersici for the management of tomato wilt. Biol. Control 53, 24–31. doi: 10.1016/j.biocontrol.2009.11.012
Syed Nabi RB, Shahzad R, Tayade R, et al. (2021) Evaluation potential of PGPR to protect tomato against Fusarium wilt and promote plant growth. PeerJ. 9: e11194. doi: 10.7717/peerj.11194
Takken F, Rep M (2010) The arms race between tomato and Fusarium oxysporum. Molecular Plant Pathology 11: 309-314. doi: 10.1111/j.1364-3703.2009.00605.x
, , , et al. (2023) The neighbouring genes AvrLm10A and AvrLm10B are part of a large multigene family of cooperating effector genes conserved in Dothideomycetes and Sordariomycetes. Molecular Plant Pathology 00: 1– 18. doi: 10.1111/mpp.13338
Teulet A, Quan C, Evangelisti E, et al. (2023) A pathogen effector FOLD diversified in symbiotic fungi. New Phytol. doi: 10.1111/nph.18996
Vicente I, Quaratiello G, Baroncelli R, et al. (2022) Insights on KP4 Killer Toxin-like Proteins of Fusarium Species in Interspecific Interactions. Journal of Fungi. 8(9): 968. doi: 10.3390/jof8090968
Wang L, Calabria J, Chen HW, Somssich M (2022) Overview: The Arabidopsis thaliana – Fusarium oxysporum strain 5176 pathosystem. Journal of Experimental Botany: erac263. doi: 10.1093/jxb/erac263
Zhou X, Wang J, Liu F, et al. (2022) Cross-kingdom synthetic microbiota supports tomato suppression of Fusarium wilt disease. Nat Commun 13: 7890. doi: 10.1038/s41467-022-35452-6
Zhou Y, Zhang Z, Bao Z, et al. (2022) Graph pangenome captures missing heritability and empowers tomato breeding. Nature. doi: 10.1038/s41586-022-04808-9