.
Condición fitosanitaria: Presente solo en algunas áreas del país
Grupo de cultivos: Cereales
Especie hospedante: Maíz (Zea mays)
Rango de hospedantes:
Etiología: Virus. (Biotrófico como todos los virus)
Agente causal: la enfermedad es el resultado de una combinación de dos virus:
* el virus del moteado clorótico del maíz (Maize Chlorotic Mottle Virus, MCMoV),
* y cualquiera de los virus de los cereales del grupo Potyviridae, como el virus del mosaico de la caña de azúcar (Sugarcane Mosaic Virus, SCMV), el virus del mosaico estriado del trigo (Wheat Streak Mosaic Virus, WSMV) o el virus del mosaico enano del maíz (Maize Dwarf Mosaic Virus, MDMV).
.
Taxonomía:
Viruses > Riboviria > Orthornavirae > Kitrinoviricota > Tolucaviricetes > Tolivirales > Tombusviridae > Procedovirinae > Machlomovirus > MCMoV
Viruses > Riboviria > Orthornavirae > Pisuviricota > Stelpaviricetes > Patatavirales > Potyviridae > Potyvirus > SCMV
Viruses > Riboviria > Orthornavirae > Pisuviricota > Stelpaviricetes > Patatavirales > Potyviridae > Tritimovirus > WSMV
Viruses > Riboviria > Orthornavirae > Pisuviricota > Stelpaviricetes > Patatavirales > Potyviridae > Potyvirus > MDMV
.
Baltimore classification: Group IV: ssRNA(+) Baltimore, 1971
.
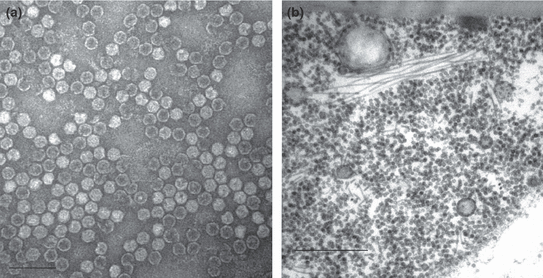
- (A) Partículas de virus isométricas purificadas del Maize Chlorotic Mottle Virus (MCMoV). La barra de escala representa 100 nm; (B) Partículas de virus isométricas de Maize Chlorotic Mottle Virus (MCMoV) observadas en el citoplasma. La barra de escala representa 500 nm. Autor: Xie et al., 2010
.
.
El MCMV tiene un genoma compacto de ARN de sentido positivo (+ ARN) de 4,4 kb que no está protegido ni poliadenilado (Nutter et al., 1989). Está encapsulado en viriones icosaédricos t = 3 de aproximadamente 30 nm de diámetro, compuestos de una sola proteína de la cápside (Paul et al., 1980). La partícula del virus es muy estable y puede retener la infectividad a 20 °C durante más de 30 días, con inactivación térmica a 80–85 ° C (Uyemoto et al., 1981). El MCMV es fácilmente transmisible mecánicamente al maíz y otros hospedantes experimentales en entornos de laboratorio. El genoma del MCMV contiene cuatro marcos de lectura abiertos (ORF) principales y un ORF pequeño, que codifica siete proteínas mediante diversas estrategias de expresión. A partir del +ARN genómico, el genoma de MCMV codifica tres proteínas: ORF1 comienza en el primer codón de inicio y codifica p32, una proteína con un modo de acción desconocido que aumenta la severidad de los síntomas y mejora el establecimiento y la acumulación del virus (Scheets, 2016). El ORF2 se superpone parcialmente al ORF1 y codifica las proteínas necesarias para la replicación, p50 y p111, que se expresa mediante la lectura de un codón de terminación UAG al final de p50 (Nutter et al., 1989; Scheets, 2016). El ORF 3′ de MCMV se expresa a partir del ARN1 subgenómico (sgRNA1) (Scheets, 2000), que expresa p7a del extremo 5′ del ORF3 y p31 mediante la lectura de un codón de terminación UAG. Se predice que otra pequeña proteína, p7b, está codificada por un pequeño ORF después del ORF3 con un codón de inicio CUG inusual (Scheets, 2016). Las proteínas p7a y p7b son proteínas de movimiento predichas (MP) basadas en la similitud de secuencia con las MP conocidas de Tombusviridae. El ORF4 codifica la CP de 25 kDa del segundo codón de inicio sgRNA1 (Scheets, 2000, Scheets, 2016).
.
.
.
Antecedentes
El MCMV se reportó en Argentina en 1981 (Teyssandier et al., 1982).
Maize lethal necrosis disease (MLN) se reportó por primera vez en Kenia en 2011 en el Valle del Rift y posteriormente se ha extendido a diferentes agroecologías de maíz donde está causando pérdidas considerables (Wangai et al., 2012). El SCMV se informó hace varios años en Kenia (Louie 1980) y Sudáfrica (Handley et al., 1998), pero el MCMV es más reciente en África. El MCMV se identificó por primera vez en Perú en 1973 (Castillo y Hebert, 1974) y posteriormente se informó en los EE. UU., Partes de América Latina y China (Niblett y Claflin, 1978; Uyemoto, 1983; Xie et al., 2011). También se ha reportado el MLN en Ruanda (Adams et al., 2014) y la República Democrática del Congo (Lukanda et al., 2014). Se han informado síntomas similares en el maíz en Uganda y Tanzania (Wangai et al., 2012). En Kenia, se registró un brote epidémico grave de la enfermedad, diagnosticado como necrosis letal del maíz (MLN) en septiembre de 2011 en la división de Longisa del distrito de Bomet (Wangai et al., 2012).
.
Síntomatología
- Plantas de maíz infectadas en el campo que muestran síntomas foliares cloróticos y necróticos. Autor: Xie et al., 2010
.
.
Diagnóstico
Varias herramientas aplicadas al monitoreo del MLND incluyen el ensayo inmunoabsorbente ligado a enzimas en sándwich de doble antígeno (DAS-ELISA) y la reacción en cadena de la polimerasa en tiempo real (qPCR) (Fatma et al., 2016), pero estos podrían tener un éxito limitado debido a la divergencia viral entre regiones (Braidwood et al., 2018) así como baja sensibilidad en semillas de maíz debido a bajos títulos virales (Quito-Avila et al., 2016). El uso de tecnologías de secuenciación de próxima generación (NGS) en la detección y caracterización de virus causante de MLND ha llevado a una mejor comprensión de estos virus (Braidwood et al., 2018; Xia et al., 2014, 2016, 2018)
.
.
Manejo Integrado
* Siembra de híbridos con germoplasma tolerante
El mejoramiento para la tolerancia a través del desarrollo de líneas endogámicas es una estrategia en curso del Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT) y otros equipos de investigación independientes (Beyene et al., 2017). Esta estrategia está respaldada por estudios que involucran análisis genéticos de loci de rasgos cuantitativos (QTLs) para la resistencia, así como estudios de asociación de todo el genoma destinados a identificar germoplasma tolerante para la reproducción contra MLND (Awata et al., 2019; Gowda et al., 2015; Gowda et al., 2018; Nyaga et al., 2019).
* Uso de semilla sana
* Rotación de cultivos
* Eliminación de malezas y plagas
.
.
.
.
Bibliografia
Adams IP, Harju VA, Hodges T, et al. (2014) First report of maize lethal necrosis disease in Rwanda. New Disease Reports 29: 22. doi: 10.5197/j.2044-0588.2014.029.022
Awata LAO, Beyene Y, Gowda M, et al. (2020) Genetic Analysis of QTL for Resistance to Maize Lethal Necrosis in Multiple Mapping Populations. Genes 11(1): 32. doi: 10.3390/genes11010032
Beyene Y, Gowda M, Suresh LM, et al. (2017) Genetic analysis of tropical maize inbred lines for resistance to maize lethal necrosis disease. Euphytica 213: 224. doi: 10.1007/s10681-017-2012-3
Braidwood L, Quito-Avila DF, Cabanas D, et al. (2018) Maize chlorotic mottle virus exhibits low divergence between differentiated regional sub-populations. Sci Rep 8: 1173. doi: 10.1038/s41598-018-19607-4
Cabanas D, Watanabe S, Higashi CHV, Bressan A (2013) Dissecting the mode of maize chlorotic mottle virus transmission (Tombusviridae: Machlomovirus) by Frankliniella williamsi (Thysanoptera: Thripidae). J Econ Entomol 106: 16–24. doi: 10.1603/EC12056
Castillo J, Hebert TT (1974) Nueva enfermedad virosa afectando al maiz en el Peru. Fitopatologia 9: 79–84. Link
Castillo-Loayza J (1977) Maize virus and virus-like diseases in Peru. In: LE Williams, DT Gordon, LR Nault. (eds) Proc Int Maize Virus Disease Colloquim Workshop, 16-19 August 1976. Wooster, OH, pp 40– 44.
Fatma HK, Tileye F, Patrick AN (2016) Insights of maize lethal necrotic disease: A major constraint to maize production in East Africa. African Journal of Microbiology Research 10: 271–279. doi: 10.5897/AJMR2015.7534
Goldberg KA, Brakke MK (1987) Concentration of maize chlorotic mottle virus in mixed infections with maize dwarf mosaic virus, strain B. Phytopathology 77: 162– 177. doi: 10.1094/Phyto-77-162
Gowda M, Das B, Makumbi D, et al. (2015) Genome-wide association and genomic prediction of resistance to maize lethal necrosis disease in tropical maize germplasm. Theor Appl Genet. 128(10): 1957-68. doi: 10.1007/s00122-015-2559-0
Gowda M, Beyene Y, Makumbi D, et al. (2018) Discovery and validation of genomic regions associated with resistance to maize lethal necrosis in four biparental populations. Mol Breed. 38(5): 66. doi: 10.1007/s11032-018-0829-7
Handley JA, Smith GR, Dale JL, Harding RM (1998) Sequence diversity in the coat protein coding region of twelve sugarcane mosaic potyvirus isolates from Australia, USA and South Africa. Arch Virol 143: 1145–1153. doi: 10.1007/s007050050362
He Z, Guo J-F, Reitz SR, et al. (2020) A global invasion by the thrip, Frankliniella occidentalis: Current virus vector status and its management. Insect Science 27: 626-645. doi: 10.1111/1744-7917.12721
Jensen SG (1991) Seed transmission of maize chlorotic mottle virus. Plant Disease 75: 497-498. doi: 10.1094/PD-75-0497
Jiang XQ, Wilkinson DR, Berry JA. (1990) An outbreak of maize chlorotic mottle virus in Hawaii and possible association with thrips. Phytopathology 80: 1060.
Jiang XQ, Meinke LJ, Wright RJ, et al. (1992) Maize chlorotic mottle virus in Hawaiian-grown maize: vector relations, host range and associated viruses. Crop Protection 11: 248-254. doi: 10.1016/0261-2194(92)90045-7
Lommel SA, Kendall TL, Siu NF, Nutter RC (1991) Characterization of maize chlorotic mottle virus. Phytopathology 81: 819– 823. doi: 10.1094/Phyto-81-819
Louie R (1980) Sugarcane mosaic virus in Kenya. Plant Disease 64: 944.
Lukanda M, Owati A, Ogunsanya P, et al. (2014) First report of Maize chlorotic mottle virus infecting maize in the democratic Republic of the Congo. Plant Disease 98: 1448. doi: 10.1094/PDIS-05-14-0484-PDN
Mahuku G, Lockhart BE, Wanjala B, et al. (2015) Maize Lethal Necrosis (MLN), an Emerging Threat to Maize-Based Food Security in Sub-Saharan Africa. Phytopathology 105(7): 956-65. doi: 10.1094/PHYTO-12-14-0367-FI
Mengesha GG, Mohammed BK, Salo KS ((2019) Management of maize lethal necrosis disease through hand weeding and insecticide applications at Arba Minch in Southern Ethiopia, Cogent Food & Agriculture 5: 1. doi: 10.1080/23311932.2019.1705746
Nault LR (1978) Transmission of maize chlorotic mottle virus by chrysomelid beetles. Phytopathology 68: 1071.
Niblett CL, Claflin LE (1978) Corn lethal necrosis – a new virus disease of corn in Kansas. Plant Dis Rep 62: 15–19. Link
Nutter RC, Scheets K, Pangnaniban LC, Lommel SA (1989) The complete nucleotide sequence of the maize chlorotic mottle virus genome. Nucleic Acids Res 17: 3163– 3177. doi: 10.1093/nar/17.8.3163
Nyaga C, Gowda M, Beyene Y, et al. (2020) Genome-Wide Analyses and Prediction of Resistance to MLN in Large Tropical Maize Germplasm. Genes 11(1): 16. doi: 10.3390/genes11010016
Paul HL, Querfurth G, Huth W (1980) Serological studies on the relationships of some isometric viruses of Gramineae. J. Gen. Virol. 47: 67–77. doi:
Quito-Avila DF, Alvarez RA, Mendoza AA (2016) Occurrence of maize lethal necrosis in Ecuador: A disease without boundaries? European Journal of Plant Pathology 146: 705–710. doi: 10.1007/s10658-016-0943-5
Redinbaugh MG, Zambrano JL (2014) Control of Virus Diseases in Maize. Advances in Virus Research 90: 391–429. doi: 10.1016/B978-0-12-801246-8.00008-1
Redinbaugh MG, Stewart LR (2018) Maize Lethal Necrosis: An Emerging, Synergistic Viral Disease. Annu Rev Virol. 5(1): 301-322. doi: 10.1146/annurev-virology-092917-043413
Scheets K. (1998) Maize chlorotic mottle machlomovirus and wheat streak mosaic tymovirus concentrations increase in the synergistic disease maize lethal necrosis. Virology 242: 28– 38. doi: 10.1006/viro.1997.8989
Scheets K (2000) Maize chlorotic mottle machlomovirus expresses its coat protein from a 1.47-kb subgenomic RNA and makes a 0.34-kb subgenomic RNA. Virol. 267: 90–101. doi: 10.1006/viro.1999.0107
Scheets K (2016) Analysis of gene functions in Maize chlorotic mottle virus. Virus Res. 222: 71–79. doi: 10.1016/j.virusres.2016.04.024
Stenger DC, French R (2008) Complete nucleotide sequence of a maize chlorotic mottle virus isolate from Nebraska. Arch Virol 153: 995– 997. doi: 10.1007/s00705-008-0069-y
Teyssandier EE, Nome SF, Dal Bo E (1982) Maize virus diseases in Argentina. In Proceedings of the International Maize Virus Disease Colloquium and Workshop, ed. DT Gordon, JK Knoke, LR Nault, RM Ritter, pp. 93–99. Wooster, OH: Ohio Agric. Res. Dev. Cent.
Tonui R, Masanga J, Kasili R, et al. (2020) Identification of maize lethal necrosis disease causal viruses in maize and suspected alternative hosts through small RNA profiling. J Phytopathol. 168: 439– 450. doi: 10.1111/jph.12908
Uyemoto JK, Bockelman DL, Claflin LE (1980) Severe Outbreak of Corn Lethal Necrosis Disease in Kansas. Plant Disease 64: 99-100. doi: 10.1094/PD-64-99
Uyemoto JK, Claflin LE, Wilson DL, Raney RJ (1981) Maize chlorotic mottle and maize dwarf mosaic viruses; effect of single and double inoculations on symptomatology and yield. Plant Disease 65: 39–41. doi: 10.1094/PD-65-39
Uyemoto JK (1983) Biology and control of maize chlorotic mottle virus. Plant Disease 67: 7. Link
Wagemans J, Holtappels D, Vainio E, et al. (2022) Going Viral: Virus-Based Biological Control Agents for Plant Protection. Annu Rev Phytopathol. 60: 21-42. doi: 10.1146/annurev-phyto-021621-114208
Wang Q, Zhang C, Wang C, et al. (2017) Further characterization of Maize chlorotic mottle virus and its synergistic interaction with Sugarcane mosaic virus in maize. Sci Rep 7: 39960. doi: 10.1038/srep39960
Wangai AW, Redinbaugh MG, Kinyua ZM, et al. (2012) First report of Maize chlorotic mottle virus and maize lethal necrosis in Kenya. Plant Disease 96: 1582. doi: 10.1094/PDIS-06-12-0576-PDN
Xia Z, Peng J, Li Y, et al. (2014) Characterization of Small Interfering RNAs Derived from Sugarcane Mosaic Virus in Infected Maize Plants by Deep Sequencing. PLoS ONE 9(5): 28–33. doi: 10.1371/journal.pone.0097013
Xia Z, Zhao Z, Chen L, et al. (2016) Synergistic infection of two viruses MCMV and SCMV increases the accumulations of both MCMV and MCMV-derived siRNAs in maize. Sci Rep. 6: 20520. doi: 10.1038/srep20520
Xia Z, Zhao Z, Li M, et al. (2018) Identification of miRNAs and their targets in maize in response to Sugarcane mosaic virus infection. Plant Physiol Biochem. 125: 143-152. doi: 10.1016/j.plaphy.2018.01.031
Xie L, Zhang J, Wang Q, et al. (2011) Characterization of Maize Chlorotic Mottle Virus Associated with Maize Lethal Necrosis Disease in China. Journal of Phytopathology, 159: 191-193. doi: 10.1111/j.1439-0434.2010.01745.x
Zhang HM, Chen JP, Xue QZ, et al. (2002) cDNA cloning and sequence analysis of genome segments S8 from rice black-streaked dwarf virus. J Zhejiang Univ–Sci A 3: 216– 222. doi: 10.1007/BF03396441
Zhang Y, Zhao W, Li M, et al (2011) Real-time TaqMan RT-PCR for detection of maize chlorotic mottle virus in maize seeds. J Virol Methods 171: 292–294. doi: 10.1016/j.jviromet.2010.11.002
, , , et al. (2024) A natural substitution of a conserved amino acid in eIF4E confers resistance against multiple potyviruses. Molecular Plant Pathology 25: e13418. doi: 10.1111/mpp.13418