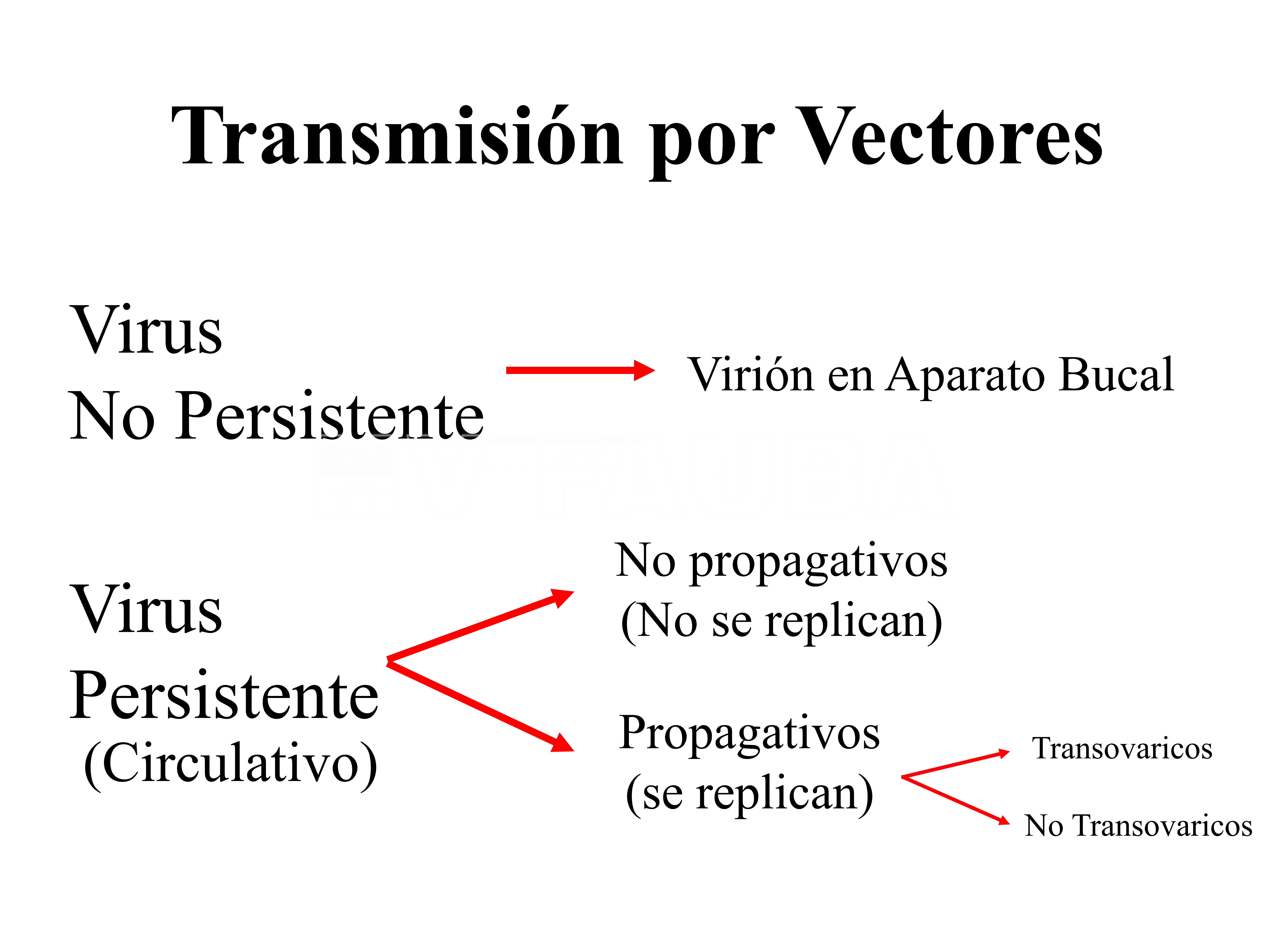
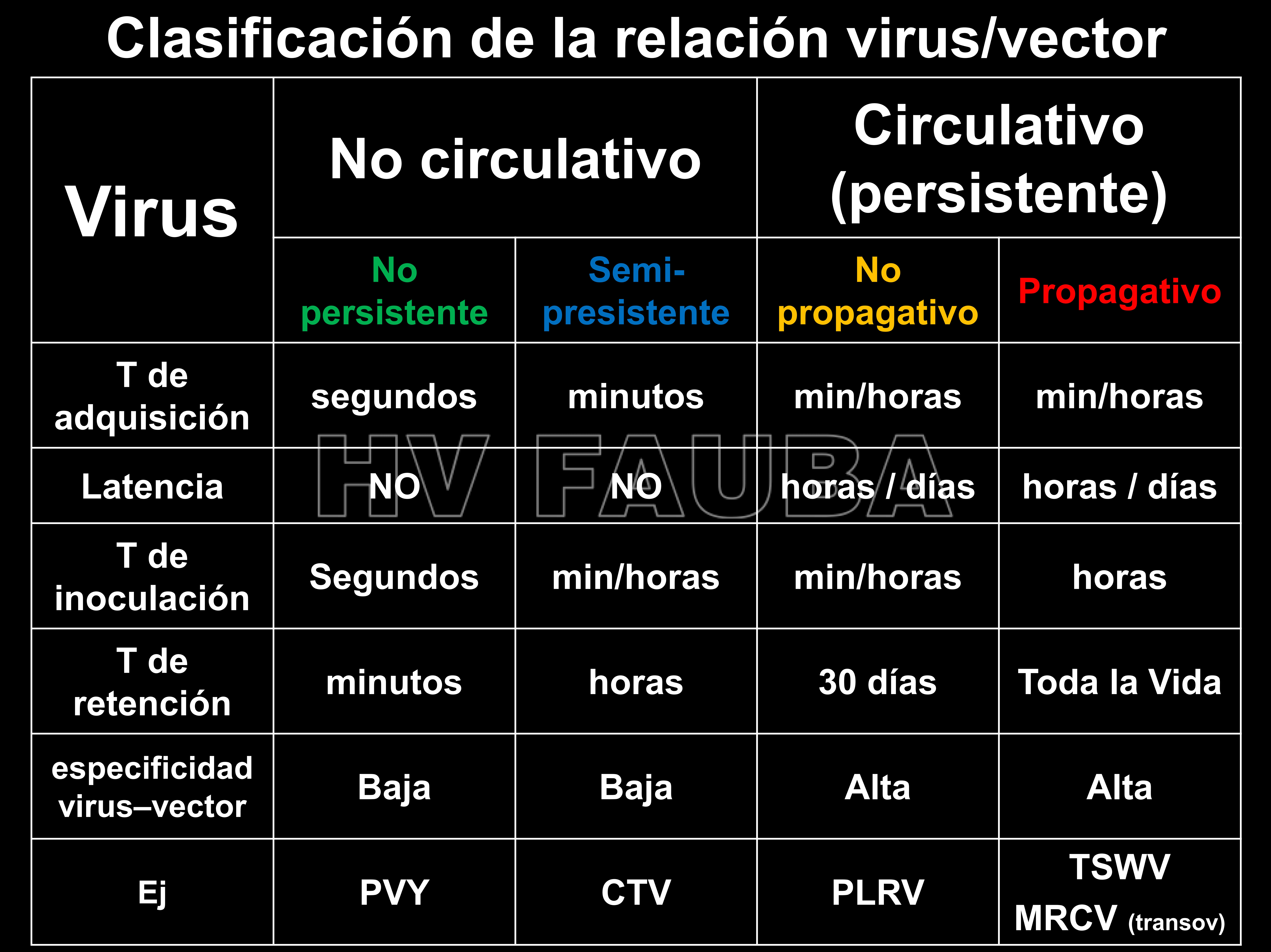
- Clasificación de Baltimore
.
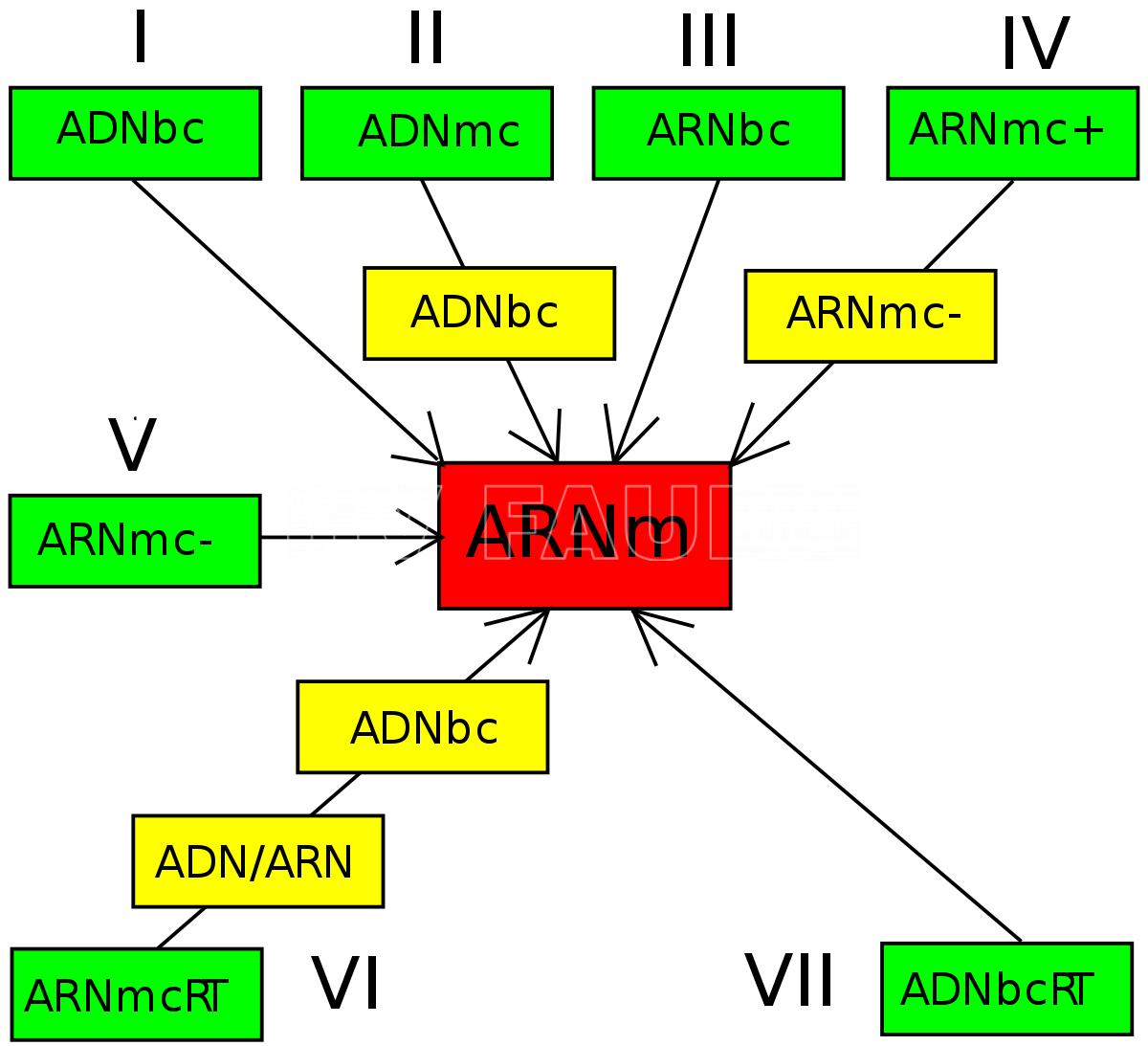
Clasificación de Baltimore: los virus están agrupados en grupos dependiendo de su tipo de genoma (ADN, ARN, monocatenario o bicatenario etc.) y en su método de replicación.
Grupo I: Virus ADN bicatenario (Virus ADNbc o Virus dsDNA): el ARNm se transcribe directamente a partir del genoma del virus, que es una doble cadena de ADN. Las proteínas reguladoras que controlan la replicación del genoma y las proteínas estructurales que forman el virión se traducen a partir de este ARNm.
Grupo II: Virus ADN monocatenario (Virus ADNmc o Virus ssDNA): el ADN viral monocatenario se convierte en bicatenario, probablemente usando la maquinaria de reparación del ADN del hospedero. El resto de las etapas de replicación son similares a las del grupo I.
Grupo III: Virus ARN bicatenario (Virus ARNbc o Virus dsRNA): a partir del ARN bicatenario se obtiene la hebra de ARN monocatenario positivo que actúa como ARNm. La traducción de este ARNm da lugar a las proteínas reguladores y estructurales. La replicación del genoma del virus se realiza en dos pasos. Primero se realiza un ensamblado parcial de la hebra de ARN monocatenario positivo y de las proteínas virales en viriones inmaduros. A continuación, se realiza la replicación del ARN monocatenario positivo a ARN bicatenario dentro de los viriones.
Grupo IV: Virus ARN monocatenario positivo (Virus ARNmc+ o Virus (+)ssRNA): La replicación del virus comienza con la traducción genética de la cadena de ARN monocatenario positivo (que tiene la misma polaridad que el ARNm) en proteínas reguladoras. En el grupo IVa este paso traduce también las proteínas estructurales, mientras que en el grupo IVb esto se realiza traduciendo un ARNm generado a partir de una cadena de ARN monocatenario positivo. Las proteínas regulan la síntesis del ARN monocatenario positivo a partir del molde de ARN monocatenario negativo. Este último a su vez funciona como molde para la síntesis del ARN monocatenario positivo de los nuevos virus.
Grupo V: Virus ARN monocatenario negativo (Virus ARNmc- o Virus (-)ssRNA): El ARN monocatenario negativo se convierte en ARNm (que es una cadena monocatenaria positiva) mediante una ARN polimerasa dependiente de ARN aportada por el virus. El ARNm generado se traduce en proteínas reguladoras y estructurales. Las proteínas regulan la replicación del ARN monocatenario negativo a través de una cadena de ARN monocatenario positivo que funciona a modo de molde. Estas cadenas se incluyen en los nuevos virus.
Grupo VI: Virus ARN monocatenario retrotranscrito (Virus ARNmcRT o Virus ssRNA-RT): Este virus integra una transcriptasa inversa que a partir del genoma ARN viral produce una cadena de ADN, primero monocatenario y luego bicatenario, que se integra en el genoma del hospedante. El ADN ya integrado en el hospedante es transcrito a ARNm, que a su vez se traduce en proteínas reguladoras y estructurales. El ADN integrado en el huésped se transcribe en el ARN monocatenario de los nuevos virus.
Grupo VII: Virus ADN bicatenario retrotranscrito (Virus ADNbcRT o Virus dsDNA-RT): El ADN viral entra en el núcleo de la célula, es reparado por la maquinaria de reparación del hospedante y se integra en el genoma del hospedante. El resto de las etapas es similar a las del grupo VI.
.
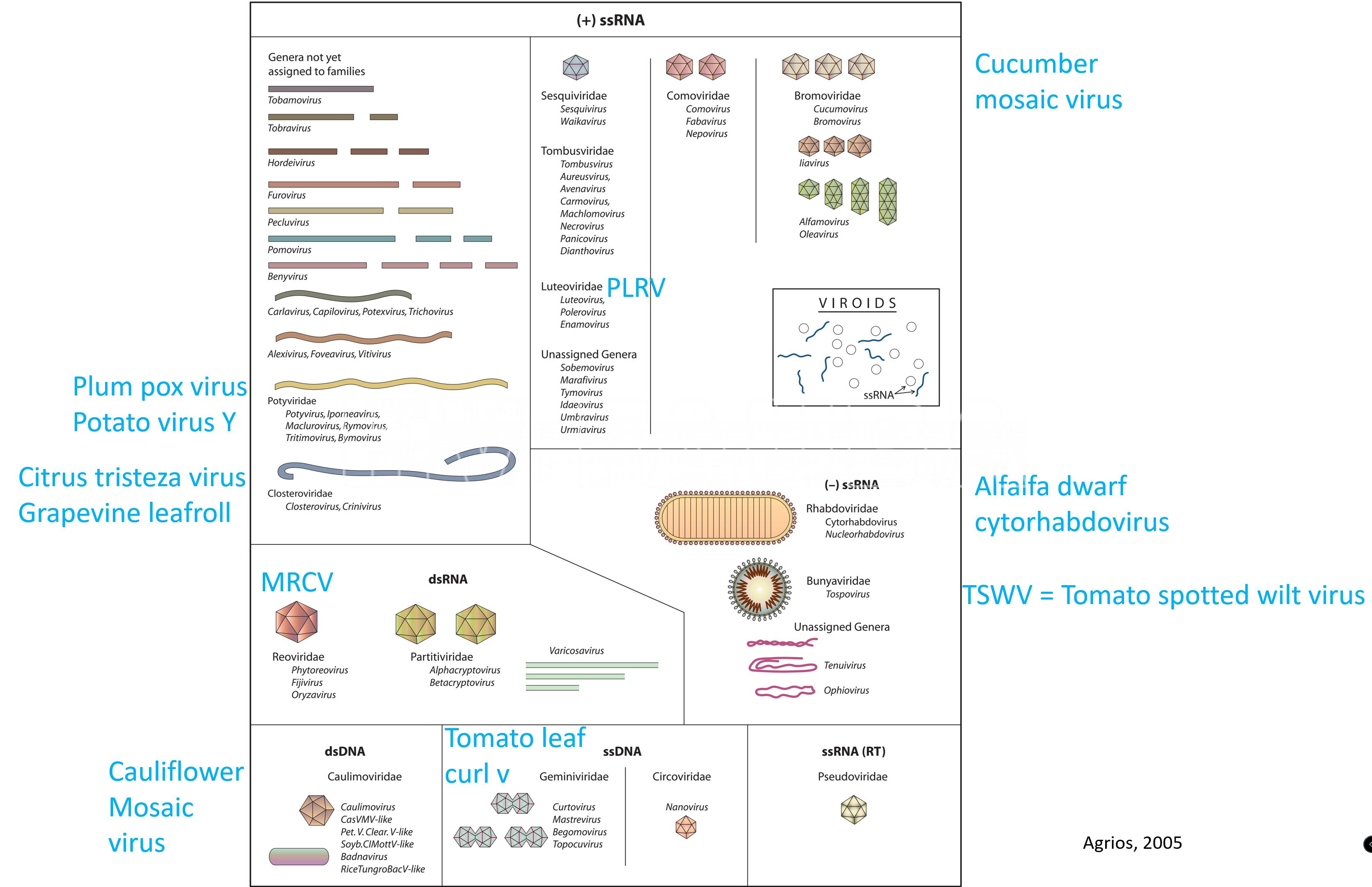
EJEMPLOS DE VIRUS FITOPATOGENOS:
.
Bibliografía
Koonin EV, Krupovic M, Agol VI (2021) The Baltimore Classification of Viruses 50 Years Later: How Does It Stand in the Light of Virus Evolution? Microbiol Mol Biol Rev. 85(3): e0005321. doi: 10.1128/MMBR.00053-21
Wagemans J, Holtappels D, Vainio E, et al. (2022) Going Viral: Virus-Based Biological Control Agents for Plant Protection. Annu Rev Phytopathol. 60: 21-42. doi: 10.1146/annurev-phyto-021621-114208