.
Condición fitosanitaria: Plaga no cuarentenaria reglamentada
Transmisión: áfidos, semipersistente.
Etiología (agente causal):
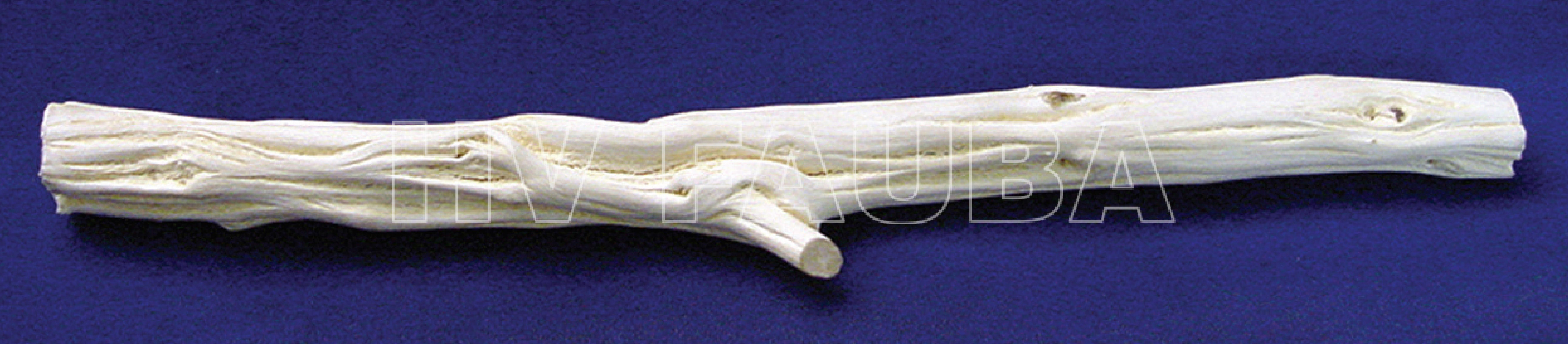
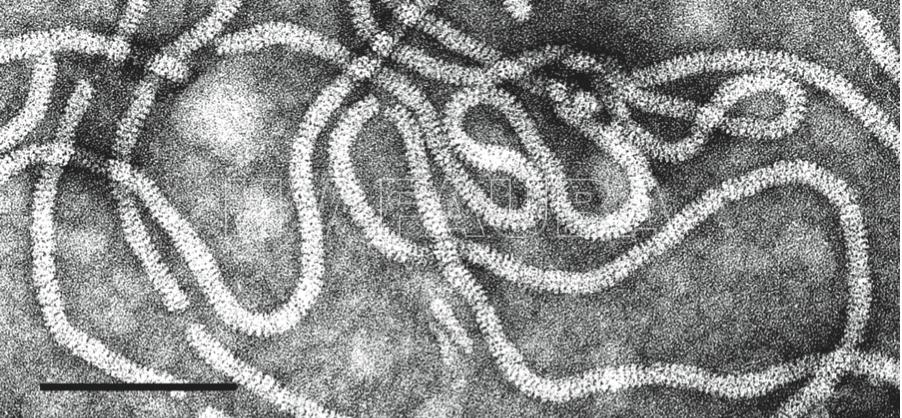
CTV (Citrus tristeza virus), miembro del grupo closterovirus cuya partícula es alargada y flexuosa de aproximadamente 2.500 nm de longitud. El genoma fue secuenciado por Karasev et al. en 1995. El CTV tiene viriones largos y flexibles (2000 nm × 10–12 nm) que están encapsidados por dos proteínas de la cápside. El genoma de ARN monocatenario de CTV, que es de ~ 19,3 kb, codifica doce marcos de lectura abiertos (ORF). Diez ORF del extremo 3′ codifican las siguientes proteínas: proteínas de recubrimiento mayor (CP) y menor (CPm), p65 [homólogo de la proteína de choque térmico 70 (HSP70)] y p61 que están involucradas en el ensamblaje de viriones (Tatineni et al., 2000); una proteína p6 hidrófoba con una función propuesta en el movimiento del virus (Dolja et al., 2006; Tatineni et al., 2008); p20 y p23, que junto con CP son supresores del silenciamiento de ARN (Lu et al., 2004); y p33, p13 y p18, que desempeñan un papel en la extensión del rango de hospedantes del virus (Tatineni et al., 2011). Sin embargo, los árboles de la mayoría de las variedades de cítricos pueden infectarse con mutantes que tienen los genes de las últimas tres proteínas eliminadas (Tatineni et al., 2008).
.
Taxonomía:
Viruses > ssRNA viruses > ssRNA positive-strand viruses > no DNA stage > Closteroviridae > Closterovirus > Citrus tristeza virus
ICTV Virus Taxonomy: 2019 Release
Closteroviridae. ICTV 9th Report (2011)
Baltimore classification: Group IV: ssRNA(+) Baltimore, 1971
.
Hospedantes:
Citrus aurantium (naranjo agrio), Citrus x paradisi (toronja), Citrus reticulata (mandarina), Citrus sinensis (naranjo de ombligo), Citrus aurantiifolia (lima), Citrus latifolia (lima de Tahití), Citrus limon (limón), Citrus grandis (pomelo), Citrus nobilis (tangor), Citrus reticulata x paradisi (tangelo), Citrus madurensis (calamondin), Fortunella margarita (oval kumquat).
Híbridos inter-específicos e híbridos inter-genéricos. Muchos de los hospederos naturales de CTV son asintomáticos cuando se infectan.
Los hospedantes naturales de CTV incluyen esencialmente especies de los géneros Citrus y Fortunella, pero se ha detectado infección por virus en parientes de cítricos inoculados experimentalmente de los géneros Aegle, Aeglopsis, Afraegle, Atalantia, Citropsis, Clausena, Eremocitrus, Hesperthusa, Merrillia, Microcitrus, Pamburus, Pleiospermium y Swinglea (Duran-Vila y Moreno, 2000; Timmer et al., 2000; Yoshida, 1996). También se logró la infección experimental en las especies no cítricas Passiflora gracilis y Passiflora coerulea utilizando vectores de áfidos (Price, 1966; Roistacher et al., 1988), y en protoplastos u hojas agroinfiltradas de Nicotiana benthamiana (Gowda et al., 2005; Navas- Castillo et al., 1997).
Se ha observado resistencia a la mayoría de las cepas de CTV en Poncirus trifoliata (L.) Raf. (Mestre et al., 1997; Rai, 2006) y se ha reportado resistencia a cepas específicas de CTV en Meiwa kumquat (Fortunella crassifolia Swing.) (Mestre et al., 1997), algunos pomelos [C. grandis (L.) Osb.] (Fang y Roose, 1999; Garnsey et al., 1996) y naranjo agria (Asíns et al., 2004). Sin embargo, se ha observado la replicación de CTV en protoplastos de P. trifoliata, pomelo o naranjo agria, lo que sugiere que solo el movimiento de CTV se ve afectado en estas especies (Albiach-Martíet al., 2004).
Pies susceptibles: naranjo agrio (Citrus aurantium) y alemow (C. macrophylla).
Portainjertos tolerantes: TRIFOLIO o naranjo espinoso o trifoliado (Poncirus trifoliata, sin. Citrus trifoliata), Trifoliata Flying Dragon, lima Rangpur (C. limonia), mandarino Cleopatra (Citrus reshni), limonero rugoso (C. jambhiri), CITRANGES (C. sinensis x P. trifoliata) (citrange Troyer, citrange Carrizo), CITRUMELO (C. paradisi x P. trifoliata), CITRUMELO CPB-4475- SWINGLE, Citrus × volkameriana, Forner Alcaide N°5.
El CTV es el patógeno viral más grave de los cítricos y provoca una amplia gama de síntomas de enfermedades devastadoras. Los cultivares de pomelo (Citrus x paradisi) son los más susceptibles a los efectos de las infecciones por CTV entre los cítricos.
.
Epidemiología: es considerada una enfermedad poliética
.
- Diagrama esquemático de la organización genética del CTV que muestra los ORF (recuadros abiertos) de cada gen. PRO, proteasas de tipo papaína; MT, dominio de tipo metil transferasa; IDR, región interdominio grande; HEL, dominio de tipo helicasa; RdRp, dominio de ARN polimerasa dependiente de ARN; HSP70h, análogo a la proteína de choque térmico; CPm y CP, proteínas de la cubierta menores y mayores. A continuación se muestran los productos proteicos esperados y los 10 ARN subgenómicos (el ORF traducido se muestra entre paréntesis). La izquierda y la derecha son análisis de hibridación Northern de ARN totales hibridados usando una sonda específica de 5 ‘(izquierda) o una sonda específica de 3’ (derecha). Autor: Dawson et al., 2013.
.
.
.
Antecedentes
El virus de la tristeza de los cítricos (CTV) es el patógeno viral considerado como el más importante de los cítricos por los daños que ha causado a nivel económico. Hasta fines del siglo XIX el naranjo dulce (Citrus sinensis) generalmente sin injertar, constituyó la base para la producción de naranjas. Como era susceptible a la “podredumbre del pie” causada por Phytophthora sp., muchos países cambiaron el pie o portainjerto por el de naranjo agrio (C. aurantium), ya que éste era resistente al ataque de ese hongo. Sin embargo, este portainjerto muy difundido en el mundo por sus características de dar buenas producciones; resistencia al frío; enfermedades virósicas y nematodes, trajo como consecuencia la aparición de la enfermedad denominada “tristeza”, con graves perjuicios económicos en nuestro país y Brasil. El virus de la tristeza de los cítricos (CTV) ha provocado la muerte de millones de árboles injertados en naranjo agrio (Citrus aurantium). Se cambió nuevamente el portainjerto del agrio a otros resistentes a la tristeza, pero esto causó la aparición de otras enfermedades que antes se desconocían o carecían de importancia, como xiloporosis y exocortis, ambas causadas por viroides. La tristeza está presente en casi todas las zonas citrícolas del mundo. En nuestro país apareció por primera vez en 1930 y durante 15 años destruyó 8 millones de plantas.
El virus se originó aparentemente en Asia, continente que es lugar original de los cítricos y se ha extendido a muchos países por medio del comercio de material vegetal infectado.
Las primeras epidemias de este virus se registran en Sudáfrica hacia fines del siglo XIX, y se publica que las plantaciones de naranjo dulce, mandarino o pomelo, injertadas sobre pies de naranjo agrio morían irremisiblemente en un plazo de dos o tres años. Se atribuyó en un principio esta afección a una incompatibilidad entre injerto y patrón, pero rápidamente tuvo que abandonarse esta hipótesis al observarse que en otros países – España por ejemplo – estas mismas combinaciones formaban plantaciones vigorosas y de excelente producción.
A partir de ese momento se emiten múltiples hipótesis para explicar dicha anormalidad, tales como: presencia de nematodes en raíces, acción nociva de hongos en el sistema radicular (como consecuencia de la desaparición de las raicillas secundarias, deficiencia de macro y microelementos, etc.). Pero ante los resultados negativos obtenidos en las experiencias llevadas a cabo para comprobarlas, comenzó a perderse interés y pronto se abandonaron. En 1928, Toxopeus describe los mismos síntomas en Java (Indonesia), postulando que la anormalidad era debida a que el naranjo dulce producía alguna sustancia tóxica para el portainjerto de naranjo agrio, sin mencionar su naturaleza. En 1930 se la reportó en Argentina, a raíz de una importación de naranjos injertados sobre pie de limón rugoso procedentes de África del Sur (Zeman, 1930). En 1937 aparece en Brasil (Moreira, 1942) y dos años más tarde ocasionó los primeros estragos en California (USA.), Uruguay, Paraguay y Australia. En 1964, Fawcet y Wallace determinaron por primera vez su naturaleza virósica al demostrar que era transmisible por injerto. Simultáneamente Meneghini, en Brasil, señala su trasmisión por el áfido Toxoptera citricidus. En este momento no existe país citrícola alguno en el mundo que pueda ser excluido de este peligroso azote de los agrios. Por alguna razón hay países como Turquía, gran productor de cítricos, donde la mayoría de plantaciones están injertadas sobre naranjo agrio y la tristeza no es un problema grave. Investigadores en la enfermedad sostienen que antes o después, la enfermedad se manifestará produciendo daños a gran escala.
La enfermedad recibe distintos nombres: tristeza de los naranjos agrios, podredumbre de las raicillas, mal seco de los agrios. El nombre más común en inglés es “quick decline of citrus”.
En Argentina, el INTA Concordia tuvo un rol clave en el estudio de la «podredumbre de las raicillas», conocida luego como «tristeza de los cítricos», el cual insumió mucho tiempo, ya que originalmente era atribuida a problemas nutricionales. Estos trabajos de investigación posteriormente dieron origen a un convenio con la Universidad de Florida, Estados Unidos, la cual envió profesionales al país. Esta enfermedad es considerada uno de los primeros casos de enfermedades causadas por virus estudiados en el país.
.
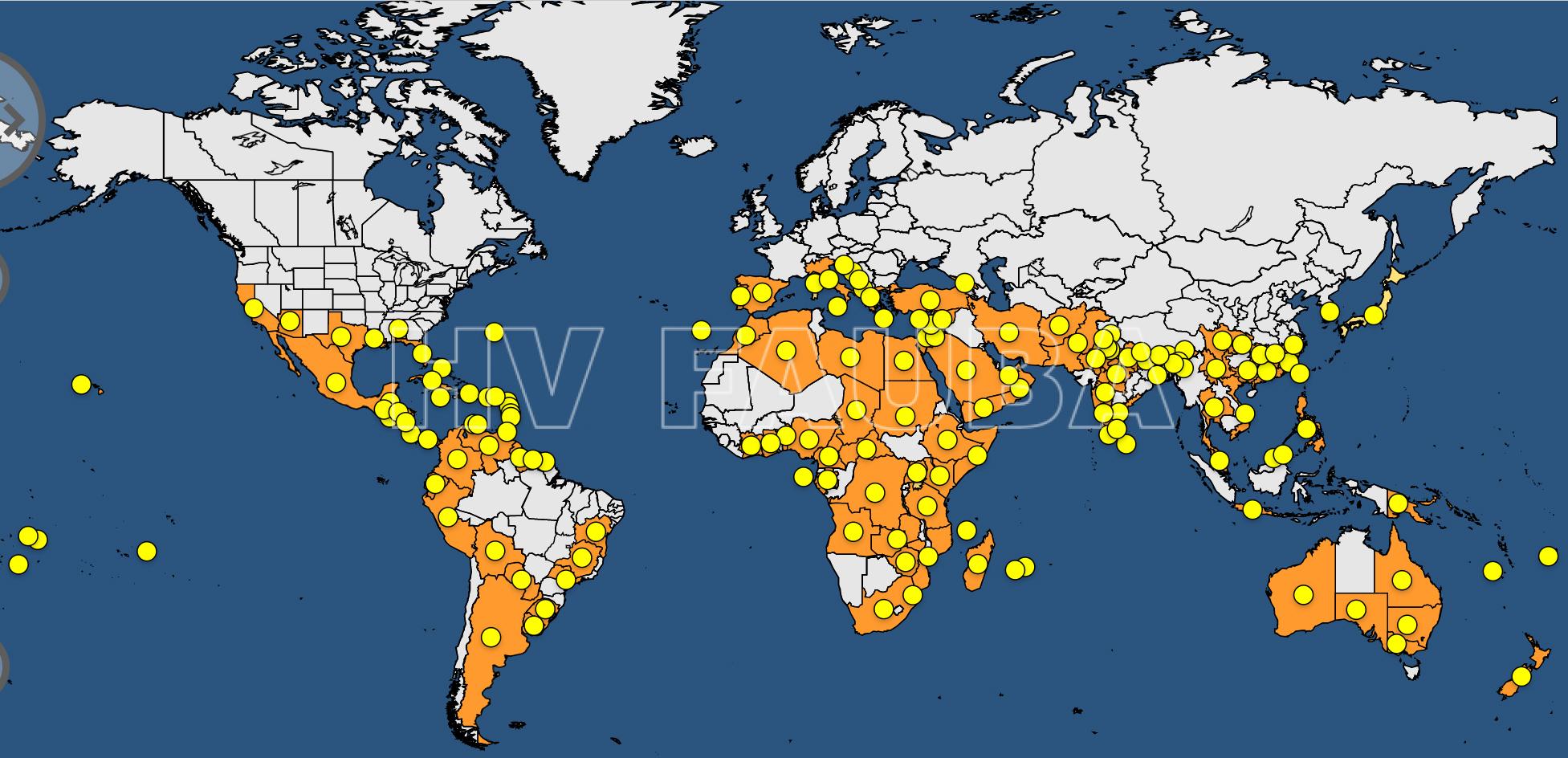
Distribución geográfica
La tristeza se encuentra en casi todos los países citrícolas del mundo.
Está ampliamente difundida en Sudamérica (en la mayoría de los países productores de cítricos). Hay países como Chile, con reciente producción de cítricos, donde no ha aparecido la enfermedad.
Asimismo se encuentra en países productores de cítricos ubicados en la cuenca del Caribe y del Mediterráneo.
.
- Distribución mundial de CTV. Fuente: EPPO
.
Importancia económica
CTV es el virus económicamente más importante en el mundo. Millones de árboles de cítricos en pie de naranjo agrio han muertos por epidemias de CTV en Argentina, Brasil, Venezuela, Perú, Florida, California, Israel, España, y otros lugares.
En Argentina, en la década del 30-60, murieron 16 millones de árboles injertados sobre naranjo agrio. En regiones donde existe el virus y se halla el principal vector Toxoptera citricidus, la pérdida de las plantaciones cítricas de naranjo agrio indefectiblemente ocurre en un plazo más o menos breve. La muerte en plena producción de plantas sobre pie agrio ha sido prácticamente total, desastre único en la historia de la fitopatología Argentina, causado por un solo agente. Vallega y Chiarappa (1964) calcularon las pérdidas ocurridas en Argentina en el orden de los 350.000.000 de U$A.
Pocas enfermedades han provocado tanta alarma entre los productores citrícolas del mundo entero como la causada por la tristeza.
Actualmente en Argentina ha disminuído la intensidad epidemiológica de esta enfermedad en las plantaciones cítricas del pais, debido por un lado a la gran cantidad de plantas cítricas certificadas que se han comercializado (unos tres millones de plantas por año aprox., tanto para iniciar montes nuevos como para replante), y porque si bien se trata de una enfermedad endémica, las cepas actuales de CTV son más suaves y se usan portainjertos tolerantes como el Mandarino Cleopatra, Trifolio, etc.
.
Daños indirectos
Imposibilidad de seguir utilizando naranjo agrio como pie de injerto o patrón en las zonas citrícolas donde esté presente la enfermedad.
Por tanto, se observa que esta virosis puede ser comparada, en sus efectos, con los producidos a finales de siglo XIX por el hongo causante de la gomosis (Phytophthora spp).
.
Sintomatología
El virus produce efectos letales en ciertas combinaciones de injertos y porta injertos como naranjo dulce, pomelo y mandarino, injertado en naranjo agrio.
Existe bastante variación en cuanto al desarrollo de síntomas, según la variedad de árbol y la raza de CTV. Las muchas combinaciones de cítricos y sus parientes, usados como pie y como material de injerto (escisiones) dificultan el reconocimiento de la tristeza.
Las partes de la planta afectadas son las hojas, tallos, raíces y frutos. Las etapas fenológicas afectadas son el crecimiento del plantín, crecimiento vegetativo, floración y fructificación.
Al iniciarse la enfermedad las hojas se tornan cloróticas, coriáceas con la nervadura central prominente. La brotación se detiene, el follaje se ralea y la planta decae notoriamente, razón por la cual los autores brasileños la denominaron tristeza. Las plantas atacadas florecen intensamente pero se forman muy pocas frutas, que no alcanzan su tamaño normal. Con el avance de la enfermedad las hojas se caen, se va muriendo la parte aérea, hasta que finalmente la planta se seca. Todo este proceso ocurre en un lapso de tres a cinco años, según la variedad y raza del virus. En las raíces se produce un proceso de desintegración. Hay necrosis en los tubos cribosos del floema por debajo de la unión del injerto, interrumpiéndose la traslocación de hidratos de carbono.
En ataques severos, los frutos pierden calidad. Algunas razas de CTV causan un síntoma conocido con el nombre de “empeoramiento” en árboles de naranjo dulce, mandarinas o pomelo injertadas en pie de naranjo agrio. El empeoramiento puede ser rápido o puede tomar años. Los síntomas en la copa del árbol incluyen marchitez, clorosis y frutos pequeños que pueden permanecer en árboles muertos por el empeoramiento rápido.
Las plantas de cítricos con decaimiento o tristeza muestran necrosis del floema debajo de la unión de la yema.
.
Acanaladuras del tallo (del Inglés: stem pits o stem pitting):
Otros síntomas pueden ser observados al sacar una sección de corteza de la cicatriz del injerto (unión del injerto y el pie). Árboles que empeoran lentamente (decaimiento), a menudo presentan corteza más gruesa inmediatamente debajo de la cicatriz, donde la cara de la superficie de la corteza tiene muchos pequeños agujeros cónicos (como panal). Los árboles afectados por un decaimiento o empeoramiento rápido carecen de agujeros cónicos, pero frecuentemente presentan una decoloración amarillo-café en la cicatriz. Los agujeros son depresiones pequeñas en la leña exterior, que corresponden a proyecciones que salen de la cara interior de la corteza. Las etapas tempranas son visibles solo al sacar la corteza. Cuando existen muchos agujeros es parecido a una lija fina. Hay una invasión general del cambium. El agujereamiento severo de tallo reduce el vigor del árbol; tiene copas ralas, baja calidad y menor producción de frutos pero no mata a los árboles. Pomelos y limas son muy sensibles al agujereamiento del tallo. El naranjo dulce es más tolerante, pero es severamente afectado por algunas razas de CTV.
Los hoyos o acalanaduras del tallo son el resultado de malformaciones de tejido no lignificado «engomado» colonizado por virus, que se inician por la invasión del virus en múltiples capas de tejido separadas espacialmente: protofloema, metafloema y, al contrario de lo que se creía anteriormente, metaxilema (Sun et al., 2021). En particular, la invasión de CTV en las células del metaxilema no especializadas interrumpe la diferenciación de los elementos traqueales del xilema. Con la propagación radial de CTV en las células adyacentes hacia la periferia del tallo, los grupos de células inmaduras del metaxilema colonizadas por virus se expanden en tamaño, fusionándose, en una determinada etapa, con las células portadoras de virus en las capas de protofloema y metafloema.
Cuando el material infectado con CTV es injertado en plantines de naranjo agrio, las plantas pueden crecer enanas y cloróticas, a pesar de que raras veces se mueren.
En naranjo agrio y toronja (pomelo rosado) puede ocurrir amarillamiento de plantines.
Otros síntomas: la muerte de ramas ocurre en limas severamente afectadas. En árboles crónicamente infectados, las ramas grandes y pequeñas se quiebran fácilmente y a veces son torcidas.
Respecto a la virulencia, la misma es diferente según la raza de CTV y es influenciada por el ambiente y los hospedantes. Las razas de CTV que aparecen naturalmente varían considerablemente en su capacidad para causar síntomas en diferentes hospedantes y en la intensidad de los síntomas expresados. Algunas razas son leves y no producen ningún efecto observable.
.
- Síntomas de la enfermedad causados por el virus Citrus tristeza (CTV). (a) Un árbol que exhibe decaimiento. (b) Un campo con árboles en decaimiento. (c) Aclaramiento de nervaduras en la hoja superior. (d) Ahuecamiento de hojas. (e) Taponamiento de venas. Fuente: Dawson et al. (2015)
- Síntomas de la Tristeza de los Cítricos causados por el virus Citrus tristeza (CTV). (a) Una serie que muestra los síntomas en plántulas amarillentas: (1) planta sana; (2) plántulas con amarillos moderados; y (3) plántulas con amarillos severos. (b) Síntomas de «Stem-pitting» en tallos con la corteza eliminada (1-5): (1) sano; (2, 3, 4) picaduras leves, moderadas y severas; (5) picadura inversa; y (6) pozos de tallo lo suficientemente grandes como para detectarse a través de la corteza. Fuente: Dawson et al. (2015).
- Síntomas de Citrus tristeza virus en Valencia, España. Autor: Pedro Moreno
- «Stem-pitting» en pomelo infectado con CTV. Fuente: http://itp.lucidcentral.org
- Acalanaduras del tejido leñoso. Autor: Ron Brlansky
.
Diagnóstico
Existen distintas formas para realizar el diagnóstico de los virus.
Como se ha visto al estudiar los postulados de Koch, hay dos grandes grupos de pruebas de diagnóstico, aquéllas que usan plantas indicadoras (indexing biológico), realizadas en invernaderos, con condiciones controladas de temperatura y las que se realizan en laboratorio, pudiendo utilizar en este caso métodos bioquímicos o moleculares. Se debe tener en cuenta que un método puede ser sensible para un cierto virus y no para otro.
* Plantas indicadoras
Se injertan yemas de la planta madre que se quiera analizar sobre plantines, porta injerto sanos.
Las plantas madres son aquellas de las que se extraen yemas para obtener plantas sanas de viveros.
La prueba de diagnóstico se debe realizar con una temperatura entre 18 y 24º, debido a que en este rango de temperatura los síntomas se visualizan fácilmente, temperaturas por debajo o arriba de las indicadas, pueden no presentar síntomas a pesar de estar infectadas con el virus.
Como planta indicadora se utiliza la lima Mexicana o lima Key (Citrus aurantifolia).
Síntomas que produce el virus CTV sobre lima mexicana: aclaramiento de las nervaduras, acorchamiento de las nervaduras, abarquillado de hojas, con gomas o sin ellas. La observación se realiza sobre hojas de brotes jóvenes y adultos después de 3 meses de inoculado, por lo menos tres brotaciones.
* Métodos de laboratorio
Inmunoimpresión directa – DAS-ELISA (se emplean anticuerpos monoclonales).
Métodos moleculares (RT-PCR).
.
- Citrus tristeza: micrografía electrónica de transmisión de partículas CTV (12nm de ancho x 2000nm de largo). Autor: M. Bar-Joseph, Volcani Institute of Agricultural Research.
- Micrografía electrónica de contraste negativo de viriones del virus de la tristeza de los cítricos (CTV) (género Closterovirus). La barra representa 100 nm. AUTOR: R.G. Milne.
.
..
Transmisión
Es fácilmente transmisible por injerto, cuando hay contacto entre floemas, cuando se utilizan yemas, trozos de corteza y tejido foliar. Esta forma de transmisión es muy importante en el movimiento a largas distancias con plantas o sus partes infectadas.
Se transmite en forma semipersistente principalmente por áfidos (pulgones), siendo el más importante por su eficiencia en la trasmisión el pulgón negro Aphis (Toxoptera) citricidus Kirkaldy (Harper et al., 2016; Shilts et al., 2020). En Argentina, T. citricidus es endémico. Otros vectores son Aphis gosspii, A. spiraecola y T. aurantii (este último presente en USA). También se transmite por moscas blancas, cochinillas pseudococcidas o cochinillas blandas (Pseudococcidae Heymons, 1915).
Además, tanto la transmisión por injerto como por insectos, son utilizados en ensayos de laboratorio. Otras formas de transmisión en condiciones controladas, pero no en la naturaleza, es por Cuscuta spp. y por transmisión mecánica, este último de baja eficacia.
El CTV no se transmite por semilla.
.
.
- Aphis citricidus (=Toxoptera citricida). Autores: izq arriba, Sunil Joshi & Poorani, J. Aphids of Karnataka.; der arriba, Marina VM; izq abajo, Marco Gaiani; centro abajo, Centre for Invasive Species Research; der abajo, Florida Division of Plant Industry Archive, Florida Department of Agriculture and Consumer Services,
.
.
La resistencia al CTV está mediada por el ácido salicílico y las vías de defensa de silenciamiento de ARN
Las plantas cítricas con “decaimiento” o “empeoramiento” muestran necrosis del floema debajo de la unión de la yema injertada. Por otro lado, el naranjo agrio es susceptible al CTV pero se considera mínimamente susceptible al CTV en comparación con otras variedades de cítricos, lo que sugiere que posee resistencia parcial (tolerancia). El silenciamiento de los genes RDR1, NPR1 y DCL2 / DCL4, asociados con el silenciamiento del ARN y las vías de defensa del ácido salicílico (SA), mejoró la propagación y acumulación de virus en plantas de naranjo agrio en comparación con los controles no silenciados, mientras que el silenciamiento de los genes NPR3 / NPR4 , asociado con la respuesta hipersensible, produjo una ligera disminución en la acumulación de CTV y redujo el retraso del crecimiento del naranjo agrio injertado en plantas de limón áspero infectadas con CTV. También se demostró que los supresores de silenciamiento de ARN de CTV, p20 y p23 también suprimen las vías de señalización de defensa activadas por el ácido salicílico, siendo la actividad supresora mayor en los aislamientos más virulentos (Gómez-Muñoz et al., 2017).
.
Epidemiología
El CTV es propagado por el movimiento de plantas y/o sus partes infectadas y por áfidos vectores. Un factor decisivo es la raza del virus. En segundo lugar está la variedad del árbol. Por ejemplo, la tasa de diseminación de CTV en naranjos dulces usualmente es más alta que la tasa en pomelos. Infecciones primarias de CTV usualmente se establecen a través de la propagación de plantas infectadas. T. citricidus es el vector más eficiente para la transmisión de árbol a árbol. El número de hospederos y el comportamiento alimenticio del áfido afectan la forma y la tasa de la diseminación de la enfermedad. La transmisión natural suele ser más lenta en regiones áridas donde el rango de la temperatura limita el desarrollo del inóculo en las plantas. El virión de CTV permanece infectivo en los vectores, junto con la hibernación de pulgones en árboles de cítricos. Las temperaturas suaves de primavera y otoño favorecen el desarrollo de los áfidos en las plantas. Factores que promueven el nuevo crecimiento (fertilización, riego) aumentan las oportunidades para la transmisión. Cuando el vector primario es Toxoptera citricidus, con frecuencia las nuevas infecciones se agrupan cerca del los árboles infectados existentes, mientras que si el vector Aphis gosspii es el vector primario, las nuevas infecciones son más raras.
Desde el punto de vista epidemiológico, es considerada una enfermedad poliética.
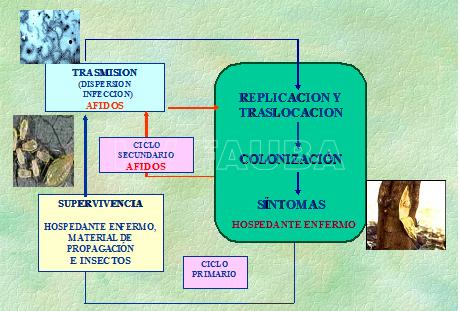
Figura. Ciclo biológico-agronómico de tristeza de los cítricos (Citrus tristeza virus – CTV). (Amorín, L., 1995; Gaetán, SA., 2010)
.
.
Manejo integrado de la enfermedad
Los métodos empleados dependen de si la enfermedad está presente o ausente en un país.
.
Métodos usados en caso que la enfermedad esté ausente
* Cuarentena vegetal
De no existir la enfermedad en un país, estrictos programas de cuarentena vegetal son eficientes pues esta media se baja en la “exclusión” que significa no permitir el ingreso de plantas infectadas; esta labor la llevan a cabo los organismos nacionales de prevención fitosanitaria, presente en todos los países (como el SENASA, pero en este caso no aplica para la enfermedad).
* Monitoreo de la enfermedad
* Erradicación de plantas enfermas sintomáticas
Métodos usados en caso que la enfermedad esté presente
* Comportamiento varietal
Pies susceptibles: naranjo agrio (Citrus aurantium) y alemow (C. macrophylla).
Portainjertos tolerantes: TRIFOLIO o naranjo espinoso o trifoliado (Poncirus trifoliata, sin. Citrus trifoliata) , lima Rangpur (C. limonia), mandarino Cleopatra (Citrus reshni), limonero rugoso (C. jambhiri), CITRANGES (C. sinensis x P. trifoliata) (citrange roger, citrange carrizo), CITRUMELO (C. paradisi x P. trifoliata).
Variedades cítricas injertadas sobre naranjo agrio son susceptibles al virus. El naranjo amargo es muy sensible, por lo que no debe usarse, con excepción de limón sobra amargo (esta combinación es resistente en regiones donde existe el virus, se halla el vector y se da la combinación susceptible de injerto-portainjerto, la pérdida de plantación cítricas ocurre en un plazo más o menos breve.
Dada la existencia de muchas razas virósicas y la constante mutación que en ellos ocurre, es posible la aparición de daños en combinaciones injerto-portainjerto hasta ahora tolerantes.
.
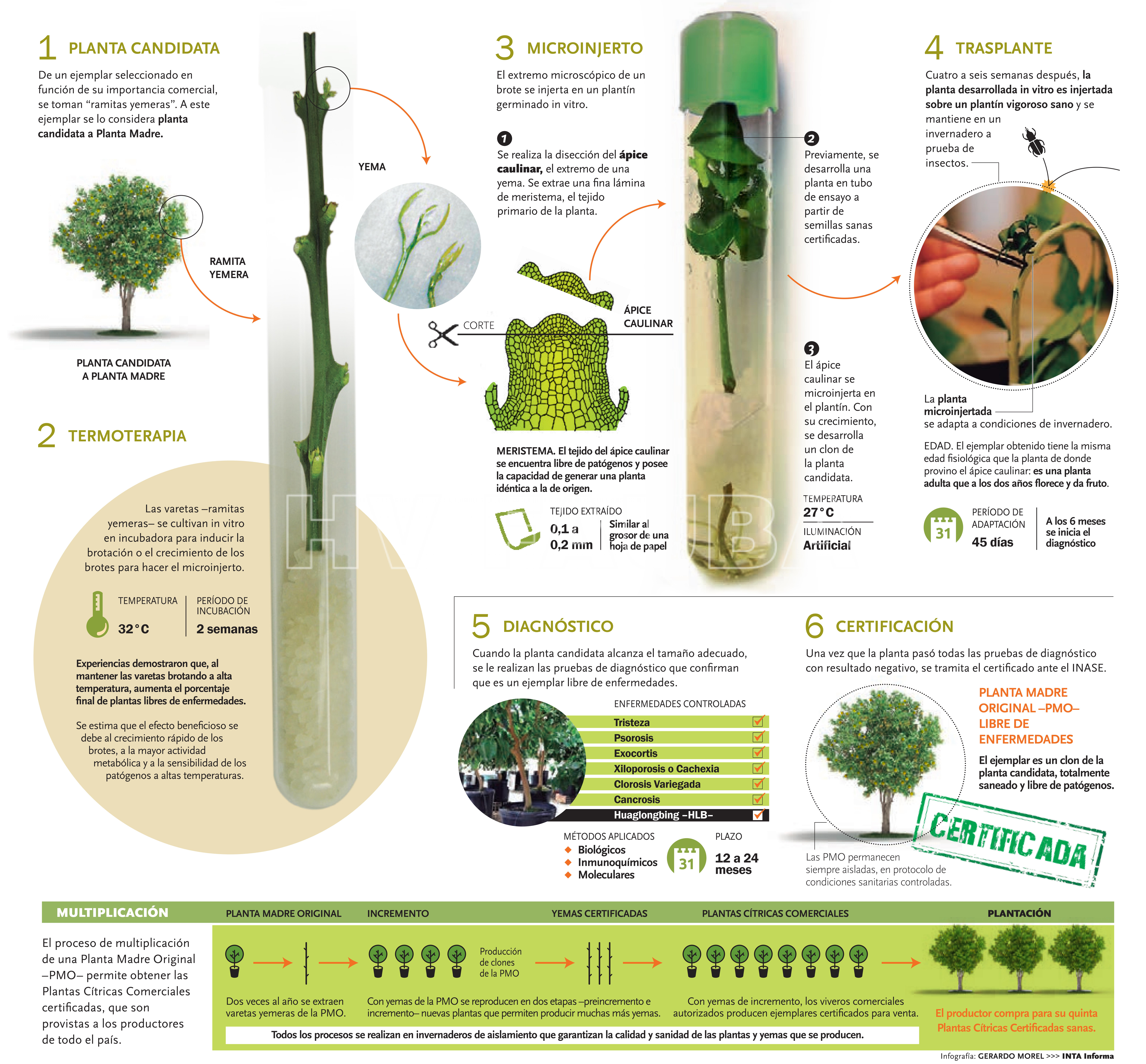
* Programas de certificación de viveros
En Argentina la certificación de cítricos es obligatoria. En 1998 el INASE y el SENASA desarrollaron un Programa de Certificación de Cítricos para la Argentina (Res. 149/98 ex SAGPyA: “Normas para la Producción, Comercialización e Introducción de Plantas Cítricas de Vivero y sus Partes”). Las plantas madres originales (primer eslabón de toda la cadena de viveros, de las cuales se extraen yemas para las futuras plantas de viveros), se encuentran en invernaderos con malla antiáfido y doble puerta de entrada. Existen en nuestro país dos “centros de saneamiento” donde se encuentran las únicas plantas madres libres de este virus y de otras identidades patógenas: uno, en INTA CONCORDIA (Entre Ríos) y otro, en la Estación Experimental Agroindustrial Obispo Colombres (EEAOC) en Tucumán. Estos centros y sus correspondientes laboratorios son habilitados y controlados anualmente por el INSTITUTO NACIONAL DE SEMILLAS (INASE) bajo estrictos controles. Sólo se pide ausencia de CTV en las plantas madres, de tal manera que las plantas que son vendidas en viveros inscriptos pueden tener la enfermedad. Esto es así debido a tres factores:
1) las razas de CTV presentes en nuestro país son suaves.
2) los pies o porta injertos utilizados son tolerantes o resistentes al virus de la tristeza.
3) la tristeza es una enfermedad endémica en Argentina, ya no es posible la erradicación debido a su amplia distribución geográfica.
* El artículo 19 de la Disposición DNPV N° 4/13 SENASA establece que todo productor de fruta tiene la obligatoriedad de demostrar con la Guía de Sanidad para el Tránsito de plantas y/o sus partes el origen del material de propagación plantado.
.
* Control de insectos vectores
Los insecticidas probablemente no evitarán la infección primaria, pero pueden reducir la tasa de transmisión secundaria y pueden ser útiles en viveros de cítricos o para proteger fuentes de material de injerto. Es preferible un insecticida sistémico con un largo período residual y un mínimo impacto sobre los agentes de control biológico. Esta medida es sumamente eficiente cuando la enfermedad recién ha ingresado en una región, donde se realiza la erradicación (extracción de árboles enfermos).
.
* Preinmunización o protección cruzada
La inoculación con una cepa suave de CTV puede proteger un árbol de la infección con una cepa fuerte y de sus síntomas. Esta estrategia se usa para controlar la enfermedad en áreas donde son endémicos los aislados severos de CTV y Toxoptera citricidus. Este método fue utilizado en Brasil. Tiene como desventaja la dispersión de un virus que puede mutar y provocar daños imprevistos.
Dos revisiones actuales de esta práctica pueden encontrarse en: Folimonova, 2013 y Lee y Keremane, 2013.
.
- Programa PROCITRUS INTA para para obtener variedades comerciales certificadas de cítricos que son libres de enfermedades y tienen excelente calidad genética. Esto permite que el productor puede contar con mayores rendimientos por hectárea y extender la vida útil de las plantas. FUENTE: INTA
.
Videos
.
Artículos
2017.Dic.04 To Save Florida’s Famous Oranges, Scientists Race To Weaponize A Virus
2017.May.06 (Ledford H) Geneticists enlist engineered virus and CRISPR to battle citrus disease. Nature News
.
.
.
Bibliografía
Catálogo de portainjertos cítricos. INTA
Akhter MS, Monirul Hasan Tipu M, Rahman MS, et al. (2022) First report of Citrus tristeza virus in Bangladesh. Australasian Plant Dis. Notes 17: 12. doi: 10.1007/s13314-022-00457-z
, , , (2004) Citrus tristeza virus replicates and forms infectious virions in protoplast of resistant citrus relatives. Mol. Breeding 14: 117–128. doi: 10.1023/B:MOLB.0000038000.51218.a7
Asins MJ, Bernet GP, Ruiz C, et al. (2004) QTL analysis of citrus tristeza virus-citradia interaction. Theor Appl Genet. 108(4): 603-11. doi: 10.1007/s00122-003-1486-7
Atta S, Cao M, Umar U, et al. (2017) Biological indexing and genetic analysis of Citrus tristeza virus in Pakistan. Journal of General Plant Pathology 83(6): 382–389. doi: 10.1007/s10327-017-0737-4
Atta S, Umar UUD, Bashir MA, et al. (2019) Application of biological and single-strand conformation polymorphism assays for characterizing potential mild isolates of Citrus tristeza virus for cross protection. AMB Express. 9(1): 174. doi: 10.1186/s13568-019-0903-5
Bar-Joseph M, Garnsey SM, Gonsalves D (1979) The closteroviruses: a distinct group of elongated plant viruses. Adv. Virus Res. 25: 93–168.
Bar-Joseph M, Dawson WO (2008) Citrus tristeza virus, in Encyclopedia of Virology, Third Edition Evolutionary Biology of Viruses, Vol. 1, eds B. W. J. Mahy and M. H. V. van Regenmortel (Amsterdam: Elsevier Ltd), 161–184.
Baruah BR, Kashyap A, Nath PD (2015) Citrus tristeza virus a Worldwide threat to Citriculture: Advances made in its Research and Future thrust. Research Journal of Agriculture and Forestry Sciences 3(8): 7-14. ISSN 2320-6063
Batuman O, Mawassi M, Bar-Joseph M (2006) Transgenes consisting of a dsRNA of an RNAi suppressor plus the 3′ UTR provide resistance to Citrus tristeza virus sequences in Nicotiana benthamiana but not in citrus. Virus Genes. 33(3): 319‐327. doi: 10.1007/s11262-006-0071-y
Benítez-Galeano MJ, Castells M, Colina R (2017) The Evolutionary History and Spatiotemporal Dynamics of the NC Lineage of Citrus Tristeza Virus. Viruses 9(10): 272. doi: 10.3390/v9100272
Brlansky RH, Howd DS, Broadbent P, Damsteegt VD (2002) Histology of sweet orange stem pitting caused by an Australian isolate of Citrus tristeza virus. Plant Disease 86:1169-1174. doi: 10.1094/PDIS.2002.86.10.1169
Brlansky RH, Roy A, Damsteegt VD (2011) Stem-pitting Citrus tristeza virus predominantly transmitted by the brown citrus aphid from mixed infections containing non-stem-pitting and stem-pitting isolates. Plant Disease 95: 913-920. doi: 10.1094/PDIS-10-10-0772
Calvo NS (2013) Transgenic resistance against Citrus tristeza virus (CTV) and analysis of the viral p23 protein as pathogenicity determinant in citrus. Tesis Doctoral, Universitat Politècnica de València, España. 205 p.
Cambra M, Gorris MT, Marroquín C, et al. (2000) Incidence and epidemiology of Citrus tristeza virus in the Valencian community of Spain. Virus Res. 71(1-2): 85-95. doi: 10.1016/S0168-1702(00)00190-8
Catara AF, Bar-Joseph M, Licciardello G (2015) Citrus Tristeza Virus. Methods and Protocols. Humana, New York, NY: Springer Science+Business Media, LLC, part of Springer Nature 2019. doi: 10.1007/978-1-4939-9558-5
, , , et al. (2021) Comparative analysis identifies amino acids critical for citrus tristeza virus (T36CA) encoded proteins involved in suppression of RNA silencing and differential systemic infection in two plant species. Molecular Plant Pathology 22: 64– 76. doi: 10.1111/mpp.13008
Chen L, Liu Y, Wu F, et al. (2023) Citrus tristeza virus Promotes the Acquisition and Transmission of ‘Candidatus Liberibacter Asiaticus’ by Diaphorina citri. Viruses 15(4): 918. doi: 10.3390/v15040918
Chen Y, Yi L, Zhong K, et al. (2023) Population genetic characteristics of citrus tristeza virus from wild mandarins in the Nanling Mountains of China. Trop. plant pathol. 48: 270–282. 10.1007/s40858-023-00567-8
Cheng CZ, Yang JW, Yan HB, et al. (2015) Expressing p20 hairpin RNA of Citrus tristeza virus confers Citrus aurantium with tolerance/resistance against stem pitting and seedling yellow CTV strains. Journal of Integrative Agriculture 14: 1767-1777. doi: 10.1016/S2095-3119(14)60937-0
Costa Aa, Martins Tr, Marques NT, Nolasco G (2016) Combined expression of p20 and p23 proteins from Citrus tristeza virus show enhanced local silencing suppressor activity. Phytopathologia Mediterranea 55(2): 172−183. doi: 10.14601/Phytopathol_Mediterr-16093
Datta S, Das B, Gopalakrishnan R, et al. (2021) Detection of ‘ancestral’ western lineage of Citrus tristeza virus virulent genotype in declining Arunachal Wakro orange. Trop. plant pathol. 46: 493–505. doi:
Davino S, Willemsen A, Panno S, Davino M, Catara A, Elena SF, et al. (2013) Emergence and Phylodynamics of Citrus tristeza virus in Sicily, Italy. PLoS ONE 8(6): e66700. doi: 10.1371/journal.pone.0066700
Dawson WO, Garnsey SM, Tatineni S, Folimonova SY, Harper SJ and Gowda S (2013) Citrus tristeza virus-host interactions. Frontiers in Microbiology 4: 88. doi: 10.3389/fmicb.2013.00088
Dawson WO, Bar-Joseph M, Garnsey SM, Moreno P (2015) Citrus Tristeza Virus: Making an Ally from an Enemy. Annual Review of Phytopathology 53: 137-155. doi: 10.1146/annurev-phyto-080614-120012
Deng Z, Huang S, Ling P, et al. (2001) Fine genetic mapping and BAC contig development for the Citrus tristeza virus resistance gene locus in Poncirus trifoliata (Raf.). Molecular Genetics and Genomics 265(4): 739–747. doi: 10.1007/s004380100471
Dolja VV, Kreuze JF, Valkonen JPT (2006) Comparative and functional genomics of closteroviruses. Virus Res. 117, 38–51. doi: 10.1016/j.virusres.2006.02.002
Dória MS, Sousa AOd, Barbosa CdJ, et al. (2015) Citrus tristeza virus (CTV) Causing Proteomic and Enzymatic Changes in Sweet Orange Variety “Westin”. PLoS ONE 10(7): e0130950. doi: 10.1371/journal.pone.0130950
Duran-Vila N, Moreno P (2000) Enfermedades de los Cítricos. Madrid: Ediciones Mundi-Prensa.
Family – Closteroviridae (2012) Ninth Report of the International Committee on Taxonomy of Viruses. Virus Taxonomy, 987-1001. doi: 10.1016/B978-0-12-384684-6.00085-9
(1999) A novel gene conferring Citrus tristeza virus resistance in Citrus maxima (Burm.) Merrill. HortScience 34: 334–335. doi: 10.21273/HORTSCI.34.2.334
Flores R, Ruiz-Ruiz S, Soler N, et al. (2013) Citrus tristeza virus p23: a unique protein mediating key virus–host interactions. Frontiers in Microbiology 4: 98. doi: 10.3389/fmicb.2013.00098
Folimonov AS, Folimonova SY, Bar-Joseph M, Dawson WO (2007) A stable RNA virus-based vector for citrus trees. Virology 368: 205–216. doi: 10.1016/j.virol.2007.06.038
Folimonova SY, Folimonov AS, Tatineni S, Dawson WO (2008) Citrus tristeza virus: survival at the edge of the movement continuum. J. Virol. 82: 6546–6556. doi: 10.1128/JVI.00515-08
Folimonova SY, Robertson CJ, Shilts T, et al. (2009) Strains of Citrus tristeza virus do not exclude superinfection by other strains of the virus. J. Virol. 84: 1314–1325.
Folimonova SY (2013) Developing an understanding of cross-protection by Citrus tristeza virus. Frontiers in Microbiology 4: 76. doi: 10.3389/fmicb.2013.00076
Folimonova SY (2020) Citrus tristeza virus: A large RNA virus with complex biology turned into a valuable tool for crop protection. PLoS Pathog 16(4): e1008416. doi: 10.1371/journal.ppat.1008416
Fraser L (1952) Seedling yellows, an unreported virus disease of citrus. Agr. Gaz. N.S.Wales 63: 125–131.
Fu S, Shao J, Zhou C and Hartung JS (2017) Co-infection of Sweet Orange with Severe and Mild Strains of Citrus tristeza virus Is Overwhelmingly Dominated by the Severe Strain on Both the Transcriptional and Biological Levels. Frontiers in Plant Science 8: 1419. doi: 10.3389/fpls.2017.01419
Garnsey SM, Su HJ, Tsai MC (1996) Differential susceptibility of pummelo and Swingle citrumelo to isolates of Citrus tristeza virus. In: Proceedings of the 13th Conference of the International Organization of Citrus Virologists (Da Graça, J.V., Moreno, P. and Yokomi., R.K., eds), pp. 138–146. Riverside, CA: IOCV.
Gómez-Muñoz N, Velázquez K, Vives MC, et al. (2017) The resistance of sour orange to Citrus tristeza virus is mediated by both the salicylic acid and RNA silencing defence pathways. Molecular Plant Pathology 18: 1253–1266. doi: 10.1111/mpp.12488
Gonçalves AP, Santos KS, Silva CC, et al. (2018) Identificação de isolados de Citrus tristeza virus (CTV) protetivos para Citrus sinensis (L.) Osbeck. Summa Phytopathologica 44(1): 17-22. doi: 10.1590/0100-5405/172270
Gottwald TR, Cambra M, Moreno P, et al. (1996) Spatial and temporal analyses of citrus tristeza virus in eastern Spain. Phytopathology 86: 45–55.
Gowda S, Satyanarayana T, Ayllón MA, et al. (2003) The conserved structures at the 5′ nontranslated region of Citrus tristeza virus are involved in replication and virion assembly. Virology 317: 50–64. doi: 10.1016/j.virol.2003.08.018
Gowda S, Satyanarayana T, Robertson CJ, et al. (2005) Infection of citrus plants with virions generated in Nicotiana benthamiana plants agroinfiltrated with binary vector based Citrus tristeza virus. In: Proceedings of the 16th Conference of the International Organization of Citrus Virologists (Hilf, M.E., Duran-Vila, N. and Rocha-Peña, M.A., eds), pp. 23–33. Riverside, CA: IOCV.
Hajeria S, Killiny N, El-Mohtara C, et al. (2014) Citrus tristeza virus-based RNAi in citrus plants induces gene silencing in Diaphorina citri, a phloem-sap sucking insect vector of citrus greening disease (Huanglongbing). Journal of Biotechnology 176: 42-49. doi: 10.1016/j.jbiotec.2014.02.010
Harper SJ, Yokomi RK, Dawson WO (2016) CHAPTER 9: Citrus tristeza virus–Aphid Interactions. Vector-Mediated Transmission of Plant Pathogens. pp. 121-130. doi: 10.1094/9780890545355.009
Herron CM, Mirkov TE, da Graça JV, Lee RF (2006) Citrus tristeza virus transmission by the Toxoptera citricida vector: In vitro acquisition and transmission and infectivity immunoneutralization experiments. Journal of Virological Methods 134: 205-211. doi: 10.1016/j.jviromet.2006.01.006
Huang Z, Rundell PA, Guan X, Powell CA (2004) Detection and Isolate Differentiation of Citrus tristeza virus in Infected Field Trees Based on Reverse Transcription-Polymerase Chain Reaction. Plant Disease 88(6): 625-629. doi: 10.1094/PDIS.2004.88.6.625
Iglesias NG, Riquelme Barrera KL, Marengo JM, et al. (2005) Genetic structure of Citrus tristeza virus (CTV) populations from field Argentinian grapefruit isolates. International Organization of Citrus Virologists Conference Proceedings (1957-2010) Volume 16, Issue 16 143-149. Link
Kang S-H, Dao TNM, Kim O-K, Folimonova SY (2017) Self-interaction of Citrus tristeza virus p33 protein via N-terminal hélix. Virus Research 233(2): 29-34. doi: 10.1016/j.virusres.2017.03.008
Karasev AV, Boyko VP, Gowda S, et al. (1995) Complete sequence of the citrus tristeza virus RNA genome. Virology. 1995;208(2):511‐520. doi: 10.1006/viro.1995.1182
King AMQ, Adams MJ, Carstens EB, et al. (2012) Family – Closteroviridae. Virus Taxonomy, pp. 987-1001. doi: 10.1016/B978-0-12-384684-6.00085-9
Korkmaz S, Cevik B, Onder S, Koc K, Bozan O (2008) Detection of Citrus tristeza virus (CTV) from Satsuma Owari mandarins (Citris unshiu) by direct tissue blot immunoassay (DTBIA), DAS‐ELISA, and biological indexing. New Zealand Journal of Crop and Horticultural Science 36: 239-246. doi: 10.1080/01140670809510240
Lee RF and Keremane ML (2013) Mild strain cross protection of tristeza: a review of research to protect against decline on sour orange in Florida. Frontiers in Microbiology 4: 259. doi: 10.3389/fmicb.2013.00259
López C, Cervera M, Fagoaga C, et al. (2010) Accumulation of transgene-derived siRNAs is not sufficient for RNAi-mediated protection against Citrus tristeza virus in transgenic Mexican lime. Mol Plant Pathol. 11(1):33‐41. doi: 10.1111/j.1364-3703.2009.00566.x
Lu R, Folimonov A, Shintaku M, et al. (2004) Three distinct suppressors of RNA silencing encoded by a 20-kb viral RNA genome. Proceedings of the National Academy of Sciences of the United States of America 101(44): 15742–15747. doi: 10.1073/pnas.0404940101
Matsumura EE, Coletta-Filho HD, Nouri S, et al. (2017) Deep Sequencing Analysis of RNAs from Citrus Plants Grown in a Citrus Sudden Death-Affected Area Reveals Diverse Known and Putative Novel Viruses. Viruses 9(4): 92. doi: 10.3390/v9040092
, , , (1997) Molecular markers flanking citrus tristeza virus resistance gene from Poncirus trifoliata (L.) Raf. Theor. Appl. Genetics, 94: 458–464. doi: 10.1007/s001220050437
, , Navarro L (1997) Efficient search for new resistant genotypes to the citrus tristeza closterovirus in the orange subfamily Aurantioideae. Theor. Appl. Genetics 95: 1282–1288. doi: 10.1007/s001220050694
Moreira, S. 1942. Observacoes sobre a «tristeza» dos citrus ou «podridao das radicelas». O. Bioi. 8:269-72
Moreno P, Ambrós S, Albiach-Martí MR, et al. (2008) Citrus tristeza virus: a pathogen that changed the course of the citrus industry. Molecular Plant Pathology 9: 251–268. doi: 10.1111/j.1364-3703.2007.00455.x
Munir N, Hameed AA, Haq R, Naz S (2019) Biochemical changes in cultivars of sweet oranges infected with citrus tristeza virus. Braz. J. Biol. [Internet]. 2019 Nov [cited 2020 May 28] ; 79( 4 ): 742-748. doi: 10.1590/1519-6984.193791
, , , (1997) Kinetics of accumulation of Citrus tristeza virus RNAs. Virology 228: 92–97. doi: 10.1006/viro.1996.8369
Nicaise V (2014) Crop immunity against viruses: outcomes and future challenges. Frontiers in Plant Science 5: 660. doi: 10.3389/fpls.2014.00660
Palacios MF, Figueroa J, Foguet L, Villafañe L, Stein B (2020) Estudio de la población del virus de la tristeza de los cítricos en cultivares de limoneros injertados en naranjo agrio y Citrus macrophylla. Rev. Ind. y Agríc. de Tucumán 97 (1): xx-xx.
Pennazio S, Roggero P, Conti M (1996) Yield losses in virus‐infected crops. Archives of Phytopathology and Plant Protection 30: 283-296. doi: 10.1080/03235409609383178
Price WC (1966) Flexuous rods in phloem cells of lime plants infected with citrus tristeza virus. Virology 29: 285–294. doi: 10.1016/0042-6822(66)90035-3
(2006) Refinement of the Citrus tristeza virus resistance gene (Ctv) positional map in Poncirus trifoliata and generation of transgenic grapefruit (Citrus paradisi) plant lines with candidate resistance genes in this region. Plant Mol. Biol. 61: 399–414. doi: 10.1007/s11103-006-0018-7
Read DA, Palacios MF, Figueroa J, et al. (2017) Survey of citrus tristeza virus (CTV) strains in Citrus x limon (L) Burm f. (lemon) in Tucumán Province, Argentina. Eur J Plant Pathol 149: 1029–1039. doi: 10.1007/s10658-017-1245-2
Read DA, Palacios MF, Kleynhans J, et al. (2018) Survey of Citrus tristeza virus (CTV) diversity in pigmented Citrus x paradisi (Macfad.) (Grapefruit) trees in north-western Argentina. Eur J Plant Pathol 151: 329–340. doi: 10.1007/s10658-017-1376-5
Read DA, Palacios MF, Figueroa J, et al. (2018) Survey of Citrus tristeza virus (CTV) on Citrus paradisi (Macfad.) cv. “Marsh” in South Africa. Eur J Plant Pathol 151: 1101–1105. doi: 10.1007/s10658-018-1433-8
Roistacher CN, Dodds JA, Bash JA (1988) Cross protection against citrus tristeza seedling yellows and stem pitting viruses by protective isolates developed in greenhouse plants. In: Proceedings of the 10th Conference of the International Organization of Citrus Virologists (Timmer, L.W., Garnsey, S.M. and Navarro L., eds), pp. 91–100. Riverside, CA: IOCV.
Rosa C, Polek M, Falk BW, Rowhani A (2007) Improved Efficiency for Quantitative and Qualitative Indexing for Citrus tristeza virus and Citrus psorosis virus. Plant Disease 91(9): 1089-1095. doi: 10.1094/PDIS-91-9-1089
Roy A, Fayad A, Barthe G, Brlansky RH (2005) A multiplex polymerase chain reaction method for reliable, sensitive and simultaneous detection of multiple viruses in citrus trees. J Virol Methods. 129(1): 47-55. doi: 10.1016/j.jviromet.2005.05.008
Roy A, Ananthakrishnan G, Hartung JS, Brlansky RH (2010) Development and application of a multiplex reverse-transcription polymerase chain reaction assay for screening a global collection of Citrus tristeza virus isolates. Phytopathology. 100(10): 1077-88. doi: 10.1094/PHYTO-04-10-0102
Ruiz-Ruiz S, Moreno P, Guerri J, Ambrós S (2007) A real-time RT-PCR assay for detection and absolute quantitation of Citrus tristeza virus in different plant tissues. Journal of Virological Methods 145: 96-105. doi: 10.1016/j.jviromet.2007.05.011
Ruiz-Ruiz S, Navarro B, Gisel A, Peña L, Navarro L, Moreno P, Di Serio F, Flores R (2011) Citrus tristeza virus infection induces the accumulation of viral small RNAs (21-24-nt) mapping preferentially at the 3′-terminal region of the genomic RNA and affects the host small RNA profile. Plant Molecular Biology 75(6): 607–619. doi: 10.1007/s11103-011-9754-4
Ruiz-Ruiz S, Soler N, Sánchez-Navarro J, et al. (2013) Citrus tristeza virus p23: determinants for nucleolar localization and their influence on suppression of RNA silencing and pathogenesis. Molecular Plant Microbe Interactions 26(3): 306–318. doi: 10.1094/MPMI-08-12-0201-R
Saponari M, Manjunath K, Yokomi RK (2008) Quantitative detection of Citrus tristeza virus in citrus and aphids by real-time reverse transcription-PCR (TaqMan). Journal of Virological Methods 147(1): 43-53. doi: 10.1016/j.jviromet.2007.07.026
Saponari M, Zicca S, Loconsole G, et al. (2019) Detection of Citrus tristeza virus and Coinfecting Viroids. Methods Mol Biol. 2015: 67‐78. doi: 10.1007/978-1-4939-9558-5_6
Satyanarayana T, Gowda S, Mawassi M, et al. (2000) Closterovirus encoded HSP70 homolog and p61 in addition to both coat proteins function in efficient virion assembly. Virology 1278, 253–265. doi: 10.1006/viro.2000.0638
Satyanarayana T, Gowda S, Ayllon MA, et al. (2002) Mutational analysis of the replication signals in the 3′-nontranslated region of Citrus tristeza virus. Virology 286: 134–151. doi: 10.1006/viro.2002.1550
Satyanarayana T, Gowda S, Ayllón MA, et al. (2002) The p23 protein of Citrus tristeza virus controls asymmetrical RNA accumulation. J. Virol. 76: 473–483. doi: 10.1128/JVI.76.2.473-483.2002
Selvaraj V, Maheshwari Y, Hajeri S, Yokomi R (2019) A rapid detection tool for VT isolates of Citrus tristeza virus by immunocapture-reverse transcriptase loop-mediated isothermal amplification assay. PLoS ONE 14(9): e0222170. doi: 10.1371/journal.pone.0222170
Sharma S (2023) A Review On Citrus tristeza virus (CTV) And Its Management Approaches. Turkish Journal of Agriculture – Food Science and Technology 11(4): 803–810. doi: 10.24925/turjaf.v11i4.803-810.5273
Shilts T, El-Mohtar C, Dawson WO, Killiny N (2020) Citrus tristeza virus P33 Protein is Required for Efficient Transmission by the Aphid Aphis (Toxoptera) citricidus (Kirkaldy). Viruses 12(10): 1131. doi: 10.3390/v12101131
Soler N, Plomer M, Fagoaga C, et al. (2012) Transformation of Mexican lime with an intron-hairpin construct expressing untranslatable versions of the genes coding for the three silencing suppressors of Citrus tristeza virus confers complete resistance to the virus. Plant Biotechnology Journal 10: 597–608. doi: 10.1111/j.1467-7652.2012.00691.x
Sun Y-D, Folimonova SY (2021) Location matters: from changing a presumption about the Citrus tristeza virus tissue tropism to understanding the stem pitting disease. New Phytol. doi: 10.1111/nph.17777
Tatineni S, Robertson CJ, Garnsey SM, et al. (2008) Three genes of Citrus tristeza virus are dispensable for infection and movement throughout some varieties of citrus trees. Virology 376, 297–307. doi: 10.1016/j.virol.2007.12.038
Tatineni S, Gowda S, Dawson WO (2010) Heterologous minor coat proteins of Citrus tristeza virus strains affect encapsidation, but the coexpression of HLP70h and p61 restores encapsidation to wild-type levels. Virology 402: 262–270. doi: 10.1016/j.virol.2010.03.042
Tatineni S, Robertson CJ, Garnsey SM, Dawson WO (2011) A plant virus evolved by acquiring multiple nonconserved genes to extend its host range. Proc. Natl. Acad. Sci. U.S.A. 108, 17366–17371. doi: 10.1073/pnas.1113227108
Tatineni S, Dawson WO (2012) Enhancement or Attenuation of Disease by Deletion of Genes from Citrus Tristeza Virus. Journal of Virology 86(15): 7850-7857. doi: 10.1128/JVI.00916-12
Taylor EL, Gottwald TR, Adkins S (2023) Structural changes in Florida citrus production, 1980-2021 and associated consequences of weather events and disease. Journal of Citrus Pathology, 10. doi: 10.5070/
, (2000) Compendium of Citrus Diseases. St Paul, MN: APS Press.
Trinh J, Li T, Franco JY, et al. (2022) Citrus relatives exhibit natural variation in perception and response magnitude to microbial features. bioRxiv 2022.07.15.500235; doi: 10.1101/2022.07.15.500235
Wagemans J, Holtappels D, Vainio E, et al. (2022) Going Viral: Virus-Based Biological Control Agents for Plant Protection. Annu Rev Phytopathol. 60: 21-42. doi: 10.1146/annurev-phyto-021621-114208
William O. Dawson, Moshe Bar-Joseph, Stephen M. Garnsey, Pedro Moreno (2015) Citrus Tristeza Virus: Making an Ally from an Enemy. Annual Review of Phytopathology 53(1): 137-155. doi: 10.1146/annurev-phyto-080614-120012
Wu F, Huang M, Fox EGP, et al. (2021) Preliminary Report on the Acquisition, Persistence, and Potential Transmission of Citrus tristeza virus by Diaphorina citri. Insects 12(8): 735. doi: 10.3390/insects12080735
Xing-kai Z, Jin-fa Z, Ying W, et al. (2022) Molecular characterization of four citrus tristeza virus isolates that cause severe symptoms in navel orange in Ganzhou, China. Trop. plant pathol. 47: 509–520. doi: 10.1007/s40858-022-00505-0
Yokomi RK, Selvaraj V, Maheshwari Y, et al. (2017) Identification and Characterization of Citrus tristeza virus Isolates Breaking Resistance in Trifoliate Orange in California. Phytopathology 107(7): 901-908. doi: 10.1094/PHYTO-01-17-0007-R
(1996) Graft compatibility of Citrus with plants in the Aurantoidea and their susceptibility to citrus tristeza virus. Plant Dis. 80: 414–417. Link
Zeman, V. 1 930. Una enfennada nueva en los naranjales de Corrientes. Physis 19:410-411.