.
Condición fitosanitaria: Presente solo en algunas áreas del país
Grupo de cultivos: Oleaginosas
Especie hospedante: Soja (Glycine max)
Etiología: Bacteria. Gram negativa. Considerada hemibiotrófica
Agente causal: Xanthomonas axonopodis pv. glycines (Nakano) (Xag)
Taxonomía: Bacteria > Proteobacteria > Gammaproteobacteria > Xanthomonadales > Xanthomonadaceae > Xanthomonas
Propuesta de reclasificación:
Xanthomonas citri pv. glycines (Constantin et al., 2016)
.
.
Síntomas
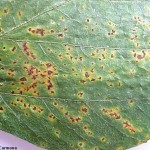
Al principio manifiesta en las hojas lesiones muy pequeñas poco evidentes, marrones a verde pálido, ligeramente elevadas, a veces con aspecto de ampolla, rodeadas con un halo clorótico delgado característico y distintivo. Si bien podrían confundirse con pústulas de roya, nunca tienen poro y además al realizarse una cámara húmeda presentan el signo característico de las bacteriosis (zooglea, de aspecto mucoso, brillante). Las lesiones pueden coalescer, al igual que en el tizón bacteriano, con desprendimiento de tejido foliar. También puede afectar vainas y semillas.
.
- Síntomas de la pustula bacteriana de la soja causada por Xanthomonas axonopodis pv. glycines. Autor: Daren Mueller, Iowa State University
- Síntomas de la pústula bacteriana de la soja en el envez de la hoja, causada por Xanthomonas axonopodis pv. glycines. Autor: A. Sisson.
- Autor: D. Pedersen, Univ. IL
.
Condiciones ambientales predisponentes para el establecimiento de la enfermedad
Temperaturas moderadas a elevadas, con óptimo de alrededor de 28ºC, alta humedad, lluvia y viento (dispersión).
.
Importancia relativa
Más difundida hacia el norte del país, al igual que para tizón bacteriano (Pseudomonas), se desconoce su incidencia en el rendimiento.
.
Manejo de la enfermedad
Utilizar semillas producidas en lotes sanos y/o certificada mediante análisis sanitario. Rotación de cultivos.
.
- Pustula Bacteriana causada por Xanthomonas spp. Autor: Direceu Gassen
- Pustula Bacteriana causada por Xanthomonas spp. Autor: Direceu Gassen
.
.
Bibliografía
Alvarez-Martinez CE, Sgro GG, Araujo GG, Paiva MRN, Matsuyama BY, Guzzo CR, Andrade MO, Farah CS (2020) Secrete or perish: The role of secretion systems in Xanthomonas biology. Computational and Structural Biotechnology Journal 19: 279-302. doi: 10.1016/j.csbj.2020.12.020
Athinuwat D, Prathuangwong S, Cursino L, Burr T (2009) Xanthomonas axonopodis pv. glycines soybean cultivar virulence specificity is determined by avrBs3 homolog avrXg1. Phytopathology 99(8): 996-1004. doi: 10.1094/PHYTO-99-8-0996
Carpenter SCD, Kladsuwan L, Han SW, Prathuangwong S, Bogdanove AJ (2019) Complete Genome Sequences of Xanthomonas axonopodis pv. glycines Isolates from the United States and Thailand Reveal Conserved Transcription Activator-Like Effectors. Genome Biology and Evolution 11(5): 1380-1384. doi: 10.1093/gbe/evz085
Catara V, Cubero J, Pothier JF, et al. (2021) Trends in Molecular Diagnosis and Diversity Studies for Phytosanitary Regulated Xanthomonas. Microorganisms 9(4): 862. doi: 10.3390/microorganisms9040862
Constantin EC, Cleenwerck I, Maes M, Baeyen S, Van Malderghem C, De Vos P, Cottyn B (2016) Genetic characterization of strains named as Xanthomonas axonopodis pv. dieffenbachiae leads to a taxonomic revision of the X. axonopodis species complex. Plant Pathology 65: 792-806. doi: 10.1111/ppa.12461
Costa J, Pothier JF, Boch J, et al. (2021) Integrating science on Xanthomonadaceae for sustainable plant disease management in Europe. Mol Plant Pathol. 22(12): 1461-1463. doi: 10.1111/mpp.13150
Domingo-Calap ML, Bernabéu-Gimeno M, Aure MC, et al. (2022) Comparative Analysis of Novel Lytic Phages for Biological Control of Phytopathogenic Xanthomonas spp. Microbiol Spectr. e0296022. doi: 10.1128/spectrum.02960-22
Kim JG, Park BK, Yoo CH, Jeon E, Oh J, Hwang I (2003) Characterization of the Xanthomonas axonopodis pv. glycines Hrp pathogenicity island. Journal of Bacteriology 185(10): 3155-66. doi: 10.1128/jb.185.10.3155-3166.2003
, , , , , , , The Arabidopsis immune receptor EFR increases resistance to the bacterial pathogens Xanthomonas and Xylella in transgenic sweet orange.
Nurcahyanti SD, Wahyuni WS, Masnilah R, Nurdika AAH (2021) Diversity of Bacillus spp. from soybean phyllosphere as potential antagonist agents for Xanthomonas axonopodis pv. glycines causal of pustule disease. Biodiversitas Journal of Biological Diversity 22. doi: 10.13057/biodiv/d221136
Ryan RP, Vorhölter FJ, Potnis N, et al. (2011) Pathogenomics of Xanthomonas: understanding bacterium-plant interactions. Nature Reviews Microbiology 9(5): 344–355. doi: 10.1038/nrmicro2558
Teper D, White FF, Wang N (2023) The Dynamic Transcription Activator-Like Effector Family of Xanthomonas. Phytopathology 113(4): 651-666. doi: 10.1094/PHYTO-10-22-0365-KD
Vieira PS, Bonfim IM, Araujo EA, et al. (2021) Xyloglucan processing machinery in Xanthomonas pathogens and its role in the transcriptional activation of virulence factors. Nat Commun 12: 4049. doi: 10.1038/s41467-021-24277-4
Violatti MR, Tebaldi ND (2016) Detecção de Xanthomonas axonopodis pv. glycines em sementes de soja. Summa Phytopathologica 42(3): 268-270. doi: 10.1590/0100-5405/2127
Zhang YJ, Pang YB, Wang XY, et al. (2022) Exogenous genistein enhances soybean resistance to Xanthomonas axonopodis pv. glycines. Pest Manag Sci. 78: 3664-3675. doi: 10.1002/ps.7009