.
Condición fitosanitaria: Plaga Cuarentenaria Ausente
Epidemiología: es considerada una enfermedad poliética
Grupo de cultivos: Frutales (Bayas)
Especie hospedante: Vid (Vitis vinifera)
Rango de hospedantes: amplio, no específico (*)
Transmisión: insectos vectores, que se alimentan del xilema
Etiología: Bacteria Gram negativa. Limitada al xilema. Bacteria fastidiosa (no cultivable).
Agente causal: Xylella fastidiosa subsp. fastidiosa Well et al., 1987
Taxonomía: Bacteria > Proteobacteria > Gammaproteobacteria > Xanthomonadales > Xanthomonadaceae > Xylella > Xylella fastidiosa
Xylella fastidiosa (Wells et al., 1987) es una bacteria gram negativa, limitada al xilema y el agente causal de la enfermedad de Pierce (EP) en V. vinifera. Esta bacteria se puede clasificar como un patógeno biotrófico y parásito obligado, ya que no mata el tejido del hospedante hasta etapas posteriores de su ciclo de vida. Esta especie es considerada una de las bacterias fitopatógenas más peligrosas del mundo.
(*) Xylella fastidiosa es una de las bacterias fitopatógenas con uno de los rangos de hospedantes más amplios , ya que puede infectar 343 especies de plantas, 163 géneros y 64 familias determinadas con dos métodos de detección diferentes, hasta 595 especies de plantas, 275 géneros y 85 familias, independientemente del método de detección aplicado (EFSA, 2020). Esta especie en su conjunto infecta a más de 560 especies de plantas en más de 260 géneros y 80 familias de plantas, incluidas monocotiledóneas, dicotiledóneas y gimnospermas (Delbianco et al., 2019), y en cientos de estas especies no causa síntomas y parece vivir como endófito. (Chatterjee et al., 2008).
.
.
Antecedentes
La enfermedad de Pierce fue descrita por primera vez en California, Estados Unidos, en 1892. Desde entonces, esta enfermedad ha sido identificada en viñedos localizados en la Costa del Golfo de Mexico y en el Atlántico Medio, incluyendo Virginia y Carolina del Norte, así como también en la parte sur del Medio Oeste hasta el norte de Oklahoma y Missouri.
.
Sintomatología
Las etapas iniciales de la infección son a menudo asintomáticas, por lo que existe un alto riesgo de contaminar nuevas áreas al importar plantas enfermas. El síntoma característico de la infección inicial es el quemado de las hojas, las cuales presentan escaldaduras, que se tornan de color café; los tejidos adyacentes cambian de color amarillo o rojo. Las áreas escaldadas llegan a ocupar hasta la mitad de las hojas. La escaldadura comienza en los márgenes y avanza de manera concéntrica hacia el punto de inserción del pecíolo. Los tallos de las plantas infectadas adquieren una apariencia marchita y la planta sufre defoliación prematura.
La rentabilidad de las plantas infectadas es improductiva, o producen pocos frutos de baja calidad, y los que llegan a formarse, se marchitan antes de la temporada de la cosecha. En general, el aspecto de las plantas enfermas se caracteriza por que las hojas presentan clorosis y decoloración, esta última inicia en los márgenes foliares, los cuales posteriormente se secan y como consecuencia se observan de color café.
Los síntomas foliares de la enfermedad de Pierce pueden confundirse con síntomas causados por estrés hídrico. Sin embargo, la banda de color amarillo o rojizo entre la zona verde(sana) y la zona seca (dañada) de las hojas solo se presenta en plantas infectadas por la enfermedad de Pierce.
.
- Autor: Olufemi J Alabi
- Autor: J Luis R
.
La progresión de la enfermedad varía ampliamente en función de las condiciones ambientales y los genotipos del patógeno y de los vástagos y portainjertos del hospedante, lo que contribuye colectivamente a diferentes microambientes químicos de la savia en los vástagos.
La proliferación exitosa de la bacteria y su movimiento a larga distancia a través de los elementos del xilema están mediados por factores de virulencia secretados, tales como la poligalacturonasa PglA (Roper et al., 2007), la lipasa / esterasa LesA (Nascimento et al., 2016) y la proteasa PrtA (Gouran et al., 2016), que pueden modificar la integridad del xilema por actividad enzimática; los cuales juntos contribuyen a la progresión de la enfermedad. Las oclusiones vasculares causadas por la formación de biofilms de la bacteria, engrosamiento de la pared celular del hospedante por deposición y lignificación de callosas, así como la formación de tilosas, contribuyen a la reducción del flujo de savia que conduce a la limitación de nutrientes y agua a medida que avanzan los síntomas (Chatterjee et al., 2008; Choi et al., 2013; Sun et al., 2013).
.
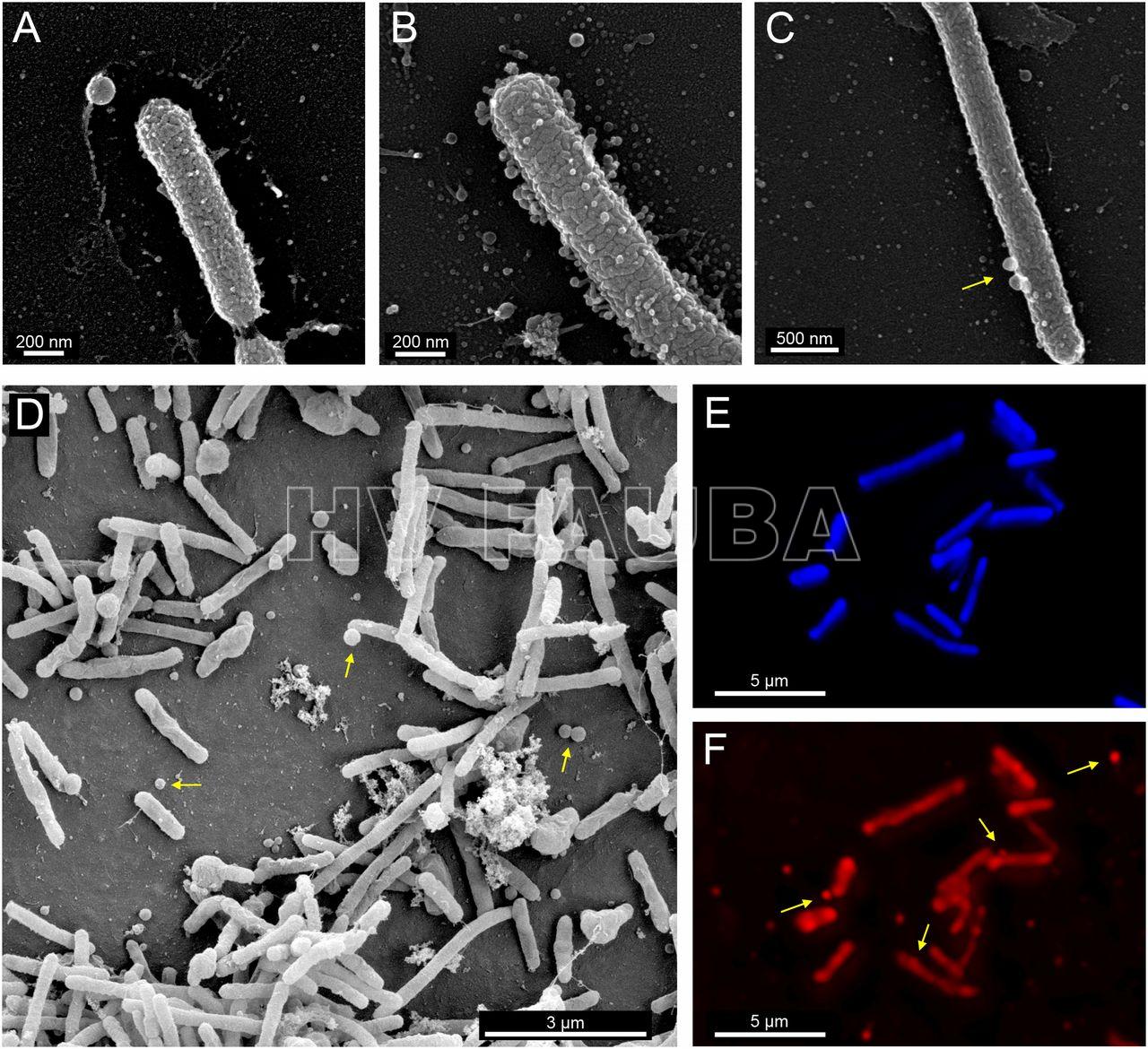
- Microscopía electrónica de barrido de Xylella fastidiosa (Ionescu et al., 2014)
- Células viables y no viables en vasos de xilema de uva colonizados por una cepa de X. fastidiosa marcada con gfp. En pecíolos de hojas que muestran síntomas muy severos de la enfermedad de Pierce se observa una alta incidencia de colonización de los vasos. En aquellos casos en que X. fastidiosa alcanza concentraciones muy altas y bloquea vasos (vasos 2 y 3), la mayoría de las células están muertas ya que se tiñen de rojo con yoduro de propidio, mientras que en aquellos vasos en los que el número de células X. fastidiosa es menor (vaso 1), la mayoría de las células permanecen vivas (células verdes). Autor: K. Newman, publicado en: Chatterjee et al., 2008.
.
De acuerdo con Chatterjee et al., 2008, aunque el número de vasos con colonias pequeñas era tres veces mayor en hojas sintomáticas que en hojas asintomáticas, el número de colonias ocluidas grandes (> 1000 células / colonia) era 20 veces mayor. Esto indica que la mayoría de las células en las hojas sintomáticas se encuentran en colonias grandes, mientras que esto no es cierto en las hojas asintomáticas. Este cambio a colonias más grandes en hojas sintomáticas también sugiere que las colonias grandes tienen un efecto más nocivo sobre el hospedante que las colonias pequeñas. Las colonias pequeñas en hojas asintomáticas aparentemente crecen y con el tiempo se transforman en colonias grandes. Sin embargo, las células también se deben dispersar a nuevos recipientes ya que las hojas sintomáticas también tienen mayor cantidad de vasos colonizados. Por lo tanto, el bloqueo del vaso por colonización bacteriana es aparentemente una variable acrítica en la expresión de los síntomas. Dado que el taponamiento de vasos es un requisito previo para la enfermedad, parece poco probable que las toxinas bacterianas o la falla vascular iniciada provoquen síntomas de la enfermedad. Aunque la mayor atención se ha dirigido al taponamiento de los vasos, la característica más obvia de la colonización del xilema por X. fastidiosa, solo recientemente se ha apreciado la extensa colonización de los vasos por pequeñas colonias de X. fastidiosa que no causan taponamiento. En promedio, solo alrededor del 10% al 15% de los vasos colonizados por X. fastidiosa están muy colonizados. Como las células de X. fastidiosa unidas a las paredes vasculares supuestamente obtienen al menos algunos de los nutrientes necesarios y otros recursos como el oxígeno de la savia del xilema que fluiría más allá de las células adheridas, se esperaría que si el flujo de savia del xilema en el vaso fuera restringido las células sufrirían. De hecho, los experimentos de Chatterjee et al., 2008 revelaron que la observación microscópica de la viabilidad de las células X. fastidiosa en el xilema de uva infectada, evaluada por tinción con yoduro de propidio, reveló que las colonias pequeñas de células X. fastidiosa contienen exclusivamente células vivas, mientras que las células en los vasos fuertemente colonizados están casi todas muertas.
.
Transmisión
La bacteria es transmitida naturalmente por los insectos vectores, que se alimentan del xilema de las plantas hospedantes (Chatterjee et al., 2008; Cornara et al., 2018).
La enfermedad puede ser transmitida por material vegetal de propagación infectado y por herramientas infestadas, así como por especies de insectos que se alimentan de savia del xilema y que transmiten el patógeno de una planta a otra.
La transmisión de este agente patógeno puede también ocurrir mecánicamente, aunque el riesgo de que esto ocurra es relativamente bajo.
La bacteria también puede transmitirse a través del uso de material vegetativo utilizado en la propagación de la vid, cuando este material proviene de plantas infectadas.
.
- Ejemplo de Chicharritas en la vid. Las formas adultas de las chicharritas son de color amarillo pálido, con marcas rojas en las alas. Miden cerca de 1/8 de pulgada de largo en forma de cuña.
- Ejemplos de chicharritas en la vid. Formas inmaduras son de color verde o blanco verdoso, y no poseen las marcas y las alas de los adultos. Estos insectos se vuelven activos en el momento en que las hojas de la planta están extendidas en un 50%
- Philaenus spumarius, uno de los posibles insectos vectores de la bacteria Xylella fastidiosa.
.
Diagnóstico y Detección
La detección e identificación de X. fastidiosa es difícil.
Las bacterias se pueden distribuir de manera irregular en los tejidos del hospedante, lo que dificulta la detección del patógeno (Baldi y La Porta, 2017).
Desde la primera mitad de los noventa, se han usado primers de PCR específicos para identificar X. fastidiosa de plantas infectadas.
Se pueden utilizar métodos inmunológicos, como el ensayo de inmunosorción ligada a enzimas (ELISA) (Chang et al., 1993; Leu et al., 1998).
Más recientemente, se ha descrito la detección de X. fastidiosa por tecnología de inmunofluorescencia (Carbajal et al., 2004; Buzkan et al., 2005).
Sin embargo, estos métodos no consideran variantes nuevas o emergentes debido a la recombinación genética del patógeno y las limitaciones de sensibilidad. Román-Reyna et al. (2021) desarrollaron un protocolo de metagenómica utilizando secuenciación interna de lectura corta como un enfoque complementario para una detección de X. fastidiosa asequible, rápida y altamente precisa.
Estas bacterias crecen lentamente (pueden necesitarse hasta 2 a 3 semanas para obtener colonias en medios de agar) y, en segundo lugar, muchos medios de cultivo comunes no son adecuados para cultivar cepas de X. fastidiosa. En cambio, deben usarse medios selectivos (PD2, PW, CS20) (Schaad et al., 2001). La identificación directa de bacterias solo es posible mediante microscopía de contraste de fase o campo oscuro, debido a las dimensiones de las células X. fastidiosa (radio de 0.2–0.4 μm y longitud de 0.9–3.5 μm) (Wells et al., 1987). Además, la identificación por microscopía requiere una formación profesional bastante específica para ser eficiente.
.
Epidemiología
X. fastidiosa sobrevive y se multiplica en los conductos que transportan el agua en la planta (xilema). Esta bacteria tiene una amplia gama de hospedantes, se conocen más de 563 especies de plantas hospedantes. Sin embargo, las cepas de X. fastidiosa muestran cierta especialización o preferencia por ciertos hospedantes. Por lo tanto, solo ciertas cepas específicas de la bacteria son capaces de infectar plantas de vid.
El proceso de diseminación de esta enfermedad inicia cuando las chicharritas (insectos vectores de la familia Cicadelidae), se alimentan de la savia de plantas enfermas y posteriormente transmiten la bacteria a plantas sanas al alimentarse de estas. Estos cicadélidos poseen aparato bucal picador-chupador y al alimentarse de la savia, los adultos y ninfas pueden adquirir la bacteria. La savia con la bacteria es absorbida y retenida en el intestino para finalmente ser transportada al esófago del insecto donde se multiplica y forma una cápsula de protección.Una vez que el insecto ha adquirido la bacteria, la puede trasmitir a un nuevo hospedante en un lapso de 1 a 2 horas, en ese momento la bacteria es inoculada directamente en el xilema y luego ocurre la infección sistemática en la planta sana. El adulto puede seguir trasmitiendo la bacteria durante toda su vida; sin embargo, las ninfas sólo pueden hacerlo hasta que cambian al siguiente estadio ninfal.
Desde el punto de vista epidemiológico, es considerada una enfermedad poliética.
.
Manejo Integrado
Si la enfermedad de Pierce es identificada en el viñedo, las plantas afectadas deben ser removidas y destruidas para limitar la propagación del patógeno a las plantas sanas. La Xylella fastidiosa normalmente se transmite de planta a planta por un insecto vector. Estos insectos vectores incluyen chicharritas, saltamontes, chapulines y langostinos.
El riesgo de propagación de la bacteria a través de las prácticas de poda en los meses de invierno es también considerado como bajo. El riesgo es mucho mayor cuando la poda se realiza durante los períodos en donde la planta de vid está creciendo. Independientemente del periodo en donde se realice la poda, es recomendable que el equipo de podado sea limpiado y desinfectado después de trabajar en cada una de las plantas de vid. Una solución al 10% de cloro es suficiente para matar la bacteria. Mantener varios juegos de tijeras de podar en una cubeta que contenga la solución con cloro es una buena práctica. Puede usarse una tijera mientras la(s) otra(s) se está(n) desinfectando. Cuando concluya el podado de una planta y se mueva hacia otra, simplemente cambie las tijeras, tomando la que ya está limpia y colocando en la cubeta la que acaba de usar. Esto no solo reducirá el riesgo de propagación de la X. fastidiosa, sino que también reducirá el riesgo de transmisión de otros agentes patógenos que pueden causar diversas enfermedades en las plantas de vid.
La propagación de esta enfermedad por el uso de materiales vegetativos contaminados representa muchos desafíos y problemas para los productores en áreas donde la enfermedad de Pierce es un problema. Muchos productores producen sus propios materiales de propagación cortando sus propios esquejes. Sin embargo, se sugiere que esta práctica se evite para reducir el riesgo de la propagación de la X. fastidiosa. Si el productor insiste en propagar su propio material vegetativo, las plantas madres deben ser probadas antes de usarlas, de manera que se esté seguro de que los materiales vegetativos que se obtendrán de esa planta están libres X. fastidiosa, y evitar que se propague la enfermedad. Los productores deben adquirir plantas en viveros certificados y resistir la tentación de de usar cortes o porta-injertos que provengan de viñedos no oficiales. La compra de material vegetativo en viveros confiables, reduce grandemente la posibilidad de introducir en su viñedo el patógeno X. fastidiosa.
Las diferentes variedades de uva responden de una manera diferente a la enfermedad de Pierce. En general se acepta que las variedades europeas de uva (Vitis vinifera) serán más susceptibles o intolerantes a la enfermedad de Pierce. La bacteria que causa la enfermedad de Pierce es nativa de Norte América, donde existen especies nativas de uva. Por lo tanto, las variedades de uva que no son nativas de Norte América tendrán poca o nula resistencia o tolerancia a esta enfermedad. Algunas variedades Americanas, o variedades que cuenta con una buena proporción de material genético serán más resistentes a esta enfermedad. Las variedades Vitis vinifera de origen como la ‘Chardonnay’ y la ‘Viognier’ son altamente susceptibles a la enfermedad de Pierce. Los productores que se vieron obligados a eliminar sus plantas de vides porque presentaron esta enfermedad de Pierce, deberán considerar plantar variedades resistentes o tolerantes a la enfermedad.
También es importante diseñar un programa de control de malezas en el viñedo. Hay evidencia circunstancial de que zonas reguladoras (buffer) de 3000 metros o más, libres de malezas alrededor de los viñedos, reduce sustancialmente la probabilidad de que el viñedo sea infectado. El mantener tiras libres de malezas, aplicando herbicidas y el uso de los llamados cultivos de cobertura, tales como pastos de invierno (Ryegrass anual, por ejemplo) entre las hileras podría reducir la propagación del patógeno a través de los insectos vectores.
En viñedos diagnosticados con la enfermedad de Pierce, podría ser necesaria la utilización de insecticidas sistémicos para reducir la propagación secundaria del patógeno entre las vides.
.
.
Bibliografía
Xylella fastidiosa in the EPPO region. Special Alert
Almeida RPP, Killiny N, Newman KL, Chatterjee S, Ionescu M, Lindow SE (2012) Contribution of rpfB to cell-cell signal synthesis, virulence, and vector transmission of Xylella fastidiosa. Molecular Plant-Microbe Interactions 25: 453-462. doi: 10.1094/MPMI-03-11-0074
Almeida RPP, Nunney L (2015) How Do Plant Diseases Caused by Xylella fastidiosa Emerge? Plant Disease 99(11): 1457-1467. doi: 10.1094/PDIS-02-15-0159-FE
Almeida RPP (2016). CHAPTER 12: Xylella fastidiosa Vector Transmission Biology. Vector-Mediated Transmission of Plant Pathogens. pp. 165-173. doi: 10.1094/9780890545355.012
Anas O, Harrison UJ, Brannen PM, Sutton TB (2008) The effect of warming winter temperatures on the severity of Pierce’s disease in the Appalachian mountains and piedmont of the southeastern United States. Plant Health Progress (online). doi: 10.1094/PHP-2008-0718-01-RS
Armijo G, Schlechter R, Agurto M, Muñoz D, Nuñez C and Arce-Johnson P (2016) Grapevine Pathogenic Microorganisms: Understanding Infection Strategies and Host Response Scenarios. Frontiers in Plant Science 7: 382. doi: 10.3389/fpls.2016.00382
Backus EA (2016) CHAPTER 13: Sharpshooter Feeding Behavior in Relation to Transmission of Xylella fastidiosa: A Model for Foregut-Borne Transmission Mechanisms. Vector-Mediated Transmission of Plant Pathogens. pp. 175-193. doi: 10.1094/9780890545355.013
Baldi P and La Porta N (2017) Xylella fastidiosa: Host Range and Advance in Molecular Identification Techniques. Frontiers in Plant Science 8: 944. doi: 10.3389/fpls.2017.00944
Burbank L, Sisterson MS, Wei W, et al. (2022) High growing season temperatures limit winter recovery of grapevines from Xylella fastidiosa infection – implications for epidemiology in hot climates. bioRxiv 2022.11.02.514902; doi: 10.1101/2022.11.02.514902
Buzkan N, Kocsis L, Walker MA (2005) Detection of Xylella fastidiosa from resistant and susceptible grapevine by tissue sectioning and membrane entrapment immunofluorescence. Microbiological Research 160: 225–231. doi: 10.1016/j.micres.2004.05.006
Carbajal D, Morano KA, Morano LD (2004) Indirect immunofluorescence microscopy for direct detection of Xylella fastidiosa in xylem sap. Current Microbiology 49: 372–375. doi: 10.1007/s00284-004-4369-5
Cavalieri V, Altamura G, Fumarola G, di Carolo M, Saponari M, Cornara D, Bosco D, Dongiovanni C (2019) Transmission of Xylella fastidiosa Subspecies Pauca Sequence Type 53 by Different Insect Species. Insects 10(10): 324. doi: 10.3390/insects10100324
Chang CJ, Garnier M, Zreik L, Rossetti V, Bove JM (1993) Culture and serological detection of the xylem-limited bacterium causing citrus variegated chlorosis and its identification as a strain of Xylella fastidiosa. Current Microbiology 27, 137–142. doi: 10.1007/BF01576010
Chatterjee S, Almeida RP, Lindow S (2008) Living in two worlds: the plant and insect lifestyles of Xylella fastidiosa. Annual Review of Phytopathology 46: 243-71. doi: 10.1146/annurev.phyto.45.062806.094342
Chen J, Wu F, Zheng Z, Deng X, Burbank LP, Stenger DC (2016) Draft Genome Sequence of Xylella fastidiosa subsp. fastidiosa Strain Stag’s Leap. Genome Announc. 4(2): e00240-16. doi: 10.1128/genomeA.00240-16
Choi MS, Kim W, Lee C, Oh CS (2013) Harpins, multifunctional proteins secreted by gram-negative plant-pathogenic bacteria. Molecular Plant Microbe Interactions 26: 1115–1122. doi: 10.1094/MPMI-02-13-0050-CR
Coletta-Filho HD, Francisco CS, Lopes JRS, Muller C, Almeida RPP (2017) Homologous recombination and Xylella fastidiosa host-pathogen associations in South America. Phytopathology 107: 305-312. doi: 10.1094/PHYTO-09-16-0321-R
Cornara D, Cavalieri V, Dongiovanni C, Altamura G, Palmisano F, Bosco D, Porcelli F, Almeida RPP, Saponari M (2017) Transmission of Xylella fastidiosa by naturally infected Philaenus spumarius (Hemiptera, Aphrophoridae) to different host plants. Journal of Applied Entomology 141: 80-87. doi: 10.1111/jen.12365
Cornara D, Sicard A, Zeilinger AR, Porcelli F, Purcell AH, Almeida RPP (2016) Transmission of Xylella fastidiosa to grapevine by the meadow spittlebug. Phytopathology 106: 1285-1290. doi: 10.1094/PHYTO-05-16-0202-R
Cornara, D., Bosco, D. & Fereres, A (2018) Philaenus spumarius: when an old acquaintance becomes a new threat to European agriculture. Journal of Pest Science 91, 957–972. doi: 10.1007/s10340-018-0966-0
Cunty A, Legendre B, de Jerphanion P, et al. (2020) Xylella fastidiosa subspecies and sequence types detected in Philaenus spumarius and in infected plants in France share the same locations. Plant Pathology 00: 1– 14. https://doi.org/10.1111/ppa.13248
Delbianco A, Czwienczek E, Pautasso M, Kozelska S, Monguidi M, Stancanelli G (2019) A new resource for research and risk analysis: the updated European Food Safety Authority database of Xylella spp. host plant species. Phytopathology 109(2):213–15. doi: 10.1094/PHYTO-09-18-0343-A
EFSA (European Food Safety Authority), 2020. Scientific report on the update of the Xylella spp. host plant database – systematic literature search up to 30 June 2019. EFSA Journal 2020;18(4):6114, 61 pp. https://doi.org/10.2903/j.efsa.2020.6114
, , , , , , et al (2021) Nanopore sequencing for the detection and identification of Xylella fastidiosa subspecies and sequence types from naturally infected plant material. Plant Pathology. doi: 10.1111/ppa.13416
Gambetta GA, Fei J, Rost TL, Matthews MA (2007) Leaf scorch symptoms are not correlated with bacterial populations during Pierce’s disease. Journal of Experimental Botany 58: 4037–4046. doi: 10.1093/jxb/erm260
Giampetruzzi A, Saponari M, Loconsole G, Boscia D, Savino VN, Almeida RPP, Zicca S, Landa BB, Chacón-Diaz C, Saldarelli P (2017) Genome-wide analysis provides evidence on the genetic relatedness of the emergent Xylella fastidiosa genotype in Italy to isolates from Central America. Phytopathology 107: 816-827. doi: 10.1094/PHYTO-12-16-0420-R
Gluck-Thaler E, et al (2020) Repeated gain and loss of a single gene modulates the evolution of vascular plant pathogen lifestyles. Science Advances: EABC4516. doi: 10.1126/sciadv.abc4516
, , , (2022) Forecasting future range shifts of Xylella fastidiosa under climate change. Plant Pathology, 00, 1– 10. doi: 10.1111/ppa.13637
Gouran H, Gillespie H, Nascimento R, Chakraborty S, Zaini PA, Jacobson A, et al. (2016) The secreted protease PrtA controls cell growth, biofilm formation and pathogenicity in Xylella fastidiosa. Scientific Reports 6: 31098. doi: 10.1038/srep31098
, , (2021) Seeing the forest for the trees: Use of phages to treat bacterial tree diseases. Plant Pathology 70: 1987– 2004. doi: 10.1111/ppa.13465
, , , et al. (2021) Xylella fastidiosa causes transcriptional shifts that precede tylose formation and starch depletion in xylem. Molecular Plant Pathology 22: 175– 188. doi: 10.1111/mpp.13016
Ingel B, Castro C, Burbank L, et al. (2023) Xylella fastidiosa Requires the Type II Secretion System for Pathogenicity and Survival in Grapevine. Mol Plant Microbe Interact. 36(10): 636-646. doi: 10.1094/MPMI-03-23-0027-R
Ionescu M, Zaini PA, Baccari C, Tran S, da Silva AM, Lindow SE (2014) Xylella fastidiosa outer membrane vesicles modulate plant colonization by blocking attachment to surfaces. Proceedings of the National Academy of Sciences 111 (37): E3910-E3918. doi: 10.1073/pnas.1414944111
Jeger M, Bragard C. (2019) The Epidemiology of Xylella fastidiosa; A Perspective on Current Knowledge and Framework to Investigate Plant Host-Vector-Pathogen Interactions. Phytopathology. 109(2): 200‐209. doi: 10.1094/PHYTO-07-18-0239-FI
Kandel PP, Almeida RPP, Cobine PA, De La Fuente L (2017) Natural competence rates are variable among Xylella fastidiosa strains and homologous recombination occurs in vitro between subspecies fastidiosa and multiplex. Molecular Plant-Microbe Interactions 30: 589-600. doi: 10.1094/MPMI-02-17-0053-R
Katam R, Chibanguza K, Latinwo LM, Smith D (2015) Proteome Biomarkers in Xylem Reveal Pierce’s Disease Tolerance in Grape. Journal of Proteomics and Bioinformatics 8(9): 217-224. doi: 10.4172/jpb.1000372
Killiny N, Hernandez-Martinez R, Dumenyo CK, Cooksey DA, Almeida RPP (2013) The exopolysaccharide of Xylella fastidiosa is essential for biofilm formation, plant virulence and vector transmission. Molecular Plant-Microbe Interactions 26: 1044-1053. doi: 10.1094/MPMI-09-12-0211-R
Labroussaa F, Ionescu M, Zeilinger AR, Lindow SE, Almeida RPP (2017) A chitinase is required for Xylella fastidiosa colonization of its insect and plant hosts. Microbiology 163: 502-509. doi: 10.1099/mic.0.000438
Leu HH, Leu LS, Lin CP (1998) Development and application of monoclonal antibodies against Xylella fastidiosa, the causal bacterium of pear leaf scorch. Journal of Phytopathology 146, 31–37. doi: 10.1111/j.1439-0434.1998.tb04747.x
, , , , , , , The Arabidopsis immune receptor EFR increases resistance to the bacterial pathogens Xanthomonas and Xylella in transgenic sweet orange.
Moll L, Baró A, Montesinos L, et al. (2022) Induction of Defense Responses and Protection of Almond Plants Against Xylella fastidiosa by Endotherapy with a Bifunctional Peptide. Phytopathology 112(9): 1907-1916. doi: 10.1094/PHYTO-12-21-0525-R
Moll L, Badosa E, De La Fuente L, et al. (2025) Mitigation of Almond Leaf Scorch by a Peptide that Inhibits the Motility of Xylella fastidiosa. Plant Dis. 109(2): 327-340. doi: 10.1094/PDIS-07-24-1414-RE
Nascimento R, Gouran H, Chakraborty S, Gillespie HW, Almeida-Souza HO, Tu A, Rao BJ, Feldstein PA, Bruening G, Goulart LR, Dandekar AM (2016) The Type II secreted Lipase/Esterase LesA is a key virulence factor required for Xylella fastidiosa pathogenesis in grapevines. Scientific Reports 6:18598. doi: 10.1038/srep18598
Newman KL, Almeida RPP, Purcell AH, Lindow SE (2003) Use of a green fluorescent strain for analysis of Xylella fastidiosa colonization of Vitis vinifera. Applied Environmental Microbiology 69: 7319–27. doi: 10.1128/AEM.69.12.7319-7327.2003
Nunney L, Schuenzel EL, Scally M, Bromley RE (2014) Stouthamer R. Large-scale intersubspecific recombination in the plant-pathogenic bacterium Xylella fastidiosa is associated with the host shift to mulberry. Appl Environ Microbiol. 80(10): 3025‐3033. doi: 10.1128/AEM.04112-13
O’Leary M, Arias-Giraldo LFF, Burbank L, et al. (2021) Complete genome resources for Xylella fastidiosa strains AlmaEM3 and BB08-1 reveal prophage-associated structural variation among blueberry-infecting strains. Phytopathology. doi: 10.1094/PHYTO-08-21-0317-A
Oliver JE, Sefick SA, Parker JK, et al. (2014) Ionome changes in Xylella fastidiosa-infected Nicotiana tabacum correlate with virulence and discriminate between subspecies of bacterial isolates. Mol Plant Microbe Interact. 27(10): 1048-58. doi: 10.1094/MPMI-05-14-0151-R
Pereira WEL, Ferreira CB, Caserta R, Melotto M, de Souza AA (2018) Xylella fastidiosa subsp. pauca and fastidiosa colonize Arabidopsis systemically and induce anthocyanin accumulation in infected leaves. Phytopathology (accepted). doi: 10.1094/PHYTO-05-18-0155-FI
Perilla-Henao LM and Casteel CL (2016) Vector-Borne Bacterial Plant Pathogens: Interactions with Hemipteran Insects and Plants. Frontiers in Plant Science 7: 1163. doi: 10.3389/fpls.2016.01163
Piqué N, Miñana-Galbis D, Merino S, Tomás JM (2015) Virulence factors of Erwinia amylovora: a review. Int. J. Mol. Sci. 16, 12836–12854. doi: 10.3390/ijms160612836
Rapicavoli JN et al. (2018) Lipopolysaccharide O-antigen delays plant innate immune recognition of Xylella fastidiosa. Nature Communications 9: 390. doi: 10.1038/s41467-018-02861-5
Rapicavoli J, Ingel B, Blanco-Ulate B, Cantu D, Roper C (2018) Xylella fastidiosa: an examination of a re-emerging plant pathogen. Molecular Plant Pathology 19(4): 786-800. doi: 10.1111/mpp.12585
Rashed A, Kwan J, Baraff B, Ling D, Daugherty MP, Killiny N, Almeida RPP (2013) Relative susceptibility of Vitis vinifera cultivars to vector-borne Xylella fastidiosa through time. PLoS ONE 8: e55326. doi: 10.1371/journal.pone.0055326
Roper MC, Greve LC, Warren JG, Labavitch JM, Kirkpatrick BC (2007) Xylella fastidiosa requires polygalacturonase for colonization and pathogenicity in Vitis vinifera grapevines. Molecular Plant Microbe Interactions 20: 411–419. doi: 10.1094/MPMI-20-4-0411
Schaad NW, Jones JB, Chun W (eds.) (2001) Laboratory Guide for the Identification of Plant Pathogenic Bacteria, 3rd Edn. St. Paul, MN: American Phytopathological Society Press.
Schaad NW, Postnikova E, Lacy G, Fatmi MB, Chang CJ (2004) Xylella fastidiosa subspecies: X. fastidiosa subsp piercei, subsp. nov., X. fastidiosa subsp. multiplex subsp. nov., and X. fastidiosa subsp. pauca subsp. nov. Systematic and Applied Microbiology 27: 763. doi: 10.1078/0723202042369848
Schaad NW, Postnikova E, Lacy G, Fatmi M, Chang CJ (2004) Xylella fastidiosa subspecies: X. fastidiosa subsp. [correction] fastidiosa [correction] subsp. nov., X. fastidiosa subsp. multiplex subsp. nov., and X. fastidiosa subsp. pauca subsp. nov.» Syst. Appl. Microbiol. (2004) 27:290-300. [Erratum: Syst. Appl. Microbiol. (2004) 27:763.]
, , , et al. (2021) Expression of putative effectors of different Xylella fastidiosa strains triggers cell death-like responses in various Nicotiana model plants. Molecular Plant Pathology 00: 1– 9. doi: 10.1111/mpp.13147
Sertedakis M, et al. (2021) Expression of putative effectors of different Xylella fastidiosa subspecies/strains reveals recognition and defense activation in various model plants. bioRxiv 2021.05.27.445625. doi: 10.1101/2021.05.27.445625
Sicard A, Zeilinger AR, Vanhove M, Schartel TE, Beal DJ, Daugherty MP, Almeida RPP (2018) Xylella fastidiosa: Insights into an Emerging Plant Pathogen. Annual Review of Phytopathology (56):181-202. doi: 10.1146/annurev-phyto-080417-045849
Sun Q, Sun Y, Walker MA, Labavitch JM (2013) Vascular occlusions in grapevines with Pierce’s disease make disease symptom development worse. Plant Physiology 161: 1529–1541. doi: 10.1104/pp.112.208157
Wallis CM, Wallingford AK, Chen J (2013) Grapevine rootstock effects on scion sap phenolic levels, resistance to Xylella fastidiosa infection, and progression of Pierce’s disease. Frontiers in Plant Science 4:502. doi: 10.3389/fpls.2013.00502
Wells JM, Raju BC, Hung HY, Weisburg WG, Mandelcopaul L, Brenner DJ (1987) Xylella fastidiosa gen-nov. sp-nov. – Gram negative, xylem-limited, fastidious plant bacteria related to Hanthomonas spp. Int. J. Syst. Bacteriol. 37: 136–143. doi: 10.1099/00207713-37-2-136
Wistrom C, Purcell AH (2005) The fate of Xylella fastidiosa in vineyard weeds and other alternate hosts in California. Plant Disease 89: 994–99. doi: 10.1094/PD-89-0994
Zaini PA, Nascimento R, Gouran H, Cantu D, Chakraborty S, Phu M, Goulart LR and Dandekar AM (2018) Molecular Profiling of Pierce’s Disease Outlines the Response Circuitry of Vitis vinifera to Xylella fastidiosa Infection. Frontiers in Plant Science 9: 771. doi: 10.3389/fpls.2018.00771