.
Condición fitosanitaria: Plaga no cuarentenaria reglamentada **
Grupo de cultivos: Hortícolas
Especie hospedante: Tomate (Solanum lycopersicum)
Rango de hospedantes: R. solanacearum tiene un rango de hospedantes inusualmente amplio, pudiendo infectar a más de 450 especies de plantas que pertenecen a más de 50 familias, que abarcan monocotiledóneas y dicotiledóneas y plantas herbáceas y leñosas (Norman et al., 2009; Cho et al., 2019).
Epidemiología: policíclica, subaguda
Etiología: Bacteria. Gram negativa
Agente causal: Ralstonia solanacearum (Smith 1896) Yabuuchi et al. 1996 emend. Safni et al. 2014
Taxonomía: Bacteria > Proteobacteria > Betaproteobacteria > Burkholderiales > Burkholderiaceae > Ralstonia
Ralstonia solanacearum es una bacteria Gram-negativa, con forma de bastón, estrictamente aeróbica, de 0.5-0.7 x 1.5-2.0 μm de tamaño. Es muy sensible a la desecación y se inhibe en cultivo por bajas concentraciones (2%) de cloruro de sodio (NaCl). Para la mayoría de las cepas, la temperatura óptima de crecimiento es de 27-32ºC, sin embargo, algunas cepas tienen una temperatura óptima inferior de 27ºC.
.
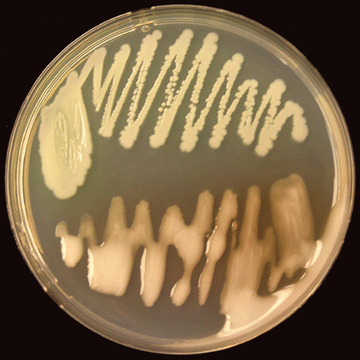
- 01 Colonias virulentas (inferior) y no virulentas (superior) de R. solanacearum en medio de crecimiento de agar CPG. Autor: P. Champoiseau, Universidad de Florida.
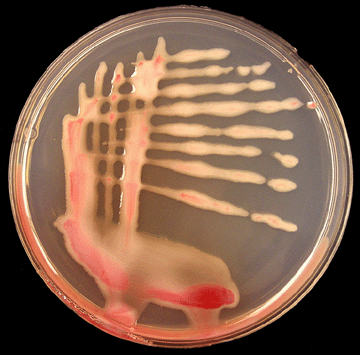
- 02 Colonias virulentas de R. solanacearum en medio de agar TZC. Autor: P. Champoiseau, Universidad de Florida.
.
** Condición fitosanitaria: Plaga Cuarentenaria Ausente para Ralstonia solanacearum raza 2
.
.
Antecedentes
El marchitamiento bacteriano causado por Ralstonia solanacearum (Smith 1896) es una de las enfermedades bacterianas de plantas más devastadoras en las regiones tropicales y subtropicales del mundo. R. solanacearum es una bacteria fitopatógena que infecta y obstruye el xilema, y es un habitante del suelo. Posee un amplio rango de hospedantes. R. solanacearum ganó importancia en el mundo debido a su naturaleza destructiva, amplio rango de hospedantes y distribución geográfica. Afecta a un amplio rango de cultivos económicamente importantes como el tomate, la papa, la berenjena, el pimiento y cultivos no solanáceos como el plátano y el maní. En Argentina es muy importante en tomate, en la zona de producción de tomate primicia de Corrientes, con temperaturas medias mas elevadas respecto a la zona de producción hortícola de Buenos Aires.
.
Síntomas
En las etapas iniciales de la enfermedad, los primeros síntomas visibles de marchitez bacteriana se observan generalmente en el follaje de las plantas. Estos síntomas consisten en la marchitez de las hojas más jóvenes en los extremos de las ramas durante las horas más calurosas del día. En esta etapa, solo uno o la mitad de un folíolo puede marchitarse, y las plantas pueden recuperarse por la noche, cuando las temperaturas son más frescas. A medida que la enfermedad se desarrolla en condiciones favorables, toda la planta puede marchitarse rápidamente y desecarse, aunque las hojas secas permanezcan verdes, lo que provoca un marchitamiento generalizado y un follaje amarillento y, finalmente, la muerte de la planta. Otro síntoma común que puede asociarse con la marchitez bacteriana en el campo es el retraso en el crecimiento de las plantas. Estos síntomas pueden aparecer en cualquier etapa del crecimiento de la planta, aunque en el campo es común que las plantas de aspecto saludable se marchiten repentinamente cuando las frutas se expanden rápidamente. En infecciones bien establecidas, secciones transversales de tallos pueden revelar decoloración marrón de tejidos infectados.
.
- Cultivo de tomate con incidencia de marchitez bacteriana por Ralstonia solanacearum, en un invernadero de La Plata, Bs. As., en diciembre. Autor: Ing. Claudio Baron
.
Las temperaturas elevadas (29-35ºC) favorecen la expresión de síntomas, los que pueden progresar rápidamente después de la infección. Sin embargo, en condiciones favorables, las plantas asintomáticas pueden permanecer infectadas de forma latente durante largos períodos de tiempo. Después de la infección, el patógeno puede sobrevivir y diseminarse desde la planta infectada.
El signo característico de la Marchitez Bacteriana del Tomate puede observarse en la superficie de secciones recién cortadas de tallos severamente infectados, como un exudado pegajoso de color blanco lechoso, que indica la presencia de masas densas de células bacterianas en haces vasculares infectados, y particularmente en el xilema.
.
- Autor: Ed Sikora
.
.
Diagnóstico del agente causal
La identificación precisa de R. solanacearum en plantas sintomáticas o asintomáticas y de muestras de agua o suelo exige múltiples métodos microbiológicos y moleculares. Se debe utilizar una batería de pruebas complementarias que difieran en su sensibilidad y/o especificidad para los análisis de campo o laboratorio para la identificación inequívoca de especies y biovar de bacterias.
Las pruebas de rutina pueden facilitar la detección temprana y la identificación de bacterias en plantas potencialmente infectadas o muestras de suelo y agua contaminadas por R. solanacearum. No pueden usarse para identificar la raza o biovar del organismo. Estas pruebas de detección incluyen transmisión por tallo, crecimiento en placas con medios semiselectivos (SMSA modificado), ensayos de inmunodiagnóstico con anticuerpos específicos de R. solanacerum, identificación basada en ácidos nucleicos utilizando primers específicos de R. solanacerum y evaluación de la patogenicidad utilizando hospedantes susceptibles (por ejemplo, plántulas de tomate). Varias pruebas de detección rápida, como por ejemplo Agdia ImmunoStrip, están disponibles comercialmente para la detección rápida a campo de R. solanacearum.
.
- Autor: Miryam Valenzuela
- Autor: Ed Sikora
.
.
A nivel de subespecies, la identificación de cepas de R. solanacearum puede evaluarse con varios métodos basados en ácidos nucleicos tales como la hibridación de la sonda de ADN y especialmente la amplificación de la reacción en cadena de la polimerasa (PCR) con sondas y primers específicos.
La determinación de la raza generalmente no es posible debido a que las cepas de R. solanacearum generalmente tienen numerosos hospedantes y no tienen especificidad raza-hospedante. Esta es la razón por la cual el sistema de subclasificación de razas ha caído en desuso, aunque todavía tiene un significado regulatorio debido a las reglas de cuarentena escritas para la «raza 3 biovar 2».
.
- 04 Síntomas de la marchitez bacteriana del tomate causada por R. solanacearum: secado del follaje y muerte de la planta. Autor: T.M. Momol, University of Florida
- Fuente: HortTechnology hortte 29, 3; 10.21273/HORTTECH04318-19
- 01 Síntomas de la marchitez bacteriana del tomate causada por R. solanacearum: marchitez del follaje y atrofia de la planta. Autor: C. Allen, University of Wisconsin
- Decoloración del sistema vascular. El marchitamiento y pudrición de la raíz de la corona por Fusarium y el marchitamiento por Verticillium pueden causar síntomas similares. La superficie del tallo puede oscurecerse y colapsarse como en el caso de necrosis de la médula. Autor: University of Florida
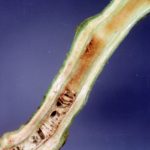
- 02 Síntomas de la Marchitez bacteriana del tomate (R. solanacearum) en xilema, corte transversal en tallo. Autor: Clemson University – USDA Cooperative Extension Slide Series
- 03 Decoloración gris-marrón de los tejidos vasculares (anillo vascular) y exudado bacteriano en el tubérculo de papa infectado por R. solanacearum. Autor: K. Tsuchiya, Japón.
- 05 Exudado bacteriano de la sección recién cortada de un tallo de geranio infectado por R. solanacearum. Autor: M. Daughtrey, Universidad de Cornell.
.
.
Ciclo de enfermedad y epidemiología
R. solanacearum es un patógeno capaz de sobrevivir en el suelo y en el agua, y dispersarse durante varios períodos de tiempo desde el suelo infestado o el agua, lo que puede formar una fuente reservorio de inóculo. La bacteria generalmente infecta a las plantas de tomate a través de las raíces (a través de heridas o en los puntos de emergencia de las raíces laterales). Los organismos «transmitidos» por el suelo, como el nematodo agallador, pueden causar daños a las raíces de las plantas y favorecer la penetración de la bacteria. La infección de las plantas también puede ocurrir a través de lesiones en el tallo causadas por prácticas culturales o daño de insectos. En algunos casos, la propagación planta a planta puede ocurrir cuando las bacterias se mueven desde las raíces de las plantas infectadas hasta las raíces de las plantas sanas cercanas, a menudo a través de prácticas de riego. Es decir, que una vez producidos los primeros focos de infección en el cultivo, la dispersión dentro del cultivo puede ocurrir entre plantas (aloinfección) por riego o labores culturales. Bajo estas condiciones, se considera que el patógeno causa epidemias policíclicas. No se conoce la propagación de la bacteria por medios aéreos y la subsiguiente contaminación de las plantas a través del follaje, por lo que R. solanacearum es un patógeno no dispersado por el aire.
Las altas temperaturas (29-35ºC) juegan un papel importante en el crecimiento de patógenos y el desarrollo de la enfermedad. Varios factores que pueden afectar la supervivencia del patógeno en el suelo y el agua también pueden favorecer el desarrollo de la enfermedad, incluyendo el tipo y estructura, contenido de humedad y materia orgánica del suelo, pH del agua y contenido de sal y la presencia de microorganismos antagonistas.
La bacteria también tiene una fase «exterior» (epífita) en la que puede residir en el exterior de la planta. Es de menor importancia en la epidemiología del patógeno ya que las bacterias no sobreviven epifíticamente durante largos períodos de tiempo cuando se exponen a condiciones de calor o cuando la humedad relativa es inferior al 95%. R. solanacearum puede sobrevivir durante días o años en material vegetal infectado en suelos, agua de irrigación superficial infestada y malezas infectadas. A partir de estas fuentes de inóculo, las bacterias pueden propagarse desde campos infestados a campos sanos mediante la transferencia de tierra en la maquinaria y el agua de escorrentía superficial después del riego o la lluvia. R. solanacearum también se puede propagar en estanques o ríos infestados y diseminarse a campos no infestados a través de cursos de agua. Las malezas semi-acuáticas infectadas también pueden jugar un papel importante en la diseminación del patógeno al liberar bacterias de las raíces en las aguas de riego.
En condiciones favorables, las plantas de tomate infectadas con R. solanacearum pueden no mostrar ningún síntoma de enfermedad. En este caso, las plantas infectadas de forma latente pueden jugar un papel importante en la propagación de la bacteria. En los casos en los que los sitios de producción de tomate con temperaturas más cálidas son una fuente importante de trasplantes de tomate para las regiones más frías, la marchitez bacteriana de los tomates se propaga ocasionalmente a través de plántulas infectadas. Sin embargo, el organismo no pasa el invierno en las zonas más frías. Las prácticas culturales en la producción a campo (alta densidad de plantas, uso de riego varias veces al día, recorte múltiple o socavación de plantas antes de la cosecha) o la producción en invernadero (riego por aspersión o manejo de plantas) pueden favorecer la infección y propagación del patógeno, y la diseminación a través de trasplantes de tomates infectados desde sitios de producción a sitios saludables.
A bajas temperaturas (<4ºC), las densidades de población bacteriana disminuyen rápidamente, pero las bacterias aún pueden sobrevivir, a menudo en un estado fisiológico latente. En hábitats naturales, R. solanacerum raza 3 biovar 2 puede sobrevivir durante el invierno en malezas semiacuáticas, en restos de plantas o en la rizófera de plantas no hospedantes que actúan como reservorios para el patógeno (Sebastià et al., 2021). Se demostró que las bacterias se liberan cada vez más de malezas semiacuáticas después del invierno cuando las temperaturas comienzan a aumentar.
.
- Autor: Ed Sikora
- Autor: Ed Sikora
.
Manejo Integrado
La marchitez bacteriana del tomate es difícil de controlar, y ninguna estrategia única aislada ha demostrado una eficacia del 100% en el control de la enfermedad hasta el momento.
Los bactericidas (cobre) y los antibióticos (estreptomicina, ampicilina, tetraciclina y penicilina) han demostrado poca eficacia en la supresión de R. solanacearum en el campo y son ambientalmente destructivos y bastante costosos para aplicar (antieconómico). Como resultado, se debe usar una combinación de diversos métodos de control, incluida la resistencia del hospedante, prácticas culturales y el uso de control químico o biológico en un enfoque integrado de manejo de la enfermedad para controlar la marchitez bacteriana del tomate en lugares donde se establece el patógeno.
* Si bien existe una gran dificultad de obtener una resistencia mundial a R. solanacearum, es posible lograr cierto nivel de control del marchitamiento bacteriano usando cultivares de tomate resistentes o moderadamente resistentes. Carmeille et al. (2006) plantearon la hipótesis de que los QTL en el cultivar Hawai 7996 pueden desplegar una resistencia filotipo específica.
* El control químico mediante fumigación del suelo (cloropicrina) o la aplicación de ácido fosforoso también es costoso. Se ha reportado que la fumigación del suelo tiene un éxito limitado si se combina con otros métodos de control. Cuando se usa, el control químico debe integrarse con otros métodos para reducir la presión de selección para la resistencia a patógenos.
* El control biológico, basado en el uso de antagonistas de R. solanacearum, y el uso de suelos supresivos ha mostrado resultados prometedores a pequeña escala experimental, pero aún necesita ser validado a mayor escala.
* El uso de Thymol, una sustancia química volátil derivada de plantas que aún no está disponible comercialmente, demostró reducir la incidencia de enfermedades y aumentar el rendimiento en experimentos de campo en Florida, USA. De manera similar, se demostró que la aplicación de Actigard (Syngenta), un inductor de resistencia de las plantas, en combinación con cultivares moderadamente resistentes mejora la resistencia contra la enfermedad a escala de campo en Florida.
La mejor estrategia para el control de la marchitez bacteriana a campo consiste principalmente en prácticas fitosanitarias y culturales. En las regiones donde la enfermedad es endémica, estos métodos han demostrado ser efectivos en algunas condiciones y deberían usarse:
Antes de la plantación:
* Realizar un control eficaz de las malezas en y alrededor de los campos de tomate y de las plantas acuáticas alrededor de los estanques de riego.
* Aplicar 3-4 años de rotación y cultivos de cobertura para los campos infestados para reducir el inóculo de R. solanacearum, malezas y nematodos.
* No regar durante la rotación los cultivos a partir de estanques o agua superficial contaminados con R. solanacearum, evitar la reinfestación.
* Usar campos bien drenados y nivelados y no usar áreas bajas.
* Aumentar el pH del suelo a 7.5-7.6 y aumentar el calcio disponible (encalado).
* Usar campos infestados (después de 3-4 años de rotación) durante los meses más fríos para la producción de tomate (Ej. temporada de primavera).
Durante la producción:
* Excluir el patógeno aplicando prácticas estrictas de saneamiento (agua de riego libre de patógenos, trasplantes, estacas, maquinaria, etc.).
* Adicionar cloro al agua de riego continuamente si se está usando agua superficial o agua de estanque infestada con R. solanacearum.
* Continuar con un control efectivo de malezas en y alrededor de los campos de tomate y estanques de riego.
* Irrigar en función de las necesidades de agua, evitar el exceso de riego.
* Aplicar inductores de resistencia vegetal en combinación con cultivares moderadamente resistentes.
* Se han explorado prácticas de control biológico con Trichoderma y Bacillus (Zhou et al., 2021)
Después de la cosecha:
* Arar enterrando los residuos de cosecha inmediatamente.
* Sembrar cultivos o cultivos de cobertura adecuados siguiendo la rotación para evitar las malezas que hospedan poblaciones de R. solanacearum.
Las prácticas de exclusión, como la cuarentena, las pruebas y la inspección visual del material importado de plantas hospedantes, la regulación y el establecimiento de protocolos mínimos de saneamiento para la producción pueden evitar la introducción del patógeno. Prácticas tales como la limpieza y desinfección de herramientas y maquinaria de campo y manejo, y el uso de buenas prácticas culturales sanitarias evitarán el movimiento del patógeno de campos infestados a campos libres de la enfermedad, evitando la introducción inadvertida del patógeno. En el invernadero, las prácticas sanitarias para la producción de trasplantes de tomate pueden incluir evitar la irrigación, la separación amplia de invernaderos de las áreas de producción de campo, la desinfección de todos los marcos, bandejas y herramientas, el uso de suelos libres de patógenos o mezcla de macetas, control de malezas, y manejo limitado de plantas.
.
.
- Autor: Ed Sikora
- Autor: Ed Sikora
.
.
VIDEOS
Patente Otri #17: Marchitez causada por la bacteria Ralstonia solanacearum. Universidad de Valencia.
.
.
Bibliografía
Abdurahman A, Parker ML, Kreuze J, Elphinstone JG, Struik PC, Kigundu A, Arengo E, Sharma K (2019) Molecular Epidemiology of Ralstonia solanacearum Species Complex Strains Causing Bacterial Wilt of Potato in Uganda. Phytopathology 109:1922-1931. https://doi.org/10.1094/PHYTO-12-18-0476-R
Beutler J, Holden S, Georgoulis SJ, et al. (2022) Whole genome sequencing suggests that “non-pathogenicity on banana (NPB)” is the ancestral state of the Ralstonia solanacearum IIB-4 lineage. PhytoFrontiers™. doi: 10.1094/PHYTOFR-06-22-0068-SC
Caldwell D, Kim BS, Iyer-Pascuzzi AS (2017) Ralstonia solanacearum Differentially Colonizes Roots of Resistant and Susceptible Tomato Plants. Phytopathology 107(5): 528-536. doi: 10.1094/PHYTO-09-16-0353-R
Carmeille A, Caranta C, Dintinger J, et al. (2006) Identification of QTLs for Ralstonia solanacearum race 3-phylotype II resistance in tomato. Theoretical and Applied Genetics 113: 110–121. doi: 10.1007/s00122-006-0277-3
Champoiseau PG, Jones JB, Allen C (2009) Ralstonia solanacearum race 3 biovar 2 causes tropical losses and temperate anxieties. Online. Plant Health Progress. doi: 10.1094/PHP-2009-0313-01-RV
Chandrashekara KN, Prasannakumar MK (2010) New host plants for Ralstonia solanacearum from India. Plant Pathology 59: 1164-1164. doi: 10.1111/j.1365-3059.2010.02358.x
, , , et al. (2024) The Ralstonia solanacearum effector RipV1 acts as a novel E3 ubiquitin ligase to suppress plant PAMP-triggered immunity responses and promote susceptibility in potato. Plant Pathology 00: 1–13. doi: 10.1111/ppa.13885
Chesneau T, Maignien G, Boyer C, Chéron J-J, Roux-Cuvelier M, Vanhuffel L, Poussier S and Prior P (2018) Sequevar Diversity and Virulence of Ralstonia solanacearum Phylotype I on Mayotte Island (Indian Ocean). Frontiers in Plant Science 8:2209. doi: 10.3389/fpls.2017.02209
Cho H, Song E-S, Heu S, Baek J, Lee YK, Lee S, Lee S-W, Park DS, Lee T-H, Kim J-G and Hwang I (2019) Prediction of Host-Specific Genes by Pan-Genome Analyses of the Korean Ralstonia solanacearum Species Complex. Front. Microbiol. 10: 506. doi: 10.3389/fmicb.2019.00506
Cao Y, Pi H, Chandrangsu P, Li Y, Wang Y, Zhou H, Xiong H, Helmann JD, Cai Y (2018) Antagonism of Two Plant-Growth Promoting Bacillus velezensis Isolates Against Ralstonia solanacearum and Fusarium oxysporum. Scientific Reports 8: 4360. doi: 10.1038/s41598-018-22782-z
Denny T (2007) Plant pathogenic Ralstonia species. In: Gnanamanickam S.S. (eds) Plant-Associated Bacteria. Springer, Dordrecht. doi: 10.1007/978-1-4020-4538-7_16
Eckshtain‐Levi N, Weisberg AJ, Vinatzer BA (2018) The population genetic test Tajima’s Didentifies genes encoding pathogen‐associated molecular patterns and other virulence‐related genes in Ralstonia solanacearum. Molecular Plant Pathology 19: 2187-2192. doi: 10.1111/mpp.12688
Elsayed TR, Jacquiod S, Nour EH, et al. (2020) Biocontrol of Bacterial Wilt Disease Through Complex Interaction Between Tomato Plant, Antagonists, the Indigenous Rhizosphere Microbiota, and Ralstonia solanacearum. Front. Microbiol. 10: 2835. doi: 10.3389/fmicb.2019.02835
Fan X, Zhao Z, Li Y, et al. (2018) The EF-Tu epitope elf26 of Ralstonia solanacearum can promote resistance to bacterial wilt disease in Nicotiana species. Canadian Journal of Plant Pathology. doi: 10.1080/07060661.2018.1483968
Fernandez E, Gutarra L, Kreuze J (2015) Evaluación del gen que codifica la enzima βHPMEH para la inhibición de la marchitez bacteriana causada por Ralstonia solanacearum. Rev. peru biol. 22(2): 193-198. Link
Flavier A, Clough S, Schell M, Denny T (1997) Identification of 3-hydroxypalmitic acid methyl ester as a novel autoregulator controlling virulence in Ralstonia solanacearum. Molecular Microbiology 26(2): 251-259. doi: 10.1046/j.1365-2958.1997.5661945.x
French ER, Anguiz R, Aley P (1998) The Usefulness of Potato Resistance to Ralstonia solanacearum, for the Integrated Control of Bacterial Wilt. In: Prior P, Allen C, Elphinstone J (eds) Bacterial Wilt Disease. Springer, Berlin, Heidelberg. doi: 10.1007/978-3-662-03592-4_58
He Y, Chen Y, Zhang Y, Qin X, Wei X, Zheng D, Lin W, Li Q, Yuan G (2021) Genetic diversity of Ralstonia solanacearum species complex strains obtained from Guangxi, China and their pathogenicity on plants in the Cucurbitaceae family and other botanical families. Plant Pathology. doi: 10.1111/ppa.13389
Hong JC, Norman DJ, Reed DL, Momol MT, Jones JB (2012) Diversity among Ralstonia solanacearum strains isolated from the southeastern United States. Phytopathology 102: 924-936. doi: 10.1094/PHYTO-12-11-0342
Hu J, Wei Z, Friman V-P, et al. (2016) Probiotic diversity enhances rhizosphere microbiome function and plant disease suppression. mBio 7: e01790-16. doi: 10.1128/mBio.01790-16
, , , et al. (2023) Positive regulation of the PhcB neighbouring regulator PrhX on expression of the type III secretion system and pathogenesis in Ralstonia solanacearum. Molecular Plant Pathology 00: 1–13. doi: https://doi.org/10.1111/mpp.13398
Huet G (2014) Breeding for resistances to Ralstonia solanacearum. Frontiers in Plant Science. 5: 715. doi: 10.3389/fpls.2014.00715
Huilan C, Bingsen W, He T, Xueao Z, Botao S (2020) Proteomic analysis of potato responding to the invasion of Ralstonia solanacearum UW551 and its type III secretion system mutant. Mol Plant Microbe Interact. doi: 10.1094/MPMI-06-20-0144-R
Janse JD, Araluppan FAX, Schans J, Wenneker M, Westerhuis W (1998) Experiences with Bacterial Brown Rot Ralstonia solanacearum Biovar 2, Race 3 in the Netherlands. In: Prior P., Allen C., Elphinstone J. (eds) Bacterial Wilt Disease. Springer, Berlin, Heidelberg. doi: 10.1007/978-3-662-03592-4_21
Ji P, Momol MT, Olson SM, Pradhanang PM (2005) Evaluation of thymol as biofumigant for control of bacterial wilt of tomato under field conditions. Plant Disease 89: 497-500. doi: 10.1094/PD-89-0497
, , , , (2020) Ralstonia solanacearum preferential colonization in the shoot apical meristem explains its pathogenicity pattern in tomato seedlings. Plant Pathology 69: 1347– 1356. doi: 10.1111/ppa.13220
Khan M, Khan AU, Bogdanchikova N, Garibo D (2021) Antibacterial and Antifungal Studies of Biosynthesized Silver Nanoparticles against Plant Parasitic Nematode Meloidogyne incognita, Plant Pathogens Ralstonia solanacearum and Fusarium oxysporum. Molecules 26(9): 2462. doi: 10.3390/molecules26092462
Kim SG, Hur O-S, Ro N-Y, et al. (2016) Evaluation of Resistance to Ralstonia solanacearum in Tomato Genetic Resources at Seedling Stage. The Plant Pathology Journal 32(1): 58-64. doi: 10.5423/PPJ.NT.06.2015.0121
Kraepiel Y, Barny MA (2016) Gram-negative phytopathogenic bacteria, all hemibiotrophs after all? Molecular Plant Pathology 17(3): 313-6. doi: 10.1111/mpp.12345. PMID: 26609783
Kwak MJ, et al. (2018) Rhizosphere microbiome structure alters to enable wilt resistance in tomato. Nature Biotechnology. doi: 10.1038/nbt.4232
Liu Q, Liu Y, Tang Y, Chen J and Ding W (2017) Overexpression of NtWRKY50 Increases Resistance to Ralstonia solanacearum and Alters Salicylic Acid and Jasmonic Acid Production in Tobacco. Front. Plant Sci. 8:1710. doi: 10.3389/fpls.2017.01710
Lowe-Power TM, Khokhani D, Allen C (2018) How Ralstonia solanacearum Exploits and Thrives in the Flowing Plant Xylem Environment. Trends in Microbiology (In Press). doi: 10.1016/j.tim.2018.06.002
Lu H, Lema AS, Planas-Marquès M, Alonso-Díaz A, Valls M, Coll NS (2018) Type III Secretion–Dependent and –Independent Phenotypes Caused by Ralstonia solanacearum in Arabidopsis Roots. Molecular Plant-Microbe Interactions 31(1): 175-184. doi: 10.1094/MPMI-05-17-0109-FI
MacIntyre AM, Meline V, Gorman Z, et al. (2021) Trehalose induces tomato defenses and increases drought tolerance and bacterial wilt disease resistance. bioRxiv 2021.07.26.453814; doi: 10.1101/2021.07.26.453814
Messiha NAS, van Diepeningen AD, Farag NS, et al. (2007) Stenotrophomonas maltophilia: a new potential biocontrol agent of Ralstonia solanacearum, causal agent of potato brown rot. European Journal of Plant Pathology 118: 211–225. doi: 10.1007/s10658-007-9136-6
Mizubuti ESG, Yamada JK, Santiago TR, et al. (2022) On the assessment of the sources of inoculum of bacterial wilt in Brazil. Trop. plant pathol. doi: 10.1007/s40858-022-00515-y
Moon H, Pandey A, Yoon H, et al. (2021) Identification of RipAZ1 as an avirulence determinant of Ralstonia solanacearum in Solanum americanum. Molecular Plant Pathology 22: 317– 333. doi: 10.1111/mpp.13030
Mori Y, Ishikawa S, Ohnishi H, Shimatani M, Morikawa Y, Hayashi K, Ohnishi K, Kiba A, Kai K, Hikichi Y (2018) Involvement of ralfuranones in the quorum sensing signalling pathway and virulence of Ralstonia solanacearum strain OE1‐1. Molecular Plant Pathology 19: 454-463. doi: 10.1111/mpp.12537
Mukhtar MS, Deslandes L, Auriac M, et al. (2008) The Arabidopsis transcription factor WRKY27 influences wilt disease symptom development caused by Ralstonia solanacearum. The Plant Journal 56: 935-947. doi: 10.1111/j.1365-313X.2008.03651.x
Nakano M, Mukaihara T (2018) Ralstonia solanacearum Type III Effector RipAL Targets Chloroplasts and Induces Jasmonic Acid Production to Suppress Salicylic Acid-Mediated Defense Responses in Plants. Plant Cell Physiol. 59(12): 2576-2589. doi: 10.1093/pcp/pcy177
Naranjo FE, Yglesia LA, García A, Martínez ZY (2015) Diversidad genética de aislados de Ralstonia solanacearum de Cuba, mediante amplificación de las regiones repetitivas del genoma (REP-PCR). Rev. Protección Veg. 30(1): 52-59. Link
Norman DJ, Zapata M, Gabriel DW, et al. (2009) Genetic diversity and host range variation of Ralstonia solanacearum strains entering North America. Phytopathology 99(9): 1070-7. doi: 10.1094/PHYTO-99-9-1070
Obregon VG (2009) Caracterización de la variabilidad poblacional de Ralstonia solanacearum en cultivos de solanáceas del Noreste Argentino. Tesis de Maestría, Universidad Nacional del Litoral. Facultad de Ciencias Agrarias; Argentina. Link
Pradhanang PM, Ji P, Momol MT, Olson SM, Mayfield JL, Jones JB (2005) Application of acibenzolar-S-methyl enhances host resistance in tomato against Ralstonia solanacearum. Plant Disease 89: 989-993. doi: 10.1094/PD-89-0989
Prieto Romo J, Morales Osorio JG, Salazar Yepes M (2012) Identification of new hosts for Ralstonia solanacearum (Smith) race 2 from Colombia. Revista de Protección Vegetal 27(3): 151-161. Link
Qi P, Huang M, Hu X, et al. (2022) Ralstonia solanacearum effector targets TGA transcription factors to subvert salicylic acid signaling, The Plant Cell koac015. doi: 10.1093/plcell/koac015
Qiu A, Lei Y, Yang S, et al. (2018) CaC3H14 encoding a tandem CCCH zinc finger protein is directly targeted by CaWRKY40 and positively regulates the response of pepper to inoculation by Ralstonia solanacearum. Molecular Plant Pathology 19: 2221-2235. doi: 10.1111/mpp.12694
Ravelomanantsoa S, Vernière C, Rieux A, et al. (2018) Molecular Epidemiology of Bacterial Wilt in the Madagascar Highlands Caused by Andean (Phylotype IIB-1) and African (Phylotype III) Brown Rot Strains of the Ralstonia solanacearum Species Complex. Frontiers in Plant Science 8: 2258. doi: 10.3389/fpls.2017.02258
Raza W, Ling N, Yang L, Huang Q, Shen Q (2016) Response of tomato wilt pathogen Ralstonia solanacearum to the volatile organic compounds produced by a biocontrol strain Bacillus amyloliquefaciens SQR-9. Scientific Reports 6: 24856. doi: 10.1038/srep24856
Robertson AE Fortnum BA, Wood TC, Kluepfel DA (2001) Diversity of Ralstonia solanacearum in the Southeastern United States. Contributions to Tobacco & Nicotine Research 19: 323-331. doi: 10.2478/cttr-2013-0719
Schandry N (2017) A Practical Guide to Visualization and Statistical Analysis of R. solanacearum Infection Data Using R. Front. Plant Sci. 8:623. doi: 10.3389/fpls.2017.00623
Sebastià P, de Pedro-Jové R, Daubech B, et al. (2021) The Bacterial Wilt Reservoir Host Solanum dulcamara Shows Resistance to Ralstonia solanacearum Infection. Front. Plant Sci. 12: 755708. doi: 10.3389/fpls.2021.755708
, , , et al. (2020) The putative sensor histidine kinase PhcK is required for the full expression of phcA encoding the global transcriptional regulator to drive the quorum‐sensing circuit of Ralstonia solanacearum strain OE1‐1. Molecular Plant Pathology 21: 1591– 1605. doi: doi.org/10.1111/mpp.12998
She X, Yu L, Lan G, Tang Y and He Z (2017) Identification and Genetic Characterization of Ralstonia solanacearum Species Complex Isolates from Cucurbita maxima in China. Front. Plant Sci. 8: 1794. doi: 10.3389/fpls.2017.01794
Shen L, Yang S, Guan D, He S (2020) CaCML13 Acts Positively in Pepper Immunity Against Ralstonia solanacearum Infection Forming Feedback Loop with CabZIP63. International Journal of Molecular Sciences 21(11): 4186. doi: 10.3390/ijms21114186
Shi JL, Zai WS, Xiong ZL (2021) NB-LRR genes: characteristics in three Solanum species and transcriptional response to Ralstonia solanacearum in tomato. Planta 254(5): 96. doi: 10.1007/s00425-021-03745-7
, , , et al. (2023) Exploring the competitive potential of Ralstonia pseudosolanacearum and Ralstonia solanacearum: Insights from a comparative adaptability study. Plant Pathology 00: 1–17. doi: 10.1111/ppa.13848
Spraker JE, Sanchez LM, Lowe TM, Dorrestein PC, Keller NP (2016) Ralstonia solanacearum lipopeptide induces chlamydospore development in fungi and facilitates bacterial entry into fungal tissues. The ISME Journal 10: 2317–2330. doi: 10.1038/ismej.2016.32
Su L, Mo X, Sun J, et al. (2021) Potassium Phosphite Modulated the Soil Microbiome and Enriched the Antagonistic Bacteria Streptomyces coelicoflavus and Paenibacillus favisporus to Inhibit the Tomato Pathogen Ralstonia Solanacearum Synergistically. Research Square. doi: 10.21203/rs.3.rs-143372/v1
Su L, Feng H, Mo X, et al. (2022) Potassium phosphite enhanced the suppressive capacity of the soil microbiome against the tomato pathogen Ralstonia solanacearum. Biol Fertil Soils 58: 553–563. doi: 10.1007/s00374-022-01634-z
Suresh P, Varathraju G, Shanmugaiah V, et al. (2021) Partial purification and characterization of 2, 4-diacetylphloroglucinol producing Pseudomonas fluorescens VSMKU3054 against bacterial wilt disease of tomato. Saudi Journal of Biological Sciences 28: 2155-2167. doi: 10.1016/j.sjbs.2021.02.073
Swanson JK, Yao J, Tans-Kersten J, Allen C (2005) Behavior of Ralstonia solanacearum Race 3 Biovar 2 During Latent and Active Infection of Geranium. Phytopathology 95(2): 136-143. doi: 10.1094/PHYTO-95-0136
Tahir HAS, Gu Q, Wu H, Raza W, Safdar A, Huang Z, Rajer FU, Gao X (2017) Effect of volatile compounds produced by Ralstonia solanacearum on plant growth promoting and systemic resistance inducing potential of Bacillus volátiles. BMC Plant Biology. doi: 10.1186/s12870-017-1083-6
, , , et al. (2023) The transcription regulator ChpA affects the global transcriptome including quorum sensing-dependent genes in Ralstonia pseudosolanacearum strain OE1-1. Molecular Plant Pathology 24: 1370–1384. doi: 10.1111/mpp.13374
Tancos MA, Lowe‐Power TM, Peritore‐Galve FC, Tran TM, Allen C, Smart CD (2018) Plant‐like bacterial expansins play contrasting roles in two tomato vascular pathogens. Molecular Plant Pathology 19: 1210-1221. doi: 10.1111/mpp.12611
Tariq A, Sachin R (2020) Organic Management of Bacterial Wilt of Tomato and Potato Caused by Ralstonia solanacearum. Link
Truchon AN, Dalsing BL, Khokhani D, et al. (2022) Plant pathogenic Ralstonia phylotypes evolved divergent respiratory strategies and behaviors to thrive in xylem. bioRxiv 2022.11.09.515861; doi: 10.1101/2022.11.09.515861
Tjou-Tam-Sin NNA, van de Bilt JLJ, Westenberg M, Gorkink-Smits PPMA, Landman NM and Bergsma-Vlami M (2017) Assessing the Pathogenic Ability of Ralstonia pseudosolanacearum (Ralstonia solanacearum Phylotype I) from Ornamental Rosa spp. Plants. Frontiers in Plant Science 8: 1895. doi: 10.3389/fpls.2017.01895
Vanitha, S.C., Niranjana, S.R., Mortensen, C.N. et al. (2009) Bacterial wilt of tomato in Karnataka and its management by Pseudomonas fluorescens . BioControl 54: 685–695. doi: 10.1007/s10526-009-9217-x
, , , Pathogenic and genetic variability of Ralstonia solanacearum strains from the Philippines. Plant Pathology 00: 1– 11. doi: 10.1111/ppa.13304
, , , et al. (2023) A bacterial type III effector targets plant vesicle-associated membrane proteins. Molecular Plant Pathology 00: 1– 14. doi: 10.1111/mpp.13360
Wang Z, Luo W, Cheng S, et al. (2023) Ralstonia solanacearum – A soil borne hidden enemy of plants: Research development in management strategies, their action mechanism and challenges. Front. Plant Sci. 14: 1141902. doi: 10.3389/fpls.2023.1141902
Wu D, Schandry N, Lahaye T (2018) A modular toolbox for Golden‐Gate‐based plasmid assembly streamlines the generation of Ralstonia solanacearum species complex knockout strains and multi‐cassette complementation constructs. Molecular Plant Pathology 19: 1511-1522. doi: 10.1111/mpp.12632
Xia Y, Zou R, Escouboué M, et al. (2021) Secondary-structure switch regulates the substrate binding of a YopJ family acetyltransferase. Nat Commun 12: 5969. doi: 10.1038/s41467-021-26183-1
Yahiaoui N, Chéron J-J, Ravelomanantsoa S, Hamza AA, Petrousse B, Jeetah R, Jaufeerally-Fakim Y, Félicité J, Fillâtre J, Hostachy B, Guérin F, Cellier G, Prior P and Poussier S (2017) Genetic Diversity of the Ralstonia solanacearum Species Complex in the Southwest Indian Ocean Islands. Frontiers in Plant Science 8: 2139. doi: 10.3389/fpls.2017.02139
Zeiss DR, Mhlongo MI, Tugizimana F, Steenkamp PA, Dubery IA (2018) Comparative Metabolic Phenotyping of Tomato (Solanum lycopersicum) for the Identification of Metabolic Signatures in Cultivars Differing in Resistance to Ralstonia solanacearum. International Journal of Molecular Sciences 19: 2558. doi: 10.3390/ijms19092558
Zhang Y, Li J, Zhang W, et al. (2018) A putative LysR‐type transcriptional regulator PrhO positively regulates the type III secretion system and contributes to the virulence of Ralstonia solanacearum. Molecular Plant Pathology 19: 1808-1819. doi: 10.1111/mpp.12660
Zhang Y, Guo S, Zhang F, et al. (2023) CaREM1.4 interacts with CaRIN4 to regulate Ralstonia solanacearum tolerance by triggering cell death in pepper. Horticulture Research: uhad053. doi: 10.1093/hr/uhad053
Zheng X, Zhu Y, Liu B, Zhou Y, Che J, Lin N (2014) Relationship Between Ralstonia solanacearum Diversity and Severity of Bacterial Wilt Disease in Tomato Fields in China. Journal of Phytopathology 162: 607-616. doi: 10.1111/jph.12234
Zhou Y, Yang L, Wang J, et al. (2021) Synergistic Effect between Trichoderma virens and Bacillus velezensis on the Control of Tomato Bacterial Wilt Disease. Horticulturae 7(11): 439. doi: 10.3390/horticulturae7110439
Zhou Y, Zhang Z, Bao Z, et al. (2022) Graph pangenome captures missing heritability and empowers tomato breeding. Nature. doi: 10.1038/s41586-022-04808-9