.
Condición fitosanitaria: Presente / Presente
Grupo de cultivos: Leñosas (Frutales, Forestales, Ornamentales)
Rango de especies hospedantes: +600 especies. Agrobacterium spp. es capaz de infectar principalmente plantas dicotiledóneas (Winans, 1992).
Epidemiología: monocíclica*, subaguda. (*) excepto que durante la misma estación de crecimiento las labores o el riego dispersen el inóculo a partir de un arbol infectado
Etiología: Bacteria. Gram negativa. Considerada biotrófica (*)
Agente causal:
Agrobacterium tumefaciens (Smith and Townsend 1907) Conn 1942 (Approved Lists 1980) nom. approb. in [Skerman VBD et al. (1980)]
Syn Agrobacterium radiobacter = Rhizobium radiobacter (Beijerinck and van Delden 1902) Pribram 1933, effective name
.
Agrobacterium rhizogenes (Alippi et al., 2010)
Agrobacterium rubi (Alippi et al., 2010) (arándanos, frambuesos, moreras)
Agrobacterium vitis (Vitis spp)
Agrobacterium larrymoorei (Ficus benjamina)
.
Taxonomía: Bacteria > Proteobacteria > Alphaproteobacteria > Rhizobiales > Rhizobiaceae > Rhizobium/Agrobacterium group > Agrobacterium > Agrobacterium tumefaciens complex
.
Las especies de Agrobacterium se clasificaron por primera vez mediante una taxonomía basada en la patogenicidad de las cepas A. tumefaciens que inducen la agalla de la corona, las cepas de A. rhizogenes que inducen raíces pilosas y cepas no patógenas de A. radiobacter (Moore et al., 2001). Además, se han descrito otras tres especies: A. vitis, A. rubi y A. larrymoorei. Estas especies inducen agallas en Vitis spp.; arándanos, frambuesos, moreras; y Ficus benjamina, respectivamente (Bouzar y Jones, 2001; Moore et al., 2001; Young et al., 2005; Alippi et al., 2010).
Sin embargo, Young y colaboradores (2001), basados en el análisis filogenético del 16SrDNA, sugirieron que las especies de Agrobacterium deberían ser renombradas como Rhizobium radiobacter, Rhizobium rhizogenes, Rhizobium rubi y Rhizobium vitis. Además, estos investigadores describieron recientemente el aislamiento de Rhizobium skierniewicense de tumores de crisantemo y Prunus spp. (Pulawska et al., 2012) y Rhizobium pusense de la rizosfera del garbanzo (Panday et al., 2011). Sin embargo, otros autores como Farrand y colaboradores (2003) encontraron que, como grupo definido y distinto dentro de las rizobiaceas, el nombre debería mantenerse como Agrobacterium.
En cualquier caso, la clasificación filogenética de Agrobacterium sugiere que se pueden separar en: Agrobacterium biovar 1, que contiene al menos nueve especies genómicas que causan agallas de la corona o raíces pilosas o no son patógenas (Costechareyre et al., 2010; Lasalle et al., 2011); y tres especies estrechamente relacionadas, A. rubi que porta un plásmido Ti e induce agallas principalmente en Rubus spp. (Anderson y Moore, 1979), A. vitis (antes conocida como biovar 3), cuyas cepas que tienen un plásmido Ti provocan agallas exclusivamente en la vid (Lim et al., 2009), y A. larrymoorei, que porta un plásmido Ti y que produce agallas en Ficus benjamina (Bouzar y Jones, 2001). Sin embargo, los ex miembros de Agrobacterium biovar 2 que también inducen agallas o raíces pilosas, ya sea que tengan el plásmido Ti o Ri o no sean patógenos, se consideran Rhizobium rhizogenes (Portier et al., 2006). El Subcomité de Taxonomía de Agrobacterium y Rhizobium confirmó que Rhizobium rhizogenes es una designación válida para la biovariedad 2 (Costechareyre et al., 2010) y también para las cepas de A. radiobacter K84 y A. tumefaciens AK10 (Velázquez et al., 2010).
.
Biotipos:
Pulawska y colaboradores (2006), basándose en el análisis filogenético de las secuencias del gen del ARNr 23S, hicieron una clasificación más simple de Agrobacterium en cuatro taxones: Agrobacterium biovar 1, Agrobacterium biovar 2, Agrobacterium vitis (biovar 3) y A. rubi . Por otro lado, A. larrymoorei se dejó como una especie separada.
.
Agrobacterium biovar 1 (Syn = A. tumefaciens) = Rhizobium radiobacter, portador de plásmidos Ti o Ri o no patógenos
Agrobacterium biovar 2 (Syn = A. rhizogenes) = Rhizobium rhizogenes, portador de plásmidos Ti o Ri o no patógenos
Agrobacterium vitis (biovar 3)
Agrobacterium rubi
.
Desde entonces, la nomenclatura y taxonomía de Agrobacterium sigue siendo oscura y permanece en debate. El Subcomité de taxonomía de Agrobacterium y Rhizobium sugirió que corresponde a los autores elegir la nomenclatura que se utilizará (Portier et al., 2006).
Una amplia gama de cepas de Agrobacterium pueden compartir el mismo entorno de suelo donde es más probable que se produzca la transferencia horizontal de ADN (Bouzar et al., 1993; Kechris et al., 2006). Teniendo esto en cuenta, en un mismo suelo podrían estar presentes elevados niveles de diversidad entre aislamientos de una misma especie y/o entre diferentes especies. Esta diversidad podría reflejarse, entre otras cosas, en la virulencia de las cepas: su capacidad para infectar y provocar enfermedades en las plantas (Ryder et al., 1985; Nesme et al., 1987). Sin embargo, de acuerdo con las interacciones hospedante/patógeno, las plantas también pueden diferir en su susceptibilidad a Agrobacterium (Gelvin, 2010), es decir, algunas variedades de uvas, arándanos y frambuesas son particularmente susceptibles a diferentes especies o biovariedades de Agrobacterium (Benjama et al., 2002).
.
A. tumefasciens es una bacteria en forma de bacilo, flagelada, habitante del suelo, que posee un amplio rango de hospedantes, capaz de afectar a más de ochenta familias de plantas herbáceas y forestales. Esta bacteria puede infectar más de 600 especies de plantas susceptibles (Thomashow et al., 1980). Algunos de los cultivos de importancia económica en que se presenta la enfermedad inlcuyen: eucalyptus, olivo, vid, mango, frutales de carozo, frutales de pepita, rosa.
(*) actualmente hay controversia sobre si se la debe considerar una bacteria biotrófica o hemibiótrofa. Agrobacterium, podría considerarse un verdadero biótrofo porque no mata a las células de la planta hospedante, sino que induce el desarrollo de tumores vegetales para que proliferen y produzcan metabolitos que puede catabolizar (Kraepiel y Barny, 2016). Sorprendentemente, algunos rasgos de la relación de parasitismo se parecen a los expresados por simbiontes en bacterias de fijación de N estrechamente relacionadas de la familia Rhizobiaceae revelando comportamientos similares entre simbiontes y biótrofos patógenos (González‐Mula et al., 2018).
Las especies de Agrobacterium son particulares porque infectan a sus hospedantes solo a través de heridas que liberan compuestos como la acetosiringona, que activan el mecanismo complejo y único codificado por los plásmidos. Esta compleja interacción da como resultado la inserción de una secuencia de Ti o Ri en el genoma de la planta en el núcleo celular (Gelvin, 2000).
.
- Imagen de microscopio electrónico de barrido de Agrobacterium tumefaciens C58 unida a Arabidopsis thaliana Columbia-0. La imagen se observó utilizando un microscopio de exploración de mesa Hitachi TM -1000 con un aumento de 10,000x. Autor: Petrovicheva et al., 2017
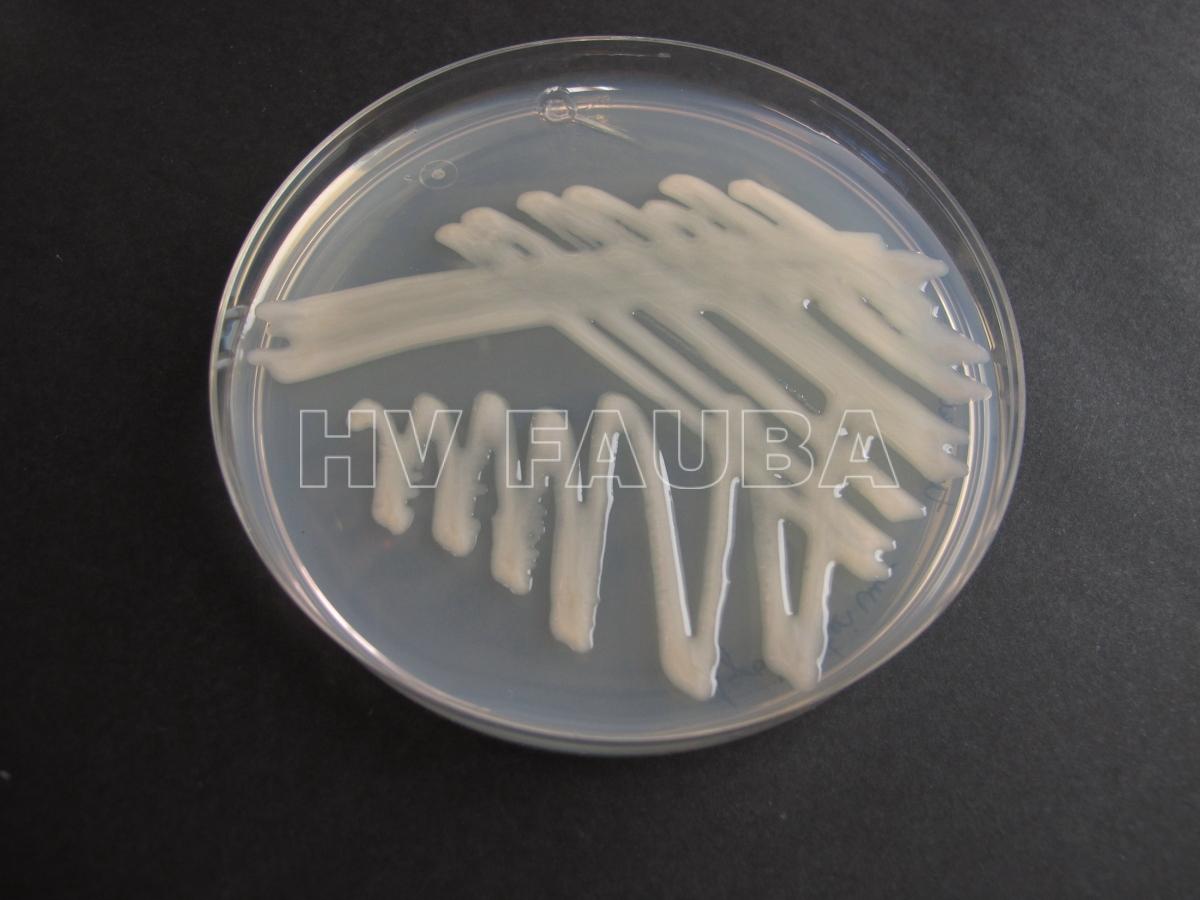
- Colonias de Agrobacterium tumefaciens creciendo en medio YMA. Autor: Universidad de Salamanca
.
.
Antecedentes
Malpighi (1679) describió las agallas en las plantas, quien creía que este crecimiento extraordinario se producía espontáneamente. Agrobacterium fue aislado por primera vez de tumores en 1897 por Fridiano Cavara en Nápoles, Italia. Después de que se reconoció que esta bacteria era la causa de la enfermedad de la agalla de la corona, surgieron dudas sobre el mecanismo por el cual causaba tumores en una variedad de plantas. Numerosos estudios muy detallados llevaron a la identificación de Agrobacterium tumefaciens como la bacteria causal que transfirió hábilmente un principio genético a las células de las plantas hospedantes y lo integró en sus cromosomas. Estos estudios han dado lugar a una variedad de mecanismos sofisticados utilizados por este organismo para ayudar en su supervivencia frente a microorganismos competidores. El conocimiento adquirido a partir de estos descubrimientos fundamentales ha abierto muchas vías para que los investigadores examinen sus principales organismos de estudio en busca de mecanismos similares de patogénesis tanto en plantas como en animales. Estos descubrimientos también avanzaron en la ingeniería genética de plantas domesticadas.
En 1977 se descubrió que las especies de Agrobacterium pueden transferir un segmento discreto de ADN oncogénico (ADN-T) al genoma de las células de la planta hospedante (Chilton et al., 1977). Este hallazgo ha estimulado un intenso interés en la biología molecular subyacente a estas asociaciones planta-microorganismo. El estudio de Agrobacterium durante los últimos 100 años ha revolucionado la genética molecular de plantas y ha dado origen a una industria completamente nueva dedicada a la modificación genética de plantas. Inicialmente, los estudios estaban destinados únicamente a identificar la causa de las agallas destructivas en plantas ornamentales y árboles frutales. En los Estados Unidos, dos fitopatólogos, Smith y Townsend (1907) reportaron que el agente causante de la enfermedad, la agalla de la corona, era una bacteria a la que llamaron Bacterium tumefaciens. Más de 30 años después, Armin Braun, un científico del Instituto Rockefeller en Princeton, Nueva Jersey, demostró que se trataba de una enfermedad vegetal muy inusual con propiedades nunca antes vistas. Sus observaciones plantearon varias preguntas intrigantes: ¿Cómo puede una bacteria causar una enfermedad que cambia las propiedades nutricionales de las células infectadas (Braun, 1958)? Y lo más sorprendente, ¿cómo podrían ocurrir estos cambios en ausencia de la bacteria (White y Braun, 1941)? Para responder a estas preguntas, se requerían tecnologías que no estaban disponibles para Braun. En la década de 1960, varios laboratorios expertos en las técnicas de genética bacteriana y química de ácidos nucleicos comenzaron a estudiar el patosistema. En un tiempo relativamente corto, se hicieron varios descubrimientos clave. Se descubrió un plásmido inusualmente grande y se demostró su asociación con la formación de agallas (Zaenen et al., 1974). A esto siguió el descubrimiento de que una parte del plásmido se transfirió y se integró aleatoriamente en el cromosoma de la célula vegetal (Chilton et al., 1977; Lemmers et al., 1980; Thomashow et al., 1980; Zambryski et al. , 1980). Durante los próximos 10 años, los estudios de laboratorios de todo el mundo respondieron a estas importantes preguntas. ¿Qué codifican los genes transferidos a la célula vegetal? ¿Qué señales se intercambian entre plantas y bacterias? ¿Y por qué Agrobacterium ha desarrollado la compleja maquinaria necesaria para formar tumores en las plantas? Las respuestas a muchas de estas preguntas han dado lugar a varios paradigmas de importancia biológica general que se relacionan no solo con las interacciones entre bacterias y plantas, sino también con las interacciones entre bacterias y animales. La comprensión de la biología básica de este sistema único hizo posible el desarrollo de Agrobacterium como actor clave en la modificación genética de las plantas. Sin embargo, esta bacteria tiene capacidades que se extienden más allá de la transformación de células vegetales. En el laboratorio, Agrobacterium puede transferir su ADN-T a algas representativas (Kumar et al., 2004), hongos (Bundock et al., 1995) e incluso células humanas (Kunik et al., 2001).
.
.
Síntomas
La Agalla de corona es una enfermedad de amplia distribución mundial, causada por la bacteria Agrobacterium tumefaciens de la familia Rhyzobiaceae. El síntoma característico de la enfermedad son los tumores que causa en plantas leñosas. Estas agallas o tumores son leñosas y mantienen la coloración y la textura del resto de la corteza.
.
.
- Autor: Ed Sikora
.
.
Las plántulas de vivero y árboles muy jóvenes atacados pueden presentar retrasos en el crecimiento y desarrollo normales, susceptibilidad a otras enfermedades y, en pocos casos, muerte por anillamiento. Árboles de más de dos años de edad presentan las agallas generalmente en la base del tronco, pero en algunos casos se han observado a lo largo de éste en los puntos de poda o en las ramas. Generalmente los árboles adultos logran desarrollarse con la presencia de la enfermedad sin efectos aparentes; sin embargo, otros patógenos de suelo pueden penetrar por las agallas decadentes y producir otras enfermedades.
Las ferritinas son una gran familia de proteínas de almacenamiento de hierro, que son utilizadas por bacterias y otros organismos para evitar la toxicidad del hierro y como una fuente segura de hierro en el citosol. Agrobacterium tumefaciens tiene dos genes que codifican ferritina: atu2771 y atu2477. La ferritina juega un papel importante en la patogénesis de A. tumefaciens mediante la regulación de la homeostasis del hierro y la supervivencia al estrés oxidativo (Yang et al., 2020).
.
- Autor: Christoph Müller
- 01 Agallas en corona o Corona de agallas en manzano. Autor: Carolina Aros Alarcón
- 02 Agalla de corona en Mango. Autor: B. Kuwana
- Agalla de corona en castaño (Aesculus hippocastanum). Autor: Ashley Watson.
- Agalla de corona en monte forestal.
- 03 Agalla de Corona. Autor: Craig Mabbett
- 02 Agalla en Forsythia (Ornamental). Autor: Jacinta Lluch Valero
- Síntomas en plantas de almendro. Autor: Rosa Cabañas
- Síntomas en plantas de almendro. Autor: Rosa Cabañas
- Síntomas en plantas de almendro. Autor: Rosa Cabañas
- Síntomas en plantas de almendro. Autor: Rosa Cabañas
.
Ciclo de la enfermedad
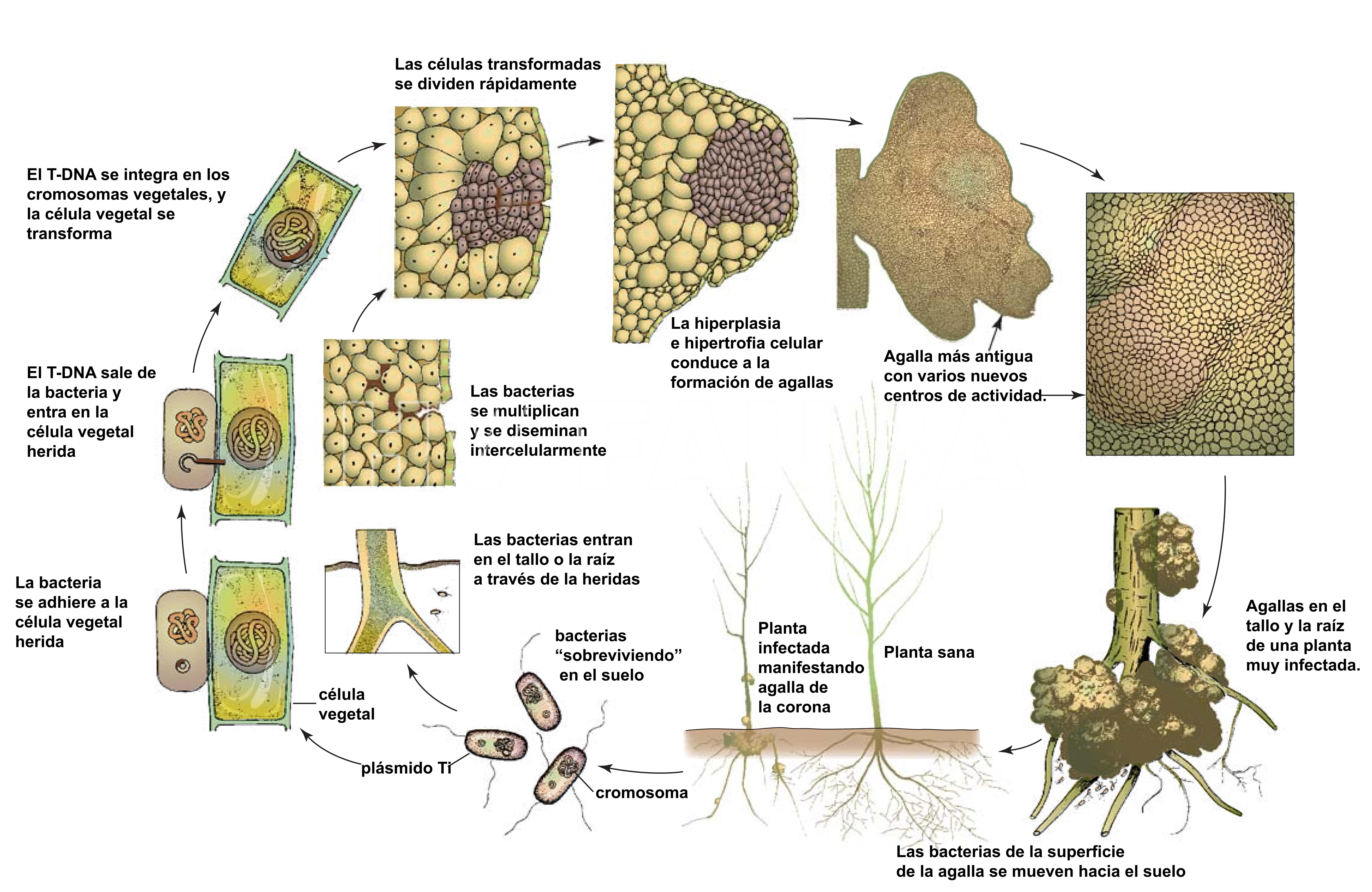
El patógeno penetra a la planta a través de heridas frescas producidas durante las labores de trasplante o mantenimiento, o por insectos y nemátodos del sistema radical. Una vez dentro del hospedante, la bacteria lo estimula a producir una gran cantidad de células (hiperplasia). Estas células continúan dividiéndose mucho más rápido de lo normal (hiperplasia) y aumentan considerablemente su tamaño (hipertrofia). Por tanto, el área afectada se transforma en un tumor o agalla. Estas alteraciones producidas en las células pueden continuar produciéndose, aunque la bacteria ya no se encuentre presente. Cuando los tejidos superficiales de las agallas se deterioran y desecan, generalmente caen al suelo junto con la bacteria, completándose de este modo su ciclo.
La bacteria se dispersa en el suelo por la remoción de éste, el agua de riego, la escorrentía y por la maquinaria utilizada para las labores de manejo (tractores, chapeadoras, etc.). Se ha observado su dispersión en el mismo árbol o entre árboles de la plantación por medio de instrumentos contaminados como cuchillos y podadoras. Las heridas innecesarias son los principales sitios de entrada de la bacteria en los árboles.
.
- Ciclo de la enfermedad de la agalla de la corona causada por Agrobacterium tumefaciens. Autor: Agrios, 2005.
.
.
Manejo de la enfermedad
El manejo de la enfermedad debe ser preventivo.
* El control de la enfermedad es de carácter obligatorio en viveros desde 1922.
* La introducción de cepas patógenas de A. tumefaciens se puede evitar mediante una inspección minuciosa de las existencias de los viveros para detectar síntomas de agallas en la corona. Antes del trasplante se deben eliminar las plantas con síntomas.
* El material de vivero debería estar idealmente certificado como libre de agallas y debe tener brotes en lugar de injertarse. Si existe la amenaza de agallas en la corona, deben evitarse todas las prácticas que eviten heridas en el tejido y los insectos masticadores.
* Las variedades muy susceptibles no se deben plantar en suelos que se sabe están infestados con el patógeno. Estos suelos deben sembrarse o plantarse con un cultivo de monocotiledónea, como por ejemplo maíz o trigo, durante varios años.
* Eliminar plantas afectadas y acidificar el hoyo (yeso) en cultivos leñosos (frutales y forestales).
* Evitar el movimiento de suelo.
* Controlar plagas de suelo.
* Utilizar sustratos y agua libre de la bacteria
* Plantar plantas sanas.
* Evitar heridas (cubrir con cicatrizantes)
* Desinfectar las herramientas entre plantas con algún desinfectante
* Tratamiento preventivo de semillas o material de propagación agámica (esqueje, estaca) con el organismo de control biológico no patógeno Agrobacterium radiobacter. En los países en los cuales estos agentes de control biológico están debidamente registrados por los organismos sanitarios competentes, esta es una herramienta de manejo relativamente económico y eficaz para administrar el desarrollo de la agalla de la corona. La cepa K84 de Agrobacterium radiobacter ha sido un agente de control biológico exitoso de la enfermedad de la agalla de la corona durante muchos años en todo el mundo. A pesar de su eficacia demostrada, el riesgo de fallo más importante cuando se usa la cepa K84 es la posibilidad de transferencia del plásmido pAgK84 a cepas patógenas de Agrobacterium. El plásmido pAgK84 codifica la producción y la inmunidad al antibiótico agrocina 84, el principal factor involucrado en el control biológico de la agalla de la corona por la cepa K84. Posteriormente, se obtuvo una segunda generación de la cepa K84, la cepa modificada genéticamente denominada K1026, que contiene una deleción en la región de transferencia de pAgK84.
El descubrimiento de la cepa K84 fue producto de una gran habilidad de observación en el campo, cuando New y Kerr (1972) observaron que la proporción de agrobacterias patógenas y no patógenas estaba estrechamente correlacionada con la incidencia de agallas de la corona en plántulas de almendros. Luego intentaron determinar si al aumentar la cantidad de cepas no patógenas en las raíces, estas bacterias inhibirían la inducción de tumores por parte de cepas patógenas. La cepa K84 se aisló en Australia a partir de suelo obtenido alrededor de una agalla de duraznero, y cuando se inoculó no causó agallas. Se seleccionó entre otros aislamientos no patógenos porque cuando se coinoculó con un patógeno en una proporción de 1:1 en las raíces de las plantas, evitó por completo la formación de agallas en la corona (Kerr, 1972; New y Kerr, 1972). La cepa K84 fue clasificada como A. radiobacter, siguiendo la taxonomía utilizada en ese momento, basada en su falta de patogenicidad, y pertenecía a la biovariedad 2 de Agrobacterium (Kerr y Panagopoulos, 1977).
La cepa K84 porta tres plásmidos autóctonos: pAgK434 (también llamado pAtK84a) de > 300 kb, que codifica la producción de agrocina 434 (Donner et al., 1993); pNoc (también llamado pAtK84b) de 173 kb, que codifica el catabolismo de la nopalina (Sciaky et al., 1978; Ellis et al., 1982) y pAgK84 de 47,7 kb, que codifica la producción e inmunidad a la agrocina 84 (Ellis et al., 1979; Ryder et al., 1987). Como pNoc tiene grandes áreas de homología (más del 50%) con el plásmido Ti de la cepa C58 de A. tumefaciens, se sugirió que pNoc podría ser un producto de deleción de un plásmido de tipo pTiC58 que se ha desarmado en el ADN-T y regiones vir que conducen a una pérdida de oncogenicidad (Clare et al., 1990).
El rasgo principal exhibido por esta cepa de control biológico es la producción del antibiótico anti-agrobacteriano altamente específico Agrocina 84 (Kerr y Htay, 1974). Este antibiótico es un análogo de nucleósido de adenina fraudulento, disustituido, que es activo contra ciertas cepas patógenas de especies de Agrobacterium (Kerr y Htay, 1974; Murphy y Roberts, 1979; Tate et al., 1979; Hayman y Farrand, 1988). La cepa K84 controla con éxito las cepas que son susceptibles a la agrocina 84 y se requiere la producción de este antibiótico para un control efectivo (Kerr y Htay, 1974; Cooksey y Moore, 1982; Lopez et al., 1989). Sin embargo, en condiciones de campo, la cepa K84 también puede controlar patógenos resistentes a la agrocina 84 (Cooksey y Moore, 1980; Lopez et al., 1987; 1989; Bouzar et al., 1991; Vicedo et al., 1993; Penyalver y Lopez, 1999). Los mecanismos utilizados por la cepa K84 para el control de patógenos resistentes a agrocina 84 aún no están completamente determinados, pero los datos disponibles sugieren que el biocontrol logrado por este agente es un fenómeno complejo, siendo la producción de agrocina 84 solo uno de los rasgos involucrados en el proceso (Farrand y Wang, 1992; Vicedo et al., 1993). La cepa K84 produce una segunda sustancia anti-agrobacteriana (agrocina 434), que es menos inhibidora que la agrocina 84 in vitro (Donner et al., 1993). Esta agrocina, que muy probablemente es un nucleósido de citidina disustituido (Fajardo et al., 1995), inhibe solo las cepas biovar 2 de Agrobacterium (ahora llamadas A. rhizogenes). Entonces, la agrocina 434 puede desempeñar un papel en el control biológico de patógenos de biovar 2 susceptibles a la agrocina 434 (McClure et al., 1998). K84 también produce una tercera sustancia similar a un antibiótico denominada ALS84, que in vitro inhibe muchas cepas tumorigénicas de Agrobacterium (Penalver et al., 1994). La actividad inhibidora de ALS84 se correlaciona con la producción de sideróforos por K84 en condiciones limitantes de hierro (Penyalver et al., 2000).
A pesar de su eficacia demostrada, el riesgo de fallo más importante cuando se usa la cepa K84 es la posibilidad de transferencia del plásmido pAgK84 a cepas patógenas de Agrobacterium. Para evitar esta transferencia y salvaguardar el control biológico de la agalla de la corona contra la ruptura debido a la transferencia de pAgK84 desde K84 a cepas patógenas de Agrobacterium, se desarrolló la cepa GEM K1026, que alberga un derivado de transferencia deficiente de pAgK84 (Tra-) (Jones et al., 1988). La cepa K1026 se obtuvo después de una cooperación exitosa entre los equipos australianos involucrados en el descubrimiento y caracterización de la cepa K84 y un equipo norteamericano que trabaja en la biología de los plásmidos de Agrobacterium spp. (Jones et al., 1988). La eficacia comparativa in vivo de las cepas K84 y K1026 contra las cepas de Agrobacterium tanto susceptibles como resistentes a la agrocina 84 fue determinada experimentalmente en varios países, incluyendo Australia, España, EE. UU., Jordania y Tunisia, demostrando claramente que la cepa K1026 es tan eficaz como la K84 en el control de la agalla de la corona en diferentes condiciones experimentales, hospedantes y países. Ambas cepas controlaron la agalla de la corona incluso bajo alta presión de inóculo. Ambos también pudieron controlar la enfermedad causada por patógenos tanto susceptibles como resistentes a la agrocina 84, lo que demuestra que la cepa K1026 también fue capaz de usar otros mecanismos no relacionados con la susceptibilidad a la agrocina 84, de la misma manera que la cepa K84. Debido a las restricciones al uso de GEM a campo en muchos países, los únicos resultados disponibles sobre la eficacia comparativa del biocontrol en condiciones de campo abierto son los de Túnez y EE. UU., donde ambas cepas mostraron resultados similares (Peñalver et al., 2000).
La aplicación de estas cepas antagonistas empapando semillas o sumergiendo los trasplantes o esquejes puede prevenir la infección de la mayoría de las cepas de A. tumefaciens debido a la producción del antibiótico agrocin 84 por la cepa K84 de A. radiobacter. Algunas propiedades curativas son exhibidas por una mezcla disponible comercialmente de 2,4-xilenol y metacresol en una emulsión de aceite-agua cuando se pinta directamente sobre tumores establecidos. Pero esto rara vez se utiliza debido al elevado costo de la mano de obra y las limitaciones de tiempo. En los países en los cuales estos agentes de control biológico están debidamente registrados por los organismos sanitarios competentes, el control biológico con la bacteria antagónica A. radiobacter se usa generalmente en los viveros forestales, sumergiendo las raíces en una suspensión con la cepa de control biológico A. radiobacter K84 o la K1026.
.
.
Bibliografía
Abrahamovich E, López AC, Alippi AM (2014) Diversidad de cepas de Agrobacterium rubi aisladas de arándanos. Revista argentina de microbiología 46(3): 237-241. doi: 10.1016/S0325-7541(14)70078-1
Ade-Ogunnowo FE, Adejoye OD, Awofodu AD, Abdulwahab MM (2017) Comparative study of the effects of artesunate and garlic extract on tomato crown gall disease. Journal of Agricultural Research and Development 16(1). doi: 10.4314/jard.v16i1.9
Alburquerque N, Faize L, Burgos L (2017) Silencing of Agrobacterium tumefaciens oncogenes iptand iaaM induces resistance to crown gall disease in plum but not in apricot. Pest Management Science 73: 2163-2173. doi: 10.1002/ps.4600
Ali H, Khalil A, Hussain A, Imran (2010) Incidence and Severity of Crown Gall Disease of Cherry, Apple and Apricot Plants Caused by Agrobacterium tumefaciens in Nagar Valley of Gilgit-Baltistan, Pakistan. Pakistan Journal of Nutrition 9(6). doi: 10.3923/pjn.2010.577.581
Alippi AM, Lopez AC, Balatti PA (2010) First Report of Agrobacterium rubi and A. rhizogenes Causing Crown and Root Gall and Hairy Root on Blueberry in Argentina. Plant Disease 94(8): 1064. doi: 10.1094/PDIS-94-8-1064C
Alippi AM, López AC, Balatti PA (2011) Métodos para la detección de Agrobacterium a partir de muestras de material vegetal, suelo y agua. Revista Argentina de Microbiología 43: 278-286. Link
Alippi AM, López AC, Balatti PA (2012) Diversity among agrobacteria isolated from diseased plants of blueberry (Vaccinium corymbosum) in Argentina. Eur J Plant Pathol 134: 415–430. doi: 10.1007/s10658-012-0001-x
Anand A, Uppalapati SR, Ryu CM, et al. (2008) Salicylic Acid and Systemic Acquired Resistance Play a Role in Attenuating Crown Gall Disease Caused by Agrobacterium tumefaciens. Plant Physiology 146(2): 703-715. doi: 10.1104/pp.107.111302
Aysan Y, Sahin F (2003) An outbreak of crown gall disease on rose caused by Agrobacterium tumefaciens in Turkey. Plant Pathology 52: 780-780. doi: 10.1111/j.1365-3059.2003.00889.x
Benjama A, Boubaker A, Khlief H, et al. (2002) Susceptibility of stone-fruit rootstocks, rose and grapevine to Agrobacterium radiobacter var. tumefaciens in Arab Mediterranean countries. EPPO Bulletin, 32: 463-466. doi: 10.1046/j.1365-2338.2002.00589.x
Bouzar H, Daouzli N, Krimi Z, et al. (1991) Crown gall incidence in plant nurseries of Algeria, characteristics of Agrobacterium tumefaciens strains, and biological control of strains sensitive and resistant to agrocin 84. Agronomie 11: 901–908. doi: 10.1051/agro:19911007
Bouzar H, Ouadah D, Krimi Z, et al. (1993) Correlative Association between Resident Plasmids and the Host Chromosome in a Diverse Agrobacterium Soil Population. Appl Environ Microbiol. 59(5): 1310-1317. doi: 10.1128/aem.59.5.1310-1317.1993
Bouzar H, Jones JB (2001) Agrobacterium larrymoorei sp. nov., a pathogen isolated from aerial tumors of Ficus benjama. International Journal of Systematics and Evolutionary Microbiology 51: 1023–1026. doi:
Braun AC (1958) A physiological basis for autonomous growth of the crown-gall tumor cell. Proc. Natl. Acad. Sci. U.S.A. 44, 344–349. doi: 10.1073/pnas.44.4.344
Bundock P, denDulk-Ras A, Beijersbergen A, Hooykaas PJJ (1995) Trans-kingdom T-DNA transfer from Agrobacterium tumefaciens to Saccharomyces cerevisiae. EMBO J. 14: 3206–3214. doi: 10.1002/j.1460-2075.1995.tb07323.x
Chandran Darbari V, Waksman G (2015) Structural Biology of Bacterial Type IV Secretion Systems. Annu Rev Biochem. 84: 603-629. doi: 10.1146/annurev-biochem-062911-102821
Chilton M-D, Drummond MH, Merlo DJ, et al. (1977) Stable incorporation of plasmid DNA into higher plant cells: the molecular basis of crown gall tumorigenesis. Cell 11: 263–271. doi: 10.1016/0092-8674(77)90043-5
Clare BG, Kerr A, Jones DA (1990) Characteristics of the nopaline catabolic plasmid in Agrobacterium strains K84 and K1026 used for biological control of crown gall disease. Plasmid 23: 126–137. doi: 10.1016/0147-619X(90)90031-7
Cooksey DA and Moore LW (1980) Biological control of crown gall with fungal and bacterial antagonists. Phytopathology 70: 506–509. doi: 10.1094/Phyto-70-506
Cooksey DA, Moore LW (1982) Biological control of crown gall with an agrocin mutant of Agrobacterium radiobacter. Phytopathology 72: 919–921. doi: 10.1094/Phyto-72-919
Correa OS, Montecchia MS, Romero AM, Wright ER (2005) Detección de Agrobacterium patógeno en material de propagación de arándanos en Argentina. I Congreso Latinoamericano de arándanos y otros berries. Libro de Resúmenes. Ciudad de Buenos Aires. 16 de septiembre. Página: 33.
Costechareyre D, Rhouma A, Lavire C, et al. (2010) Rapid and efficient identification of Agrobacterium species by recA allele analysis. Microb Ecol. 60(4): 862-72. doi: 10.1007/s00248-010-9685-7
Czolkoss S, Safronov X, Rexroth S, et al. (2021) Agrobacterium tumefaciens Type IV and Type VI Secretion Systems Reside in Detergent-Resistant Membranes. Front. Microbiol. 12: 754486. doi: 10.3389/fmicb.2021.754486
Donner SC, Jones DA, McClure NC, et al. (1993) Agrocin 434, a new plasmid encoded agrocin from the biocontrol Agrobacterium strains K84 and K1026, which inhibits biovar 2 agrobacteria.
Physiological and Molecular Plant Pathology 42: 185–194. doi: 10.1006/pmpp.1993.1017
Ellis JG, Kerr A (1979) Transfer of agrocin 84 production from strain 84 to pathogenic recipients: a comment on previous paper. In: Schippers B and Gams W (eds) Soil-borne pathogens (pp 579–583) Academic Press, New York.
Ellis JG, Kerr A, Van Montagu M, Schell J (1979) Agrobacterium genetic studies on agrocin 84 production and the biological control of crown gall. Physiological Plant Pathology 15: 311–319. doi: 10.1016/0048-4059(79)90082-1
Ellis JG, Kerr A, Petit A, Tempe J (1982) Conjugal transfer of nopaline and agropine Ti-plasmids – the role of agrocinopines. Molecular and General Genetics 186: 269–274. doi: 10.1007/BF00331861
Fajardo NN, Tate ME, Clare BG (1995) Agrocin 434: an additional biological control component for crown gall. In: Ryder MH, Stephens PM and Bowen GD (eds) Improving Plant Productivity with Rhizosphere Bacteria (pp 128–130) CSIRO Division of Soils, Adelaide, Australia
Farrand SK, Wang C, Hong S, et al. (1992) Deletion derivatives of pAgK84 and their use in the analysis of Agrobacterium plasmid functions. Plasmid 28: 201–212. doi: 10.1016/0147-619X(92)90052-C
Farrand SK, van Berkum P, Oger P (2003) Agrobacterium is a definable genus of the family Rhizobiaceae. International Journal of Systematics and Evolutionary Microbiology 53: 1681–1687.
Frikha-Gargouri O, Ben Abdallah D, Bhar I, Tounsi S (2017) Antibiosis and bmyB Gene Presence As Prevalent Traits for the Selection of Efficient Bacillus Biocontrol Agents against Crown Gall Disease. Frontiers in Plant Science 8: 1363. doi: 10.3389/fpls.2017.01363
Gelvin SB (2000) Agrobacterium and plant genes involved in T-DNA transfer and integration. Annu Rev Plant Physiol Plant Mol Biol. 51: 223-256. doi: 10.1146/annurev.arplant.51.1.223
González‐Mula A, Lang J, Grandclément C, et al. (2018) Lifestyle of the biotroph Agrobacterium tumefaciens in the ecological niche constructed on its host plant. New Phytologist 219: 350-362. doi: 10.1111/nph.15164
Gonzalez-Mula A, Lachat J, Mathias L, et al. (2019) The biotroph Agrobacterium tumefaciens thrives in tumors by exploiting a wide spectrum of plant host metabolites. New Phytol 222: 455-467. doi: 10.1111/nph.15598
, , (2021) Seeing the forest for the trees: Use of phages to treat bacterial tree diseases. Plant Pathology 70: 1987– 2004. doi: 10.1111/ppa.13465
Hayman GT, Farrand SK (1988) Characterization and mapping of the agrocinopine-agrocin 84 locus on the nopaline Ti plasmid pTiC58. Journal of Bacteriology 170: 1759. doi: 10.1128/jb.170.4.1759-1767.1988
Hooykaas PJJ (2023) The Ti Plasmid, Driver of Agrobacterium Pathogenesis. Phytopathology 113(4): 594-604. doi: 10.1094/PHYTO-11-22-0432-IA
Jones DA, Ryder MH, Clare BG, et al. (1988) Construction of a Tra- deletion mutant of pAgK84 to safeguard the biological control of crown gall. Molecular General Genetics 212: 207–214. doi: 10.1007/BF00334686
Jones DA, Kerr A (1989) Agrobacterium radiobacter Strain K1026, a Genetically Engineered Derivative of Strain K84, for Biological Control of Crown Gall. Plant Disease 73: 15-18. doi: 10.1094/PD-73-0015
Jones DA, Ryder MH, Clare BG, et al. (1991) Biological control of crown gall using Agrobacterium strains K84 and K1026. In: Komada H, Kiritani K and Bay-Petersen J (eds) The biological control of plant diseases (pp 161–170) FFTC Book Series no. 42. Food and Fertilizer Technology Center for the Asian and Pacific Region, Taipei, Taiwan
Kado CI (2014) Historical account on gaining insights on the mechanism of crown gall tumorigenesis induced by Agrobacterium tumefaciens. Front Microbiol. 5: 340. doi: 10.3389/fmicb.2014.00340
Kahla Y, Zouari-Bouassida K, Rezgui F, et al. (2017) Efficacy of Eucalyptus cinerea as a Source of Bioactive Compounds for Curative Biocontrol of Crown Gall Caused by Agrobacterium tumefaciens Strain B6,” BioMed Research International, vol. 2017, Article ID 9308063, 10 pages. doi: 10.1155/2017/9308063
Keane PJ, Kerr A, New PB (1970) Crown gall of stone fruit. 11. Identification and nomenclature of Agrobacterium isolates. Australian Journal of Biological Sciences 23: 585-595.
Kechris KJ, Lin JC, Bickel PJ, Glazer AN (2006) Quantitative exploration of the occurrence of lateral gene transfer by using nitrogen fixation genes as a case study. Proceedings of the National Academy of Sciences 103: 9584–9589. doi: 10.1073/pnas.0603534103
Kerr A (1972) Biological control of crown gall: seed inoculation. Journal of Applied Bacteriology 35: 493–497. doi: 10.1111/j.1365-2672.1972.tb03727.x
Kerr A, Htay K (1974) Biological control of crown gall through bacteriocin production. Physiological Plant Pathology 4: 37–44. doi: 10.1016/0048-4059(74)90042-3
Kerr A, Panagopoulos CG (1977) Biotypes of Agrobacterium radiobacter var. tumefaciens and their biological control. Phytopathologische Zeitschrift 90: 172–179.
Knauf VC, Panagopoulos CG, Nester EW (1983) Comparison of Ti plasmids from three different biotypes of Agrobacterium tumefaciens isolated from grapevines. J Bacteriol. Mar 153(3): 1535-1542. doi: 10.1128/jb.153.3.1535-1542.1983
Kraepiel Y, Barny M‐A (2016) Gram‐negative phytopathogenic bacteria, all hemibiotrophs after all?. Molecular Plant Pathology 17: 313-316. doi: 10.1111/mpp.12345
, , , et al. (2022) Cryo-EM structure of the Agrobacterium tumefaciens type IV secretion system-associated T-pilus reveals stoichiometric protein-phospholipid assembly. bioRxiv 2022.09.25.509369; doi: 10.1101/2022.09.25.509369
Kumar SV, Misquitta RW, Reddy VS, et al. (2004) Genetic transformation of the green alga Chlamydomonas reinhardtii by Agrobacterium tumefaciens. Plant Sci. 166: 731–738. doi: 10.1016/j.plantsci.2003.11.012
Kunik T, Tzfira T, Kapulnik Y, et al. (2001) Genetic transformation of HeLa cells by Agrobacterium. Proc. Natl. Acad. Sci, U.S.A. 98: 1871–1876. doi: 10.1073/pnas.98.4.1871
Lasalle F, Campillo T, Vial L, et al. (2011) Genomic species are ecological species as revealed by comparative genomics in Agrobacterium tumefaciens. Genome Biology and Evolution 3: 762–781. doi: 10.1093/gbe/evr070
Lemmers M, Holsters M, Zambryski P, et al. (1980) Internal organization, boundaries and integration of Ti-plasmid DNA in nopaline crown gall tumors. J. Mol. Biol. 144: 353–376. doi: 10.1016/0022-2836(80)90095-9
Lim SH, Kim JG, Kang HW (2009) Novel SCAR primers for specific and sensitive detection of Agrobacterium vitis strains. Microbiol Res. 164(4): 451-60. doi: 10.1016/j.micres.2007.02.006
Lopez MM, Gorris MT, Temprano FJ, Orive RJ (1987) Results of seven years of biological control of Agrobacterium tumefaciens in Spain. Bulletin EPPO/OEPP Bulletin 17: 273–280. doi: 10.1111/j.1365-2338.1987.tb00039.x
Lopez MM, Gorris MT, Salcedo CI, et al. (1989) Evidence of biological control of Agrobacterium tumefaciens strains sensitive and resistant to agrocin 84 by different Agrobacterium radiobacter strains on stone fruit trees. Applied and Environmental Microbiology 55: 741–746. doi: 10.1128/aem.55.3.741-746.1989
Ma LS, Hachani A, Lin JS, et al. (2014) Agrobacterium tumefaciens Deploys a Superfamily of Type VI Secretion DNase Effectors as Weapons for Interbacterial Competition In Planta. Cell Host & Microbe 16(1): 94-104. doi: 10.1016/j.chom.2014.06.002
Martin DM, Rossini M, Gallo S, et al. (2014) Et-ByM-4. Primer reporte de agalla de corona (Agrobacterium tumefasciens) en avellano en Argentina. En: 3º Congreso Argentino de Fitopatología. Libro de Resúmenes. Editor/es: Leonardo Daniel Ploper. Página/s: 166. ISBN/ISSN: 978-987-24373-1-2.
McClure NC, Ahmadi AR, Clare BG (1998) Construction of a range of derivatives of the biological control strain Agrobacterium rhizogenes K84: a study of factors involved in biological control of crown gall disease. Applied and Environmental Microbiology 64: 3977–3982. doi: 10.1128/AEM.64.10.3977-3982.1998
Moore LW, Bouzard H, Burr T (2001) Agrobacterium. In Schaad NW, Jones JB, Chun W (Eds.), Laboratory guide for the identification of plant pathogenic bacteria (pp. 17–35). St. Paul: APS Press.
Murphy PJ, Roberts WP (1979) A basis for agrocin 84 sensitivity in Agrobacterium radiobacter. Journal of General Microbiology 114: 207–213. doi:
Nesme X, Michel MF, Digat B (1987) Population Heterogeneity of Agrobacterium tumefaciens in Galls of Populus L. from a Single Nursery. Appl Environ Microbiol. 53(4): 655-9. doi: 10.1128/aem.53.4.655-659.1987
New PB and Kerr A (1972) Biological control of crown gall: field measurements and glasshouse experiments. Journal of Applied Bacteriology 35: 279–287. doi: 10.1111/j.1365-2672.1972.tb03699.x
Omarini A, Alet AI, Massone E, et al. (2006) Efectividad de la cepa de Agrobacterium radiobacter K84 en el control biológico de la agalla de corona. Congreso; XXVI Reunión de la Asociación Argentina de Fisiología Vegetal. Link
Panday D, Schumann P, Das SK (2011) Rhizobium pusense sp. nov., isolated from the rhizosphere of chickpea (Cicer arietinum L.). Int J Syst Evol Microbiol. 61(Pt 11): 2632-2639. doi:
Peñalver R, Vicedo B, Salcedo CI, Lopez MM (1994) Agrobacterium radiobacter strain K84, K1026 and K84 Agr− produce an antibiotic-like substance, active in vitro against A. tumefaciens and phytopathogenic Erwinia and Pseudomonas. Biocontrol Science and Technology 4: 259–267. doi: 10.1080/09583159409355334
Peñalver R, Lopez MM (1999) Cocolonization of the rhizosphere by pathogenic Agrobacterium strains and nonpathogenic strains K84 and K1026, used for crown gall biocontrol. Applied and Environmental Microbiology 65: 1936–1940. doi: 10.1128/AEM.65.5.1936-1940.1999
Peñalver R, Vicedo B, López MM (2000) Use of the Genetically Engineered Agrobacterium Strain K1026 for Biological Control of Crown Gall. European Journal of Plant Pathology 106: 801–810. doi: 10.1023/A:1008785813757
Peñalver R, Oger P, López MM, Farrand SK (2001) Iron-binding compounds from Agrobacterium spp.: biological control strain Agrobacterium rhizogenes K84 produces a hydroxamate siderophore. Appl Environ Microbiol. 67(2): 654-64. doi: 10.1128/AEM.67.2.654-664.2001
Peñalver R, Marco-Noales E, López MM (2018) Tumores causados por Agrobacterium (Rhizobium) spp. Pág. 221- 237. En: Enfermedades de plantas causadas por bacterias. Ed. López MM, Murillo J, Montesinos E, Palacio- Bielsa A.
Pérez B, Fernández R, Wright E, et al. (2005) Agalla de Corona y Royas sobre “Berries” en algunas localidades de la Argentina En: XIII Congreso Latinoamericano de Fitopatología. 19-22 de abril, 2005. Villa Carlos Paz, Córdoba. Libro de resúmenes: 540. Página/s: 1.
Pérez BA, Wright ER, Fernández RR, Asciutto K (2006) Blueberry crown gall in Argentina. APS Annual Meeting. Proceedings. Austin. Texas. USA. 30 de julio – 3 de agosto.
Petrovicheva A, Joyner J, Muth TR (2017) Quantification of Agrobacterium tumefaciens C58 attachment to Arabidopsis thaliana roots. FEMS Microbiology Letters 364(18): fnx158. doi: 10.1093/femsle/fnx158
Pitzschke A (2013) Agrobacterium infection and plant defense—transformation success hangs by a thread. Front. Plant Sci. 4: 519. doi: 10.3389/fpls.2013.00519
Portier P, Fischer-Le Saux M, Mougel C, et al. (2006) Identification of genomic species in Agrobacterium biovar 1 by AFLP genomic markers. Appl Environ Microbiol. 72(11): 7123-31. doi: 10.1128/AEM.00018-06
Puławska J, Willems A, Sobiczewski P (2006) Rapid and specific identification of four Agrobacterium species and biovars using multiplex PCR. Syst Appl Microbiol. 29(6): 470-9. doi: 10.1016/j.syapm.2005.11.002
Pulawska J, Willems A, Sobiczewski P (2012) Rhizobium skierniewicense sp. nov. isolated from tumors on chrysanthemum and Prunus in Poland. International Journal of Systematic and Evolutionary Microbiology 62: 895–899.
Quispe-Huamanquispe DG, Gheysen G, Kreuze JF (2017) Horizontal Gene Transfer Contributes to Plant Evolution: The Case of Agrobacterium T-DNAs. Frontiers in Plant Science 8: 2015. doi: 10.3389/fpls.2017.02015
Rhouma A, Ferchichi A, Hafsa M, Boubaker A (2004) Efficacy of the Non-Pathogenic Agrobacterium Strains K84 and K1026 against Crown Gall in Tunisia. Phytopathol. Mediterr. 43: 167-176. doi: https://doi.org/10.14601/Phytopathol_Mediterr-1745
Ryder MH, Tate ME, Kerr A (1985) Virulence properties of strains of Agrobacterium on the apical and Basal surfaces of carrot root discs. Plant Physiol. 77(1): 215-21. doi: 10.1104/pp.77.1.215
Ryder MH, Slota JE, Scarim A, Farrand SK (1987) Genetic analysis of agrocin 84 production and immunity in Agrobacterium spp. Journal of Bacteriology 169: 4184–4189. doi: 10.1128/jb.169.9.4184-4189.1987
Rouhrazi K, Rahimian H (2014) Biochemical and genetic characterisation of Agrobacterium tumefaciens the causal agent of walnut crown gall disease in Iran. Archives of Phytopathology and Plant Protection 47(20): 2493-2500. doi: 10.1080/03235408.2014.880575
Seleme F, González Vera C, Di Barbaro G, et al. (2006) Agalla de corona en plantas de olivo (Olea europea l.) causada por Agrobacterium tumefaciens (Smith y Thownsend) Conn. en la provincia de la Rioja. Revista del CIZAS 7: 55-63. ISSN 1515-0453
Setti B, Bencheikh M (2013) Isolation and characterization of the Agrobacterium tumefaciens from almond nurseries in chlef region in western Algeria. European Scientific Journal 9(30). eISSN 1857- 7431.
Skerman VBD, McGowan V, Sneath PHA (1980) Approved Lists of Bacterial Names. Intl. J. Syst. Bacteriol. 30(1): 225-420. doi:
Sciaky D, Montoya AL, Chilton MD (1978) Fingerprints of Agrobacterium Ti plasmids. Plasmid 1: 238–253. doi: 10.1016/0147-619X(78)90042-2
Smith EF, Townsend CO (1907) A plant tumor of bacterial origin. Science 25: 671–673. doi: 10.1126/science.25.643.671
Süle S (1978) Biotypes of Agrobacterium tumefaciens in Hungary. Journal of Applied Bacteriology 44: 207-213. doi: 10.1111/j.1365-2672.1978.tb00792.x
Stockwell VO, Moore LW, Loper JE (1993) Fate of Agrobacterium radiobacter K84 in the environment. Appl Environ Microbiol. 59(7): 2112-2120. doi: 10.1128/aem.59.7.2112-2120.1993
Tate ME, Murphy PJ, Roberts WP, Kerr A (1979) Adenine N6-substituent of agrocin 84 determines its bacteriocin-like specificity. Nature (London) 280: 697–699. doi: 10.1038/280697a0
Terán Baptista ZP, de Los Angeles Gómez A, Kritsanida M, et al. (2020) Antibacterial activity of native plants from Northwest Argentina against phytopathogenic bacteria. Nat Prod Res. 34(12): 1782-1785. doi: 10.1080/14786419.2018.1525716
Thomashow M, Panagopoulos C, Gordon M, et al. (1980) Host range of Agrobacterium tumefaciens is determined by the Ti plasmid. Nature 283: 794–796. doi: 10.1038/283794a0
Thomashow MF, Nutter R, Montoya AL, et al. (1980) Integration and organization of Ti plasmid sequences in crown gall tumors. Cell 19: 729–739. doi: 10.1016/S0092-8674(80)80049-3
Torres Tejerizo GA (2011) Estudios genómicos y caracterización funcional de rizobios tipo Oregon noduladores de alfalfa y otras leguminosas. Tesis doctoral, Universidad de La Plata. Link
Velázquez E, Palomo JL, Rivas R, et al. (2010) Analysis of core genes supports the reclassification of strains Agrobacterium radiobacter K84 and Agrobacterium tumefaciens AKE10 into the species Rhizobium rhizogenes. Syst Appl Microbiol. 33(5): 247-51. doi: 10.1016/j.syapm.2010.04.004
Vicedo B, Penalver R, Asins MJ, Lopez MM (1993) Biological control of Agrobacterium tumefaciens, colonization, and pAgK84 transfer with Agrobacterium radiobacter K84 and the Tra-mutant strain K1026. Applied Environmental and Microbiology 59: 309–315. doi: 10.1128/aem.59.1.309-315.1993
Vicedo B (1995) Plasmid transfer between Agrobacterium tumefaciens and A. radiobacter in biological control assays: characterization and behavior of the transconjugants. PhD Thesis. University of Valencia, Valencia, Spain.
Vicedo B, Lopez MJ, Asins MJ, Lopez MM (1996) Spontaneous transfer of the Ti plasmid of Agrobacterium tumefaciens and the nopaline catabolism plasmid of A. radiobacter strain K84 in crown gall tissue. Phytopathology 86: 528–534. doi: 10.1094/Phyto-86-528
Wang Y, Zhang S, Huang F, et al. (2018) VirD5 is required for efficient Agrobacterium infection and interacts with Arabidopsis VIP2. New Phytologist 217: 726-738. doi: 10.1111/nph.14854
, , , et al. (2022) The antibacterial T6SS of Agrobacterium tumefacien promotes disease occurrence and influences crown gall microbiota composition. bioRxiv 2022.10.28.514271; doi: 10.1101/2022.10.28.514271
Watson B, Nester EW (2005) A plasmid was present after all. En: Nester E, Gordon MP, Kerr A (Eds.), Agrobacterium tumefaciens. From plant pathology to biotechnology (pp. 53-65). Minnesota, UK: APS Press.
Winans SC (1992) Two-way chemical signaling in Agrobacterium-plant interactions. Microbiol. Rev. 56, 12–31. doi: 10.1128/mr.56.1.12-31.1992
White PR, Braun AC (1941) Crown gall production by bacteria-free tumor tissues. Science 94: 239–241. doi: 10.1126/science.94.2436.239
Wu L, Xiao H, Zhao L, Cheng Q (2022) CRISPR/Cas9-mediated generation of fls2 mutant in Nicotiana benthamiana for investigating the flagellin recognition spectrum of diverse FLS2 receptors. Plant Biotechnol J, 20: 1853-1855. doi: 10.1111/pbi.13898
, , , et al. (2021) Reconstruction and analysis of a genome‐scale metabolic model for Agrobacterium tumefaciens. Mol Plant Pathol. 00: 1– 13. https://doi.org/10.1111/mpp.13032
, , , et al. (2021) Reconstruction and analysis of a genome‐scale metabolic model for Agrobacterium tumefaciens. Molecular Plant Pathology 22: 348– 360. doi: 10.1111/mpp.13032
Yang J, Pan X, Xu Y, et al. (2020) Agrobacterium tumefaciens ferritins play an important role in full virulence through regulating iron homeostasis and oxidative stress survival. Mol Plant Pathol. 21(9): 1167-1178. doi: 10.1111/mpp.12969
Young JM, Kuykendall LD, Martínez-Romero E, et al. (2001) A revision of Rhizobium Frank 1889, with an emended description of the genus, and the inclusion of all species of Agrobacterium Conn 1942 and Allorhizobium undicola de Lajudie et al. 1998 as new combinations: Rhizobium radiobacter, R. rhizogenes, R. rubi, R. undicola and R. vitis. International Journal of Systematics and Evolutionary Microbiology 51: 89–103. doi:
Young JM, Kerr A, Sawada H (2005) Genus II. Agrobacterium. In: Garrity GM, Brenner DJ, Krieg NR, Staley JR (Eds.), Bergey’s manual of systematic bacteriology, volume two: The Proteobacteria, parts A – C (pp. 340–345). Athens: Springer – Verlag.
Zaenen I, Van Larabeke N, Teuchy H, et al. (1974) Supercoiled circular DNA in crown gall inducing Agrobacterium strains. J. Mol. Biol. 86: 109–127. doi: 10.1016/S0022-2836(74)80011-2
Zambryski P, Holsters M, Kruger K, et al. (1980) Tumor DNA structure in plant cells transformed by A. tumefaciens. Science 209: 1385–1391. doi: 10.1126/science.6251546
Zhang L, Li X, Zhang F, Wang G (2014) Genomic analysis of Agrobacterium radiobacter DSM 30147(T) and emended description of A. radiobacter (Beijerinck and van Delden 1902) Conn 1942 (Approved Lists 1980) emend. Sawada et al. 1993. Standards in Genomic Sciences 9: 9030574. doi: 10.4056/sigs.4688352